Бенчмаркинг детекции геномных полиморфизмов
Для характеризации точности детекции часто применяются характеристики чувствительности и специфичности1. Применительно к детекции геномных вариантов чувствительность означает долю всех истинных вариантов относительно референсного генома, которую удалось задетектировать с помощью пайплайна.
Специфичность обычно обозначает долю результатов, идентифицированных как отрицательные среди истинно отрицательных событий. Определение всевозможных "отрицательных" событий в случае с геномными вариантами становится расплывчатым. В связи с этим можно (1) охарактеризовать долю ложноположительных вариантов среди всех вариантов, т.е. подсчитать FDR, либо, (2) если рассматриваются одни и те же геномные регионы, сравнить количество ложноположительных результатов в абсолютном выражении.
Если для тестов используются результаты секвенирования некоторой панели генов или полноэкзомного секвенирования, то важно рассматривать варианты внутри только этих регионов, т.к. остальные районы генома не будут иметь покрытия ридами.
Тем не менее, даже внутри регионов направленного секвенирования могут быть районы, не покрытые ридами, что приведет к ложноотрицательным исходам. Недопокрытие может появиться от того, что действительно не было ридов, исходящих из этих регионов по "мокрым причинам" (на небиоинформатических стадиях, т.е. до стадии получения ридов), а также и по причинам ошибок выравниваниия на геном. Долю таких исходов также важно оценить.
Команды, используемые для генерации сравнений, приведены ниже.
Тестирование герминального пайплайна#
Тестовые образцы#
Известным источником "золотого стандарта" для поиска геномных вариантов является консорциум "Genome in a Bottle". Этот консорциум создан на базе Национального Интитута Стандартов и Технологий США и, в частности, занимается характеризацией человеческих генов для создания стандартов для валидации.
В рамках этого консорциума подробно охарактеризованы образец NA12878/HG001 из проекта HapMap и два семейных трио: трио евреев Ашкенази и трио китайцев Хань.
При подготовке данного раздела была использована версия v4.2.1 датасета "Genome In a Bottle". В качестве сырых данных использовались продукты полноэкзомного секвенирования панелью Nextera Rapid Capture.
Результаты сравнения#
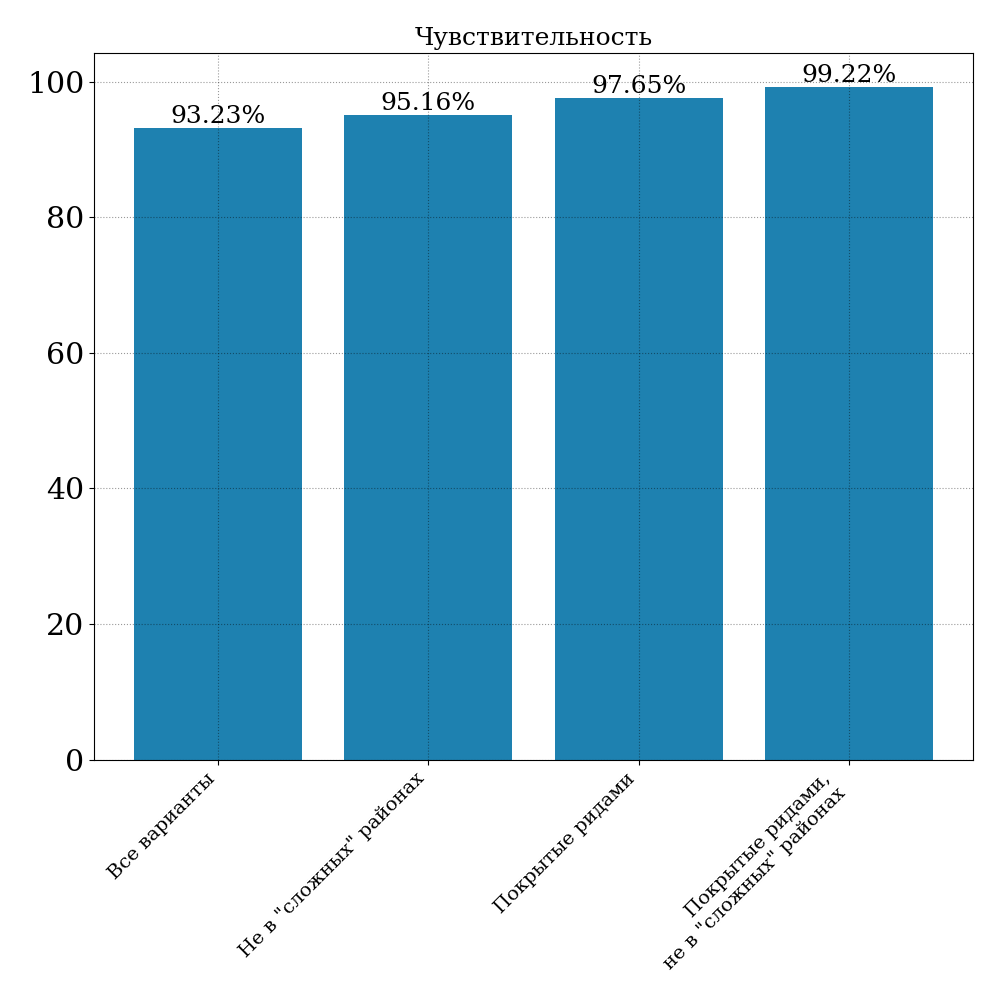
Была проверена чувствительность методики детекции вариантов. Оказывается, что если рассмотреть все варианты, попадающие в заявленную панель Nextera и в регионы уверенности (т.е. те, в которые консорциум гарантирует верные коллы), то чувствительность оказывается 93.23%.
Рассмотрение незадетектированных вариантов позволяет заметить, что одной из основных причин является отсутствие ридов, покрывающих этот вариант. Пример такого варианта представлен на рис. 1:
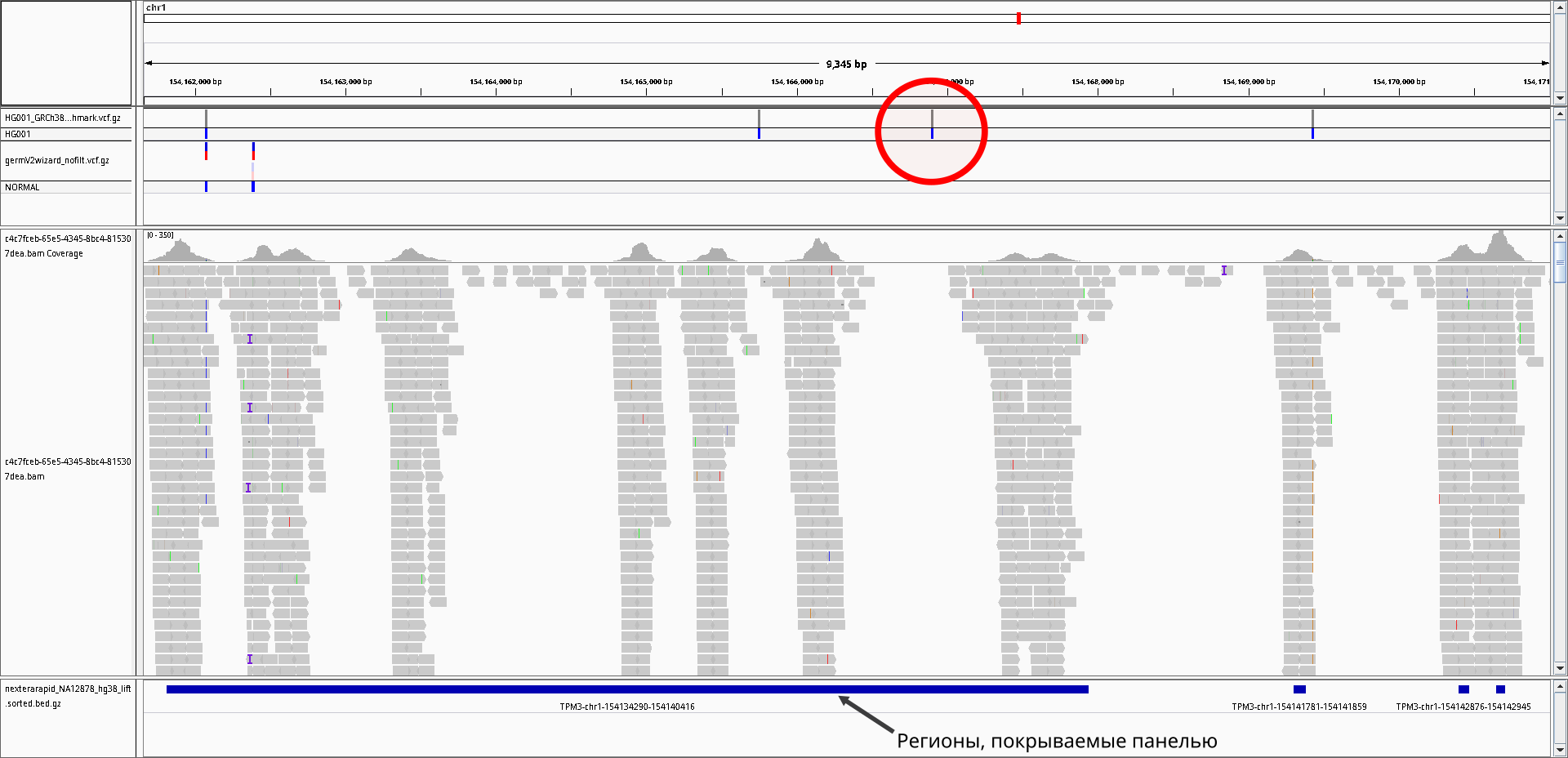
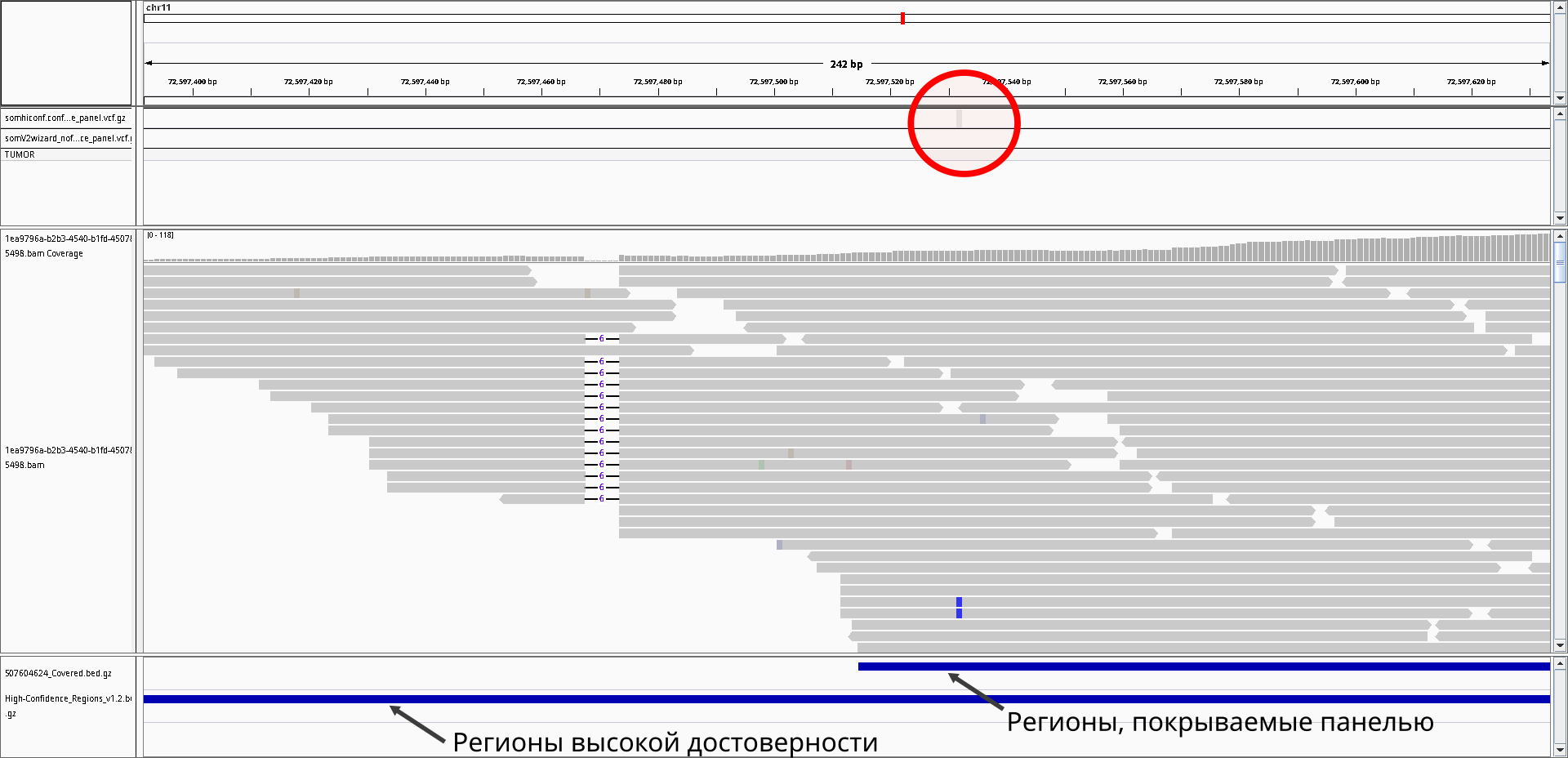
В то же время, незадетектированные варианты иногда могут быть даже при наличии покрывающих участок ридов, т.к. они могут попадать в сложнокартируемые регионы, где, например, нет уникальных ридов или куда попадают неспецифичные риды. Данные консорциума GIAB включают трэки этих "сложных" районов и позволяют оценить чувствительность, выбросив их. Пример варианта, расположенного в "сложном" регионе и незадетектированного коллером, несмотря на имеющиеся риды с полиморфизмом, приведен на рис. 2:
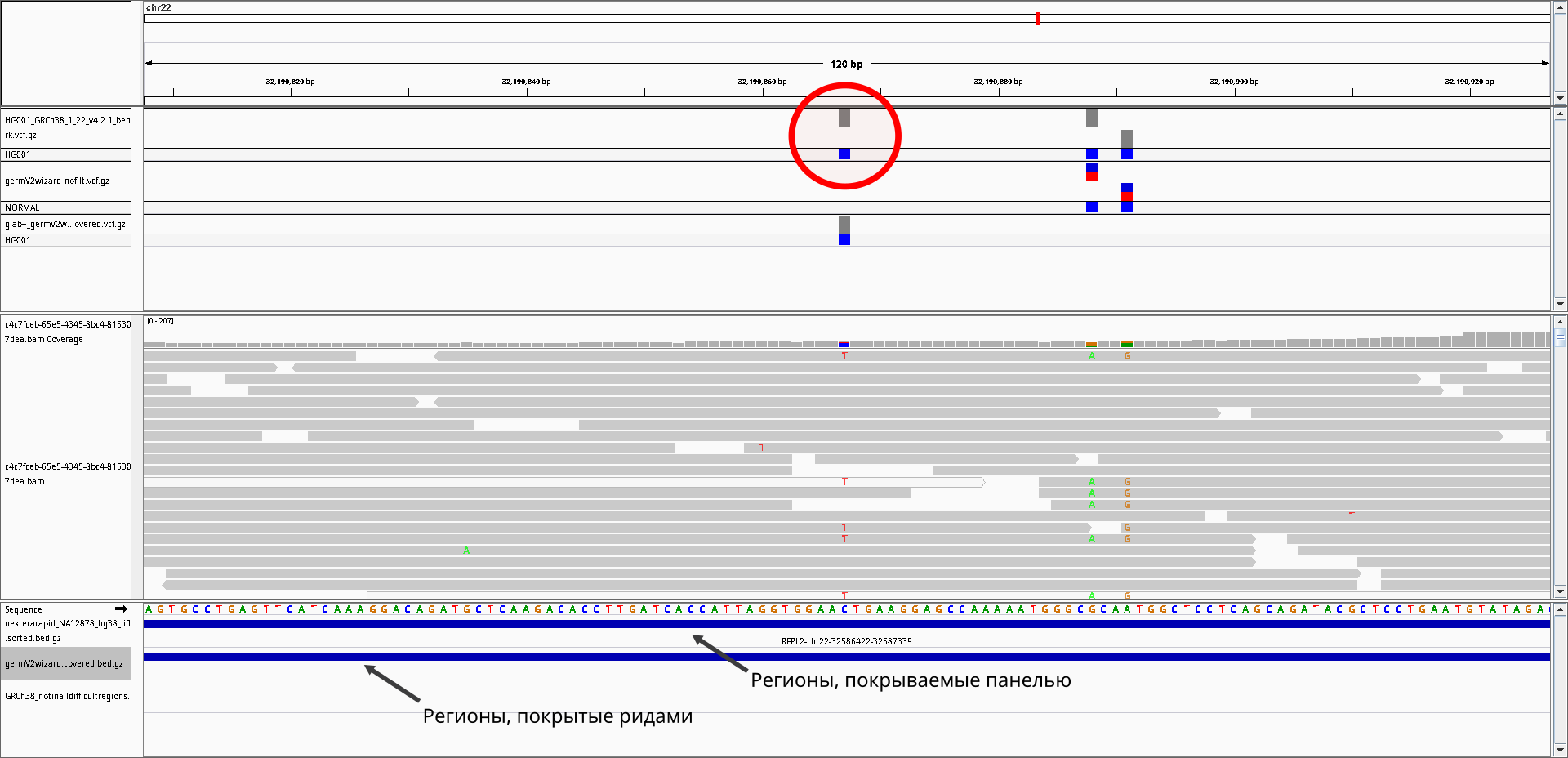
В связи с этими эффектами при подсчете чувствительности можно сузить область рассмотрения до тех регионов, где есть, скажем, хотя бы 2 рида. В таком случае чувствительность возрастает до 97,65% (по сравнению с 93,23%). Или, например, если сузить область до регионов, не входящих в "сложные", чувствительность возрастает до 95,16%. Наконец, если рассмотреть только регионы, в которых имеется ненулевое покрытие (хотя бы 2 рида) и которые не относятся к "сложным" (т.е. пересечение этих двух областей), то чувствительность составляет 99.22%.
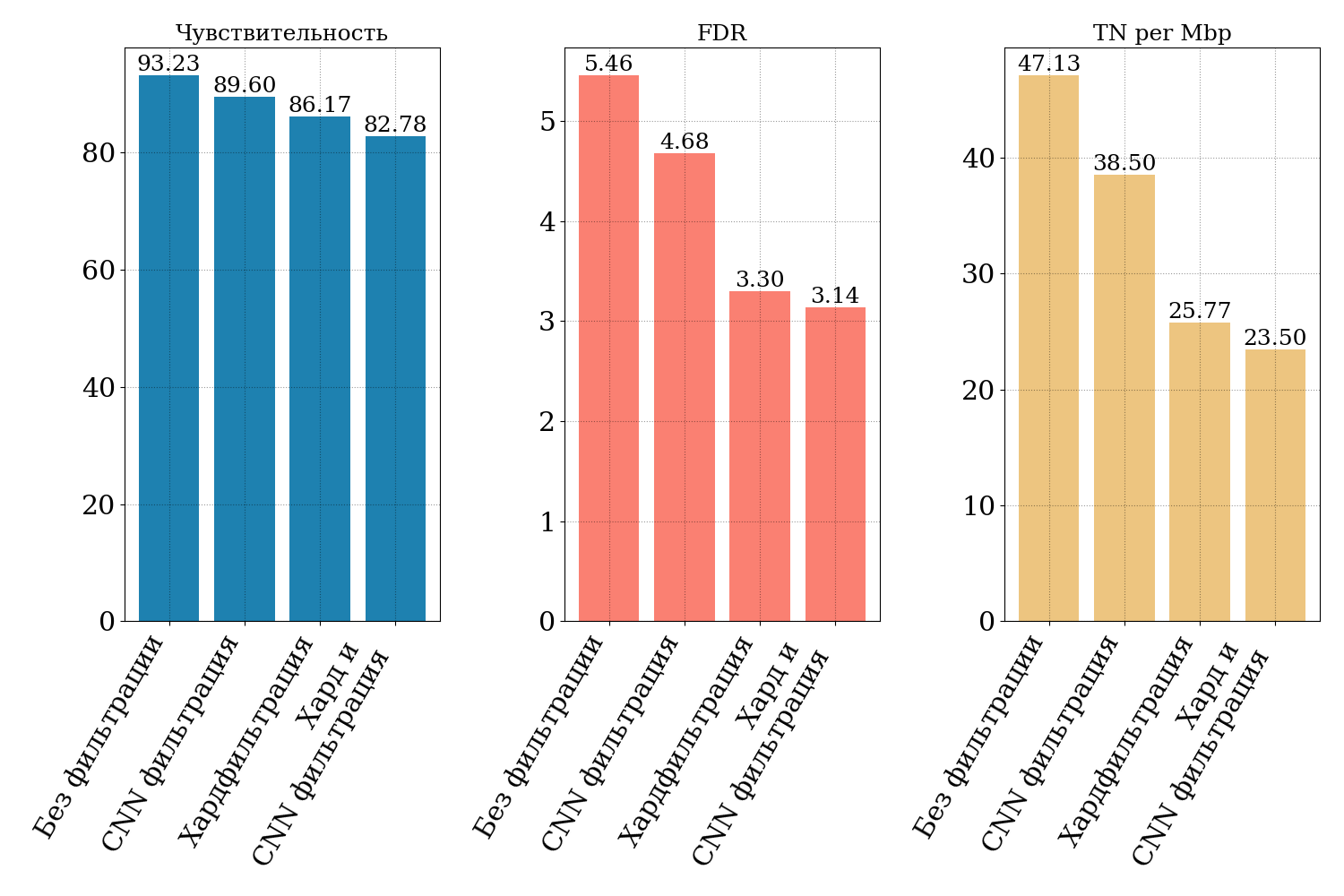
Также важно знать, какое количество ложных вариантов продуцирует пайплайн. Для оценки можно, например, использовать FDR (False discover rate), т.е. долю ложноположительных вариантов среди всех обнаруженных вариантов. Значения для вышеупомянутых регионов можно посмотреть на рис. 4:
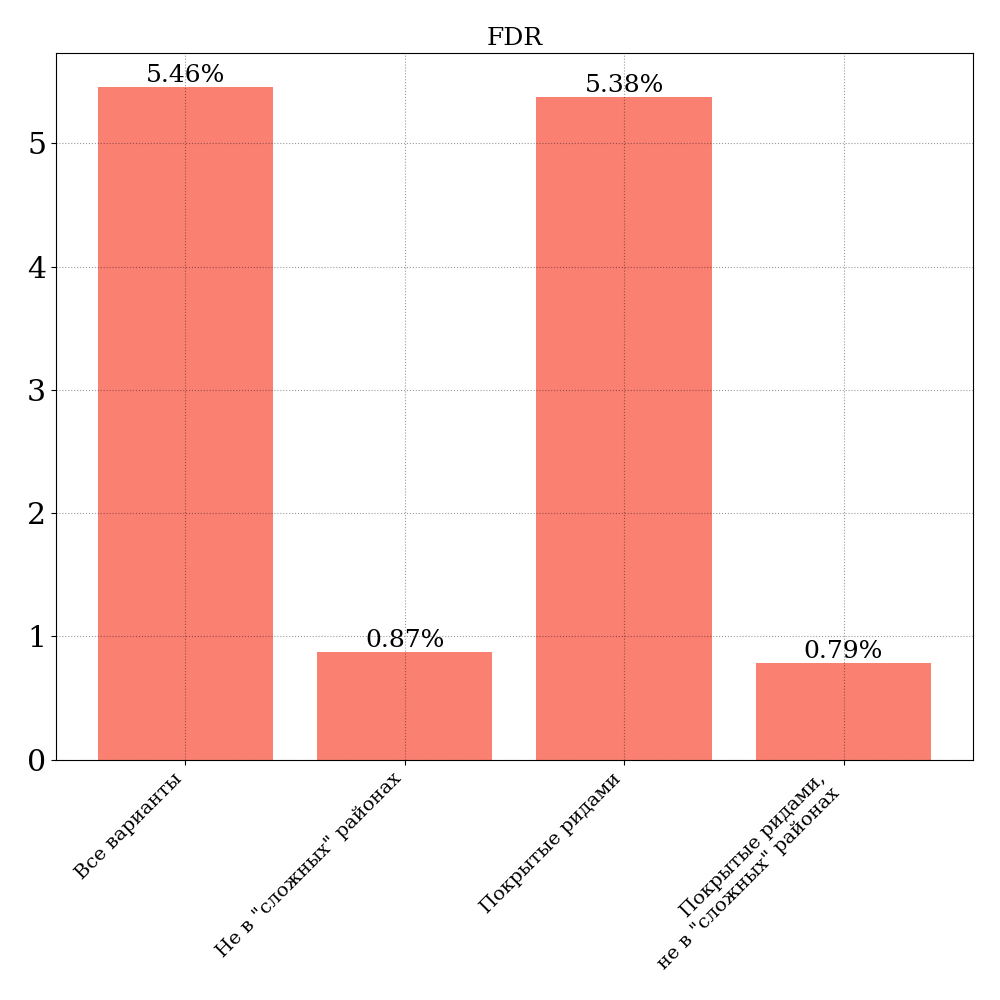
В первую очередь бросается в глаза огромная разница в количестве ложноположительных вариантов между всеми участками, покрытыми панелью, (колонка "Все варианты" на рис. 4) и теми, которые не относятся к "сложным" регионам. Если во всех заявленных регионах панели FDR составляет 5,46% (т.е. более 5% всех обнаруживаемых в этих регионах вариантов - ложноположительные), то с отбрасываением "сложных" регионов FDR падает до 0.87%.
Т.к. в приложениях нам зачастую интересны варианты в проблематичных регионах, то их следует тщательно рассматривать и продумывать методики фильтрации. Далее рассмотрим, как можно уменьшить количество ложноположительных вариантов в регионах панели.
Влияние фильтрации на полученные значения#
В нашей структуре пайлайна по умолчанию используются два типа фильтрации, предлагаемые утилитой GATK: это т.н. хардфильтрация (hard filtering) и фильтрация с помощью нейросетей (CNN подпрограммы).
На рис. 5 можно увидеть чувствительность и FDR для прогонов пайплайна с различными методиками фильтрации. Видно, что FDR удается снизить с 5,46% до 3,30% при использовании хардфильтрации. При этом чувствительность падает с 93,23% до 86,17%. Также видно, что совместное использование хардфильтрации и CNN фильтрации приводит к наименьшему значению FDR, хотя добавочный вклад CNN методов к отфильтровыванию ложноположительных вариантов в дополнение к хардфильтрации невелик (FDR падает с 3,30% до 3,14%).
Количество ложноположительных вариантов, отнесенное к исследуемой геномной дистанции в млн пар оснований, в целом повторяет картину FDR:
Тестирование соматического пайплайна#
Для детекции соматических вариантов нами используется коллер Mutect2 из тулов GATK.
Тестовые образцы#
В качестве источника стандартных образцов, содержащих известный набор соматических мутаций, мы использовали данные консорциума SEQC2 (2Fang et al., 2021). Его подход в некоторой степени аналогичен подходу, примененному для создания стандартов детекции герминальных вариантов, описанному выше.
Этот консорциум содержит как данные полногеномного секвенирования, так и полноэкзомные данные. При этом и те, и другие секвенировались в различных местах и институтах. Нами использовались данные архива SRR7890883 (WES_IL_T_1) в качестве образца опухоли и данные архива SRR7890874 (WES_IL_N_1) в качестве соответствующего нормального образца. Проводился как анализ опухолевого образца в т.н. tumor-only режиме работы пайплайна (без соответствующего неопухолевого или "нормального" образца), так и анализ в режиме tumor-normal (с применением неопухолевого образца).
Сравнение с "золотыми" вариантами#
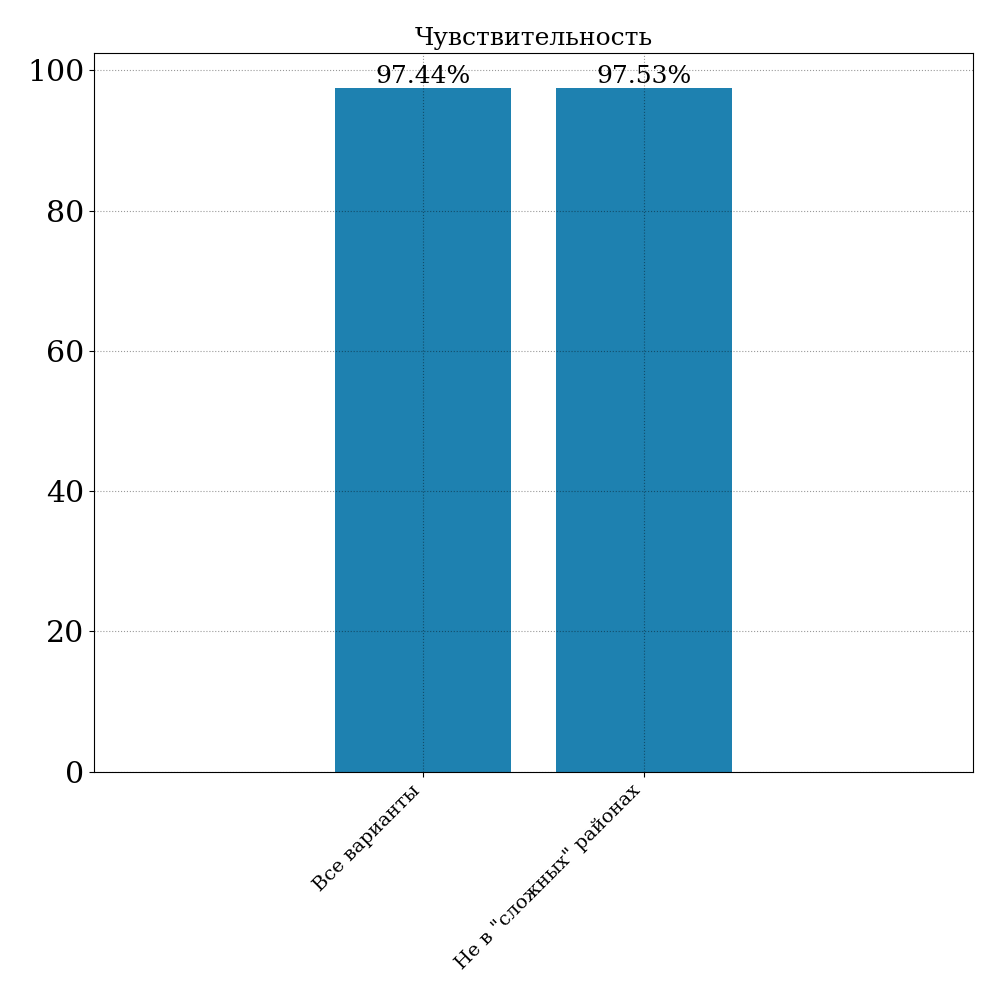
При сравнении вариантов внутри регионов надежности и покрытия панели чувствительность составила 97.44% для пайплайна в режиме tumor-only. Рассмотрение незадетектированных вариантов показывает, что основной причиной ложноотрицательных результатов является низкая частота мутаций. Пример такого незадетектированного варианта приведен на рис. 6:
Эта черта характерна именно для соматического пайплайна, но не для герминального. В случае герминальных вариантов мы ожидаем хотя бы половину молекул ДНК содержать вариант (для гетерозиготы), однако доля молекул, несущих соматический вариант, может быть очень мала.
Применение анализа регионов "сложности" от консорциума "Genome in a Bottle" показывает, что сужение области анализа до "несложных" регионов позволяет повысить чувствительность до 97.53%:
Специфичность, фильтрация и использование неопухолевого образца#
Попытка посчитать метрики, характеризующие ложноположительные
результаты (например, FDR), приводит к очень высоким относительным
количествам ложных результатов в tumor-only анализе, что связано с тем,
что герминальных вариантов намного больше, чем соматических.
Фильтрация с помощью баз известных вариантов (dbSNP) и
утилит вроде FilterMutectCalls позволяет частично
отфильтровать эти варианты.
Конечно, лучше всего для этого пользоваться анализом в режиме tumor-normal, хотя в клинической практике далеко не всегда имеется такая возможность.
Результаты по чувствительности и FDR обоих режимов пайплайна с использованием фильтрации и без показаны на рис. 8:
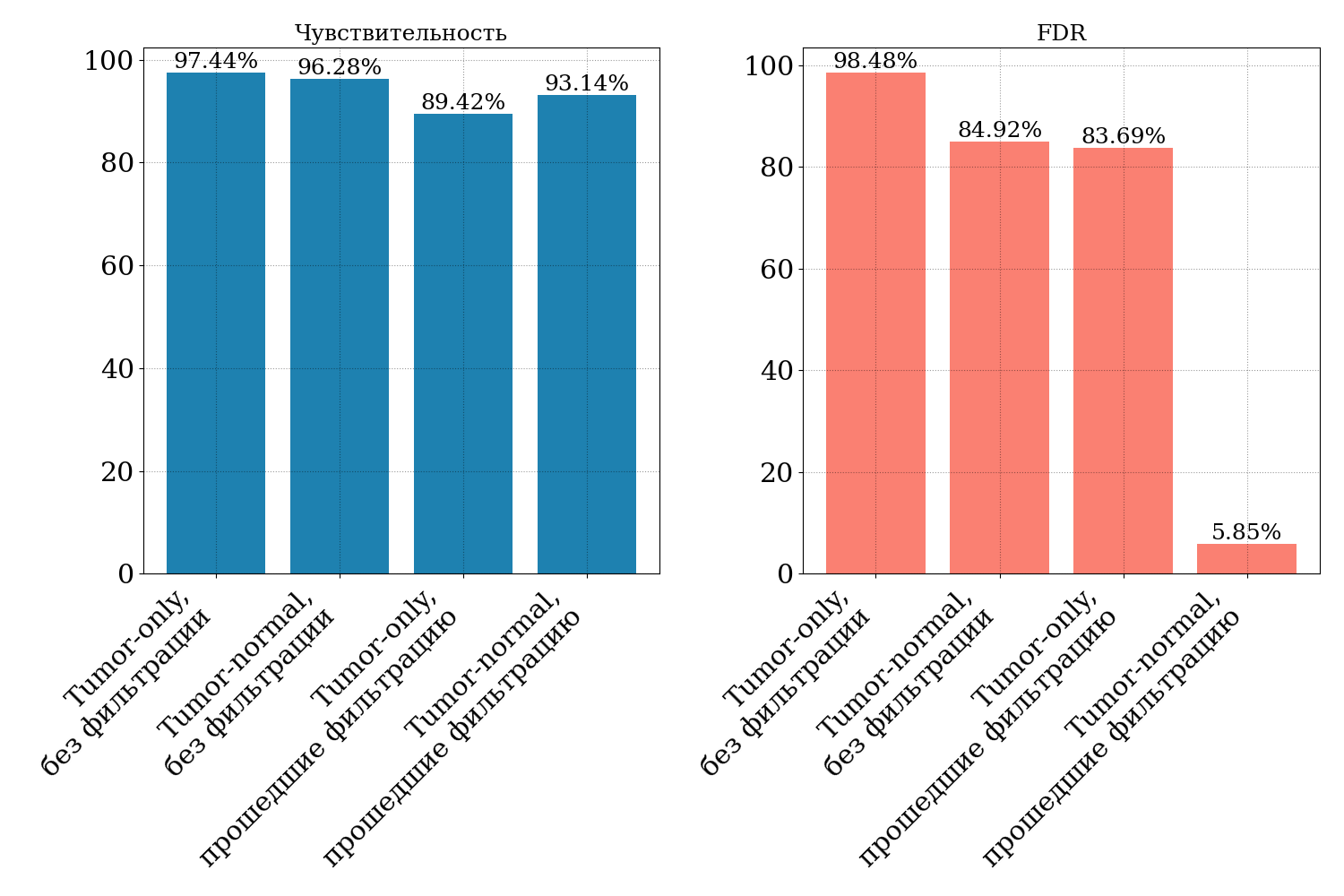
По графикам видно, что применение фильтрации (используя все доступные
фильтры FilterMutectCalls) понижает чувствительность как в
tumor-only, так и в tumor-normal режиме анализа. Например, для
tumor-only анализа чувствительность падает с 97,44% до 89,42%.
FDR имеет наибольшее значение для tumor-only анализа без фильтрации (98,48%). Это означает, что из всех обнаруживаемых с помощью Mutect2 вариантов лишь малая часть (менее 2%) является истинно соматическими вариантами. FDR падает до уровня 5,85% при использовании сиквенсов неопухолевой ткани. Применение же методов фильтрации in silico понижает FDR лишь незначительно до уровней выше 80%.
Команды, использованные при генерации файлов#
Сравнение вариантов производилось утилитой bcftools
isec. Предварительно файлы нормализовались с помощью bcftools
norm, чтобы унифицировать представление вариантов. Кроме того, VCF
"сужались" до необходимых регионов с помощью bedtools intersect.
Например, для сравнения герминальных "золотых вариантов", прошедших хардфильтрацию и CNN фильтрацию, нужны следующие команды (использовались при построении рис. 5):
cat pipeline_output.vcf | bcftools norm -a -f reference.fasta -m -both -O v | bcftools norm -d exact -f reference.fasta -O v | bcftools sort > norm_pipeline_out.vcfbedtools intersect -u -header -a norm_pipeline_out.vcf -b nexterarapid_NA12878_hg38_lifted.regchrom.sorted.bed | bedtools intersect -u -header -a - -b HG001_GRCh38_1_22_v4.2.1_benchmark.bed | bgzip -c > norm_pipeline_out.considered_regions.vcf.gzbcftools view --exclude 'FILTER~"HDFLT_FS"||FILTER~"HDFLT_MQ"||FILTER~"HDFLT_MQRankSum"||FILTER~"HDFLT_QD"||FILTER~"HDFLT_QUAL"||FILTER~"HDFLT_ReadPosRankSum"||FILTER~"HDFLT_SOR"||FILTER~"CNN_2D_INDEL_Tranche_99.40_100.00"||FILTER~"CNN_2D_SNP_Tranche_99.95_100.00"' norm_pipeline_out.considered_regions.vcf.gz | bgzip -c > norm_pipeline_out_HARDnCNN.considered_regions.vcf.gztabix norm_pipeline_out_HARDnCNN.considered_regions.vcf.gzgunzip -c HG001_GRCh38_1_22_v4.2.1_benchmark.vcf.gz | bcftools norm -a -f reference.fasta -m -both -O v | bcftools norm -d exact -f reference.fasta -O v | bcftools sort | bgzip -c > HG001_GRCh38_1_22_v4.2.1_benchmark.norm.vcf.gzbedtools intersect -u -header -a HG001_GRCh38_1_22_v4.2.1_benchmark.norm.vcf.gz -b nexterarapid_NA12878_hg38_lifted.regchrom.sorted.bed | bedtools intersect -u -header -a - -b HG001_GRCh38_1_22_v4.2.1_benchmark.bed | bgzip -c > giab.considered_regions.vcf.gzbcftools isec norm_pipeline_out_HARDnCNN.considered_regions.vcf.gz giab.considered_regions.vcf.gz -p norm_pipeline_out_HARDnCNN_vs_giabНа выходе получается папка norm_pipeline_out_HARDnCNN_vs_giab,
содержащая общие варианты и варианты, уникальные для полученного и
скачанного файлов VCF.
Здесь использовались следующие файлы, полученные с FTP проекта GIAB:
HG001_GRCh38_1_22_v4.2.1_benchmark.vcf.gz- файл VCF с вариантами;HG001_GRCh38_1_22_v4.2.1_benchmark.bed- регионы высокой достоверности;nexterarapid_NA12878_hg38_lifted.regchrom.sorted.bed- файл панели, примененной при получении использованных здесь файлов.
При исследовании соматических вариантов используется аналогичный подход. Например, чтобы сравнить фильтрованные варианты с вариантами "золотого" стандарта, нужны следующие команды (использовались при построении рис. 8):
cat pipeline_output.vcf | bcftools norm -a -f reference.fasta -m -both -O v | bcftools norm -d exact -f reference.fasta -O v | bcftools sort > norm_pipeline_out_nofilt.vcfbedtools intersect -u -header -a norm_pipeline_out_nofilt.vcf -b S07604624_Covered.bed | bedtools intersect -u -header -a - -b High-Confidence_Regions_v1.2.bed | bgzip -c > norm_pipeline_out_nofilt.considered_regions.vcf.gzbcftools view -f .,PASS norm_pipeline_out_nofilt.considered_regions.vcf.gz | bgzip -c > norm_pipeline_out_filt.considered_regions.vcf.gztabix norm_pipeline_out_filt.considered_regions.vcf.gzbcftools merge high-confidence_sINDEL_in_HC_regions_v1.2.vcf.gz high-confidence_sSNV_in_HC_regions_v1.2.vcf.gz | bcftools norm -f reference.fasta -m -both -O v | bcftools norm -d exact -f reference.fasta -O v | bcftools sort > somhiconf.vcfbedtools intersect -u -header -a somhiconf.vcf -b S07604624_Covered.bed | bedtools intersect -u -header -a - -b High-Confidence_Regions_v1.2.bed | bgzip -c > somhiconf.considered_regions.vcf.gztabix somhiconf.considered_regions.vcf.gzbcftools isec norm_pipeline_out_filt.considered_regions.vcf.gz somhiconf.considered_regions.vcf.gz -p norm_pipeline_out_filt_vs_somhiconfАналогично, здесь использовались следующие файлы, полученные с FTP проекта GIAB:
high-confidence_sINDEL_in_HC_regions_v1.2.vcf.gzиhigh-confidence_sSNV_in_HC_regions_v1.2.vcf.gz- варианты высокой достоверности;High-Confidence_Regions_v1.2.bed- регионы высокой достоверности;S07604624_Covered.bed- регионы панели Sureselect V7.