Настройки пользователя
На странице "Настройки пользователя" находятся ваши персональные настройки: настройки аккаунта (email, логин, пароль), настройки панели навигации Программы, страницы пациента, SNV Viewer и CNV Viewer.
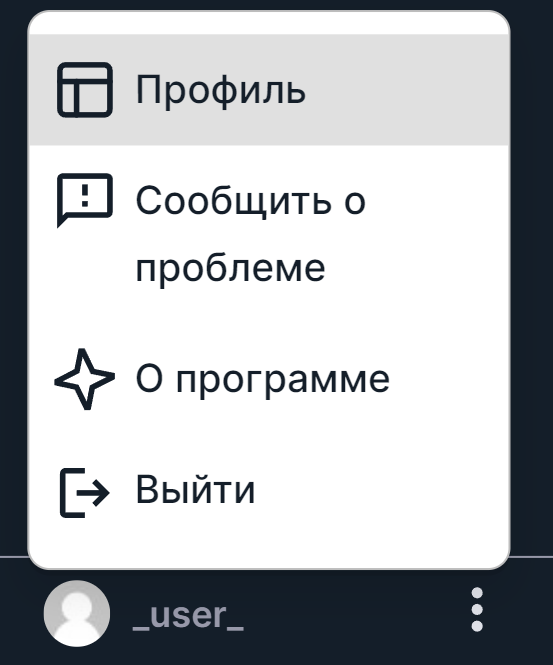
Чтобы открыть страницу "Настройки пользователя", наведите курсор мыши на ваш логин внизу боковой
панели навигации и нажмите на появивишиеся
три точки . В открывшемся
меню выберите "Профиль":
Вы увидите страницу с настройками:
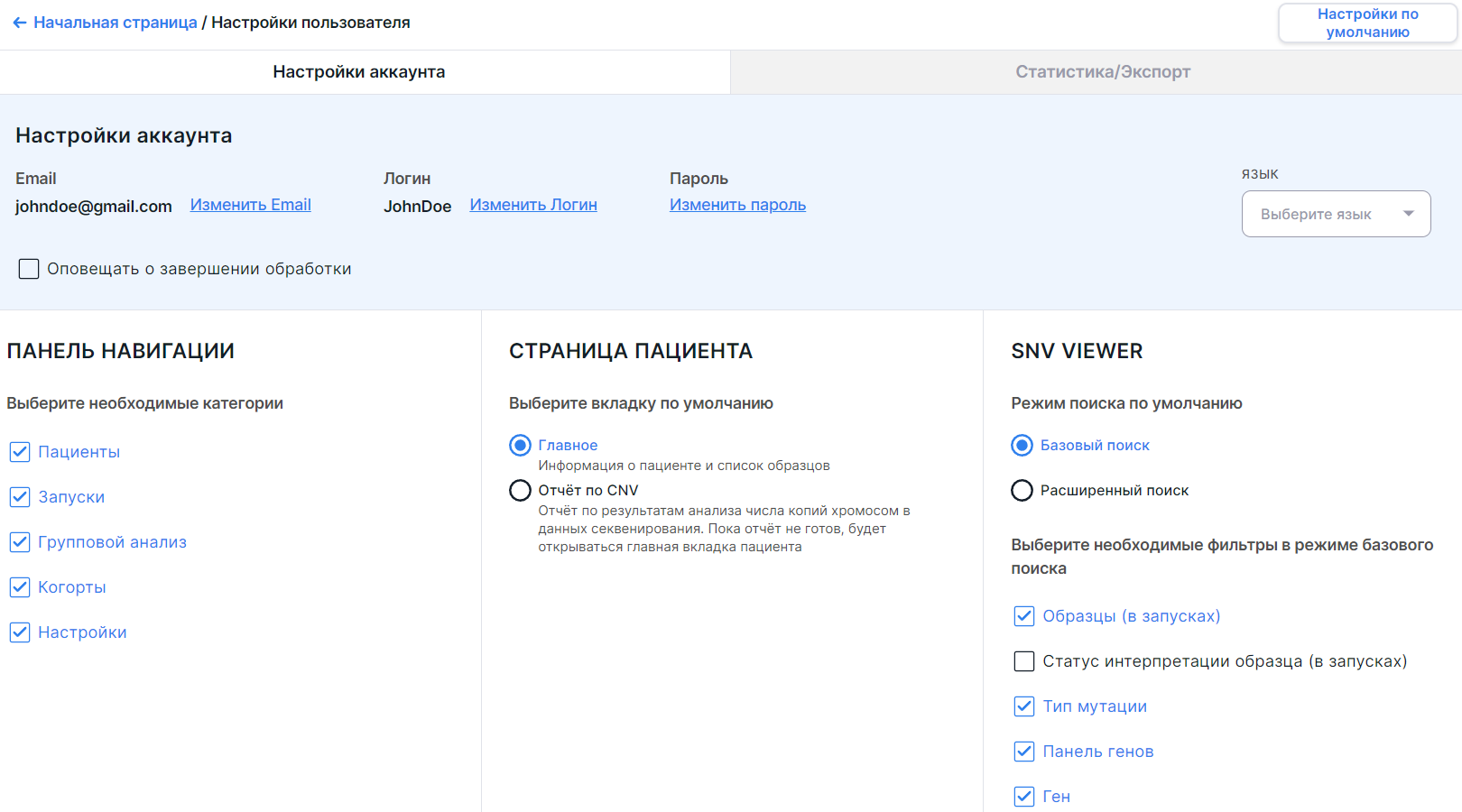
Настройки аккаунта#
Адрес электронной почты#
Здесь вы можете посмотреть, какой email вы использовали при регистрации в Системе.
Для того чтобы его поменять, нажмите на ,
а затем в открывшемся окне введите новый адрес почты и ваш действующий пароль аккаунта для подтверждения
действия:
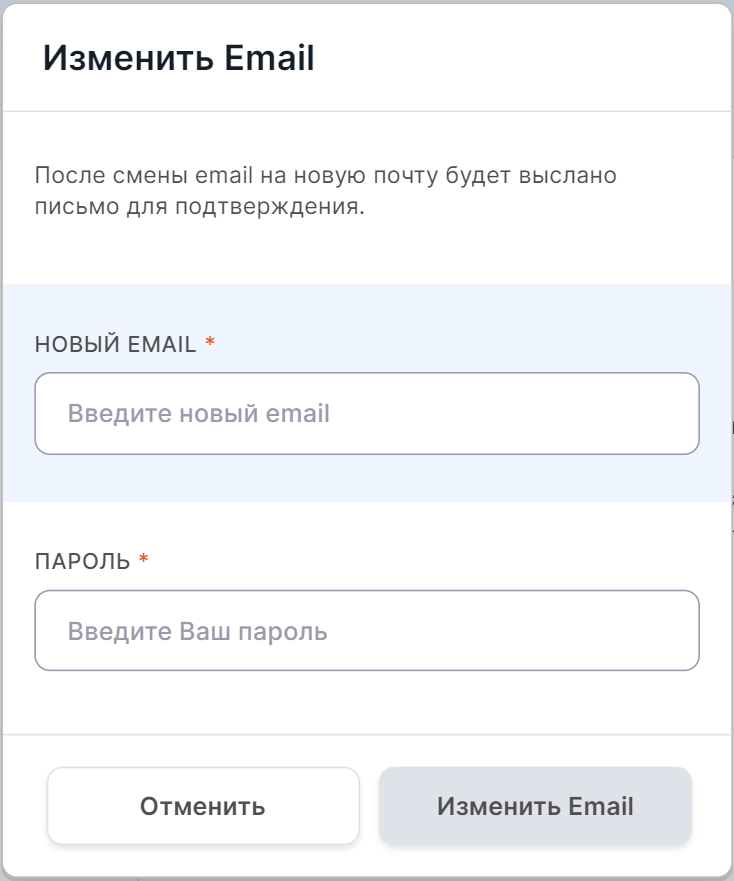
После заполнения формы нажмите на .
Теперь проверьте ящик той почты, которую вы указали как новую, и подтвердите смену адреса в письме.
Логин#
Здесь вы можете посмотреть, какой логин вы использовали при регистрации в Genomenal.
Для того чтобы его поменять, нажмите на ,
а затем в открывшемся окне введите новый логин и ваш действующий пароль аккаунта для подтверждения
действия:
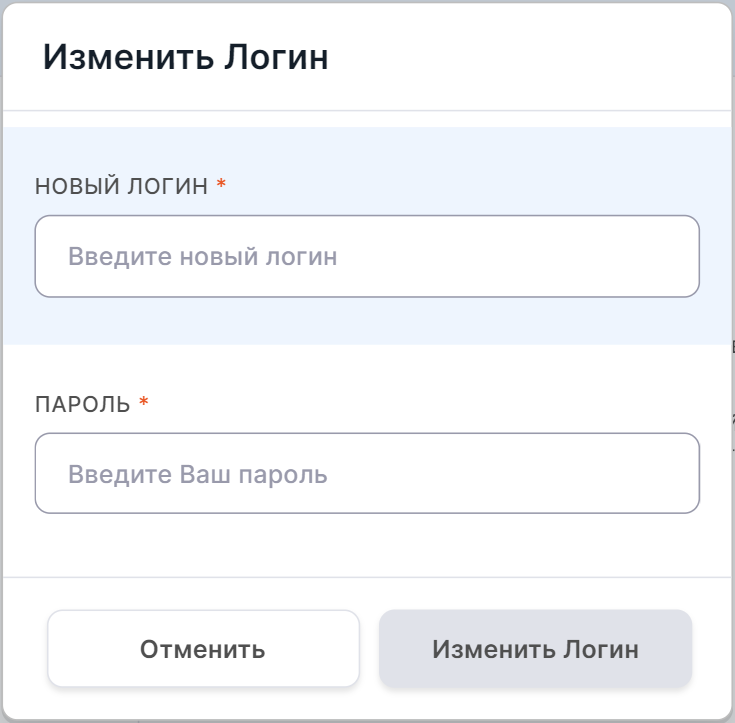
После заполнения формы нажмите на .
Логин успешно изменен!
Логин может состоять не более, чем из 128 любых символов.
Пароль#
Чтобы поменять пароль для входа в Систему,
нажмите на ,
а затем в открывшемся окне введите ваш действующий пароль аккаунта для подтверждения
действия и два раза - новый пароль:
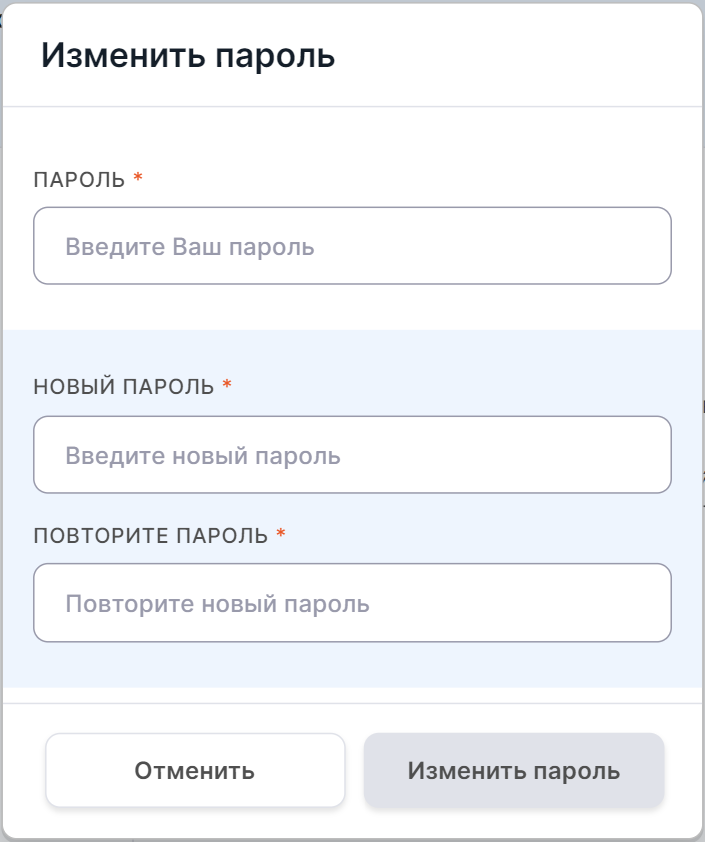
После заполнения формы нажмите на .
Пароль успешно изменен!
Пароль может состоять не более, чем из 128 любых символов.
Язык Программы#
Здесь вы можете выбрать удобный для вас язык Программы. Для этого нажмите на поле выбора языка и выберите нужный язык в выпавшем списке:
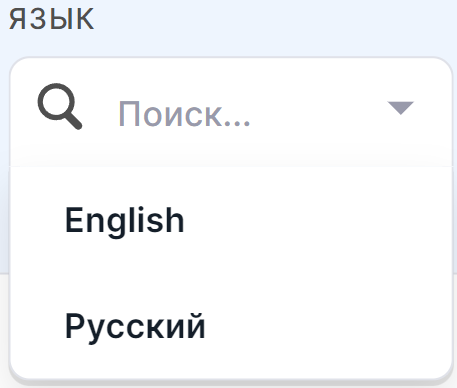
Оповещение о завершении обработки#
Здесь можно настроить получение оповещений о завершении обработки образцов и групповых анализов. Оповещения о завершении обработки образцов отправляются, когда завершилась обработка всех образцов, загруженных в одного пациента или один запуск. При перезапуске анализа образца или группового анализа также придёт письмо о завершении обработки. Оповещения приходят на электронную почту, указанную в настройках аккаунта. В письме вы найдёте ссылку на страницу пациента или запуска, обработка образцов которого завершилась, либо ссылку на завершённый групповой анализ. Для пациентов и запусков будут приведены ссылки на образцы, обработка которых завершилась - успешно или с ошибкой. Чтобы получать оповещения о завершении обработки образцов и групповых анализов, включите соответствующую опцию "Оповещать о завершении обработки".
Панель навигации#
В разделе отображено то, какие блоки страниц показаны на боковой панели навигации в Программе помимо начальной страницы "Образцы" (по умолчанию выбраны все):
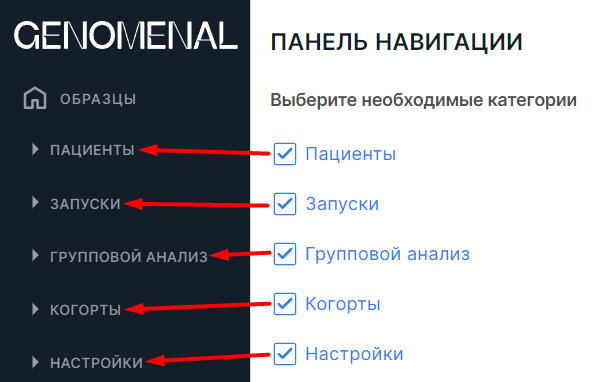
Чтобы убрать ненужный блок из панели, снимите с него галочку.
Страница пациента#
На странице пациента может быть две вкладки - "Главное" и "Отчёт по CNV", если "Отчёт по CNV" был построен (см. условия в соответствующем разделе):
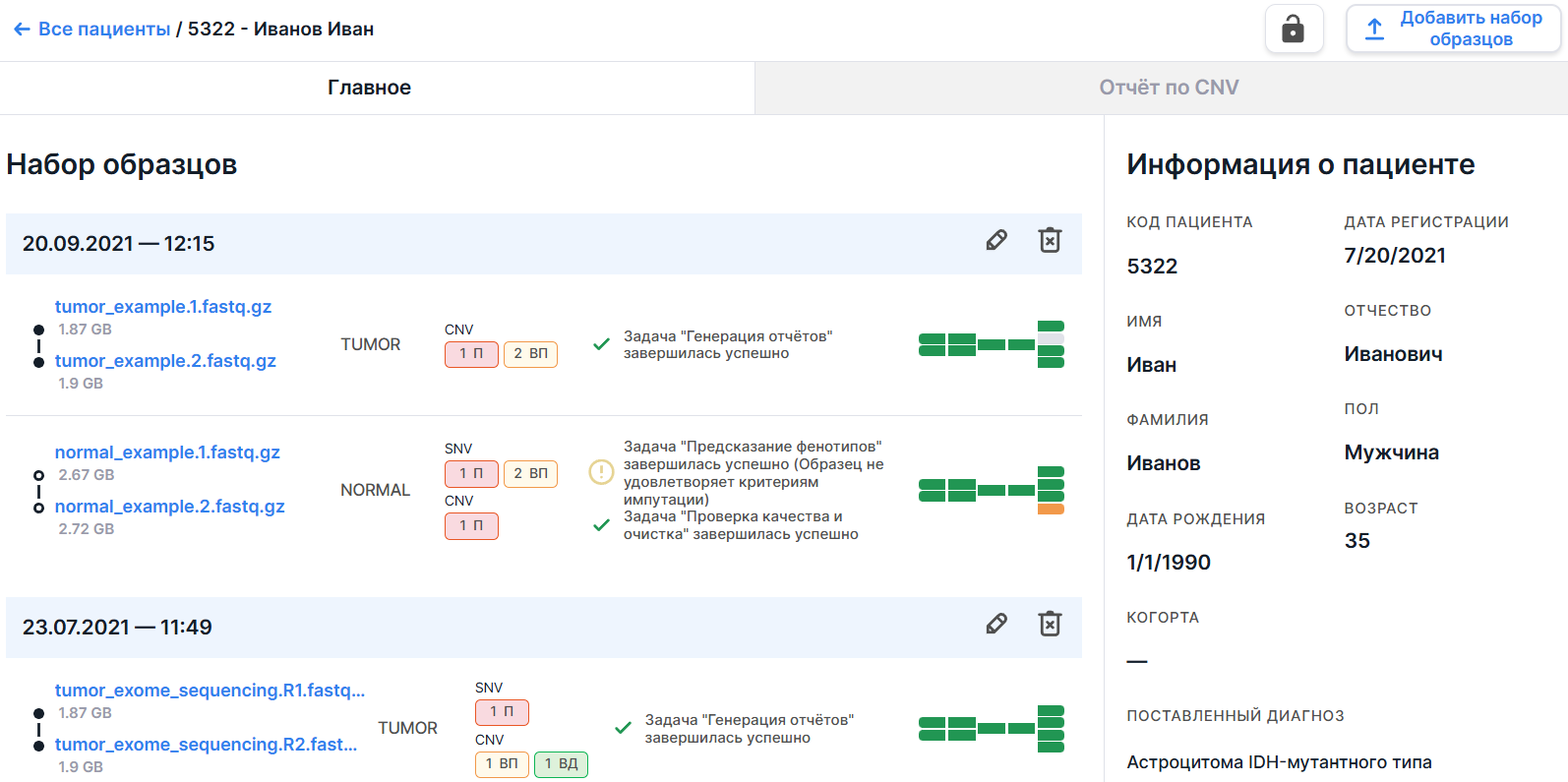
В данном разделе можно настроить, на какую вкладку вы будете попадать, когда открываете страницу пациента:
- на вкладку "Главное", на которой находятся информация о пациенте и наборы загруженных образцов (по умолчанию);
- на вкладку "Отчёт по CNV" с отчётом по результатам анализа числа копий хромосом в образцах пациента.

Внимание!
Если "Отчёт по CNV" для пациента не был построен совсем или ещё строится, то даже если вы здесь выбрали приоритетной вкладку "Отчёт по CNV", открываться будет вкладка "Главное".
SNV Viewer#
SNV Viewer - это встроенный модуль для просмотра и анализа вариантов. Подробно описан в соответствующем разделе.
Режим поиска#
В SNV Viewer есть два режима поиска вариантов: базовый поиск с основными параметрами фильтрации и расширенный поиск, в котором есть больше параметров фильтрации и можно составлять сложные запросы с несколькими условиями фильтрации.
В данном разделе можно настроить, в какой режим поиска вы будете попадать, когда впервые открываете страницу SNV Viewer образца (по умолчанию открывается базовый поиск):

Основные параметры фильтрации в режиме базового поиска#
Параметры фильтрации, выбранные по умолчанию, описаны здесь:

Выбор параметров можно поменять (убрать или добавить новые):
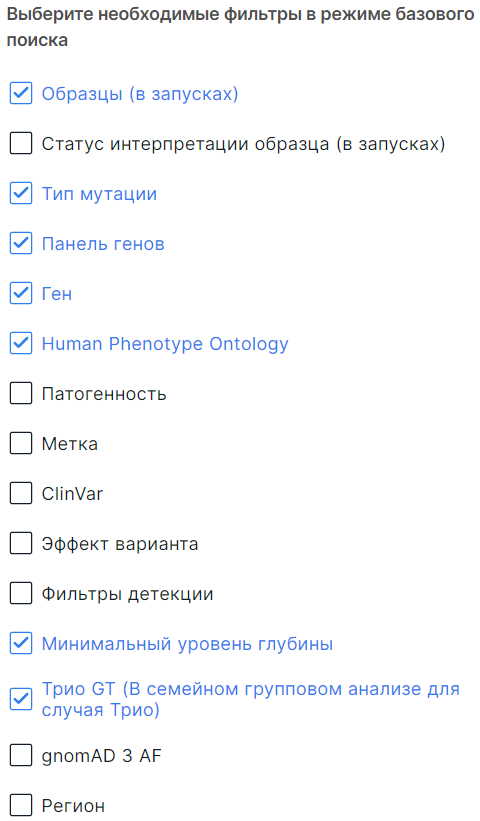
Доступные параметры фильтрации:
- Образцы - фильтр вариантов по образцам из запуска, в которых эти варианты были выявлены. Отображается только в SNV Viewer запуска. Добавлен по умолчанию.
- Статус интерпретации образца - фильтр вариантов по статусу интерпретации образцов запуска, в которых они были выявлены, (завершенная или незавершенная интерпретация). Отображается только в SNV Viewer запуска.
- Тип мутации - фильтр вариантов по типу: Somatic (соматические) или Germline (герминальные). Добавлен по умолчанию. Не отображается в базовом режиме поиска в SNV Viewer с герминальными мутациями и в SNV Viewer группового анализа.
- Панель генов - фильтр вариантов по панели генов. Добавлен по умолчанию.
- Ген - фильтр вариантов по транскриптам генов, в которых они расположены. Добавлен по умолчанию.
- Human Phenotype Ontology (HPO) - фильтр вариантов по генам, ассоциированным с терминами из словаря фенотипических аномалий человека HPO. Добавлен по умолчанию.
- Патогенность - фильтр вариантов по патогенности, определенной пользователем самостоятельно, по критериям ACMG или по базе патогенности:
- Pathogenic - Патогенный;
- Likely pathogenic - Вероятно патогенный;
- Uncertain significance conflict - Противоречивые интерпретации патогенности;
- Uncertain significance - Неопределенная значимость;
- Likely benign - Вероятно доброкачественный;
- Benign - Доброкачественный;
- Undefined - Нет интерпретации.
- Метка - фильтр вариантов по их значимости, определенной пользователем:
- High significance - Высокая значимость;
- Moderate significance - Средняя значимость;
- Low significance - Низкая значимость;
- Sequencing error - Ошибка секвенирования;
- Undefined - Неопределенная значимость.
- ClinVar - фильтр вариантов по клинической значимости фенотипа по данным базы ClinVar (определение значений можно посмотреть тут).
- Эффект варианта - фильтр вариантов по эффектам на гены. Значения описаны здесь.
- Фильтры детекции - фильтрация по фильтрам вариантов GATK.
Нажмите, чтобы посмотреть определения фильтров детекции
| Значение фильтра детекции | Тип мутации | Инструмент GATK | Описание фильтра |
| alignment | Соматическая | FilterMutectCalls | Фильтр артефактов выравнивания |
| base_qual | Соматическая | FilterMutectCalls | Среднее качество оснований прочтений, поддерживающих альтернативный аллель |
| clustered_events | Соматическая | FilterMutectCalls | Кластерные события, наблюдаемые в опухоли, - соматические мутации, которые совместно встречаются в одном и том же интервале сборки |
| CNN_2D | Герминальная | CNNScoreVariants | Фильтрация на основе оценок предварительно обученной сверточной нейронной сети (convolutional neural network; CNN) 2D |
| contamination | Соматическая | FilterMutectCalls | Фильтр контаминации данных секвенирования |
| duplicate | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля чрезмерно представлены очевидными дубликатами |
| FAIL | Соматическая | FilterMutectCalls | Ошибка сайта, если все аллели не проходят по разным причинам |
| fragment | Соматическая | FilterMutectCalls | Разница медианных длин фрагментов прочтений, поддерживающих альтернативный и референсный аллели |
| FS | Герминальная | FisherStrand | Вероятность смещения цепей на сайте, оцененная с помощью точного критерия Фишера. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. FisherStrand определяет, существует ли смещение цепи между прямой и обратной цепями для референсного или альтернативного аллеля (то есть встречается ли альтернативный аллель чаще или реже на прямой или обратной цепи, чем референсный аллель). FisherStrand хуже учитывает большие объемы данных в ситуациях с высоким покрытием по сравнению с StrandOddsRatio. Кроме того, FisherStrand имеет тенденцию "штрафовать" варианты, которые встречаются на концах экзонов. Варианты на концах экзонов, как правило, покрываются прочтениями только в одном направлении цепи, и FisherStrand дает таким вариантам плохую оценку, тогда как StrandOddsRatio учитывает соотношение прочтений, охватывающих оба аллеля. |
| germline | Соматическая | FilterMutectCalls | Доказательства указывают на то, что этот сайт является герминальным, а не соматическим |
| haplotype | Соматическая | FilterMutectCalls | Вариант расположен рядом с отфильтрованным вариантом того же гаплотипа |
| low_allele_frac | Соматическая | FilterMutectCalls | Доля аллеля ниже установленного порога |
| LowQual | Соматическая | FilterMutectCalls | Низкое качество |
| map_qual | Соматическая | FilterMutectCalls | Среднее качество картирования прочтений, поддерживающих альтернативный аллель |
| MQ | Герминальная | RMMSMapping- Quality | Среднеквадратичное значение качества сопоставления прочтений по всем образцам. Представляет собой оценку общего качества картирования прочтений, покрывающих сайт варианта, усредненную по всем образцам в когорте, т.е. квадратный корень среднего квадрата качества картирования сайта варианта. Таким образом, оценка учитывает стандартное отклонение качества картирования. |
| MQ_RANK_SUM | Герминальная | MappingQuality- RankSumTest | Тест суммы рангов для сопоставления качества картирования прочтения REF и качества картирования прочтения ALT. MQRankSum на уровне варианта сравнивает качество картирования прочтений, поддерживающих референсный аллель, с прочтениями, поддерживающими альтернативный аллель. Обнаружение любой статистически значимой разницы в качестве предполагает, что процесс секвенирования и/или картирования мог быть искажен артефактом. |
| mt_many_low_hets | Соматическая | FilterMutectCalls | Фильтр сайтов с низкой гетероплазмией |
| multiallelic | Соматическая | FilterMutectCalls | Мутация располагается в мультиаллельном сайте: слишком много альтернативных аллелей удовлетворяют LOD score (вероятный признак артефакта) |
| n_ratio | Соматическая | FilterMutectCalls | Отношение количества неизвестных нуклеотидов N к количеству альтернативных аллелей превышает установленный порог |
| normal_artifact | Соматическая | FilterMutectCalls | Нормальный артефакт: вариант, наблюдаемый в опухоли, также присутствует как артефакт в контрольном образце |
| Not available | Любая | Значение колонки неизвестно (точка в VCF-файле) | |
| numt_chimera | Соматическая | FilterMutectCalls | Вариант, расположенный в ядерной копии гена мтДНК (NuMT), со слишком большим количеством прочтений, поддерживающих альтернативный аллель, происходящих из аутосомы |
| numt_novel | Соматическая | FilterMutectCalls | Глубина альтернативного аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| orientation | Соматическая | FilterMutectCalls | Фильтр смещения ориентации - смещения, возникающего из-за химического изменения нуклеотида во время подготовки библиотеки, которое приводит к неправильному спариванию основанию (G с A, T c C и т.д.) (артефакт секвенирования) |
| OTHER | Любая | Значение колонки не распознано | |
| panel_of_normals | Соматическая | FilterMutectCalls | Сайт находится в "черном списке" нормальной панели (PON). Как выбрать панель, описано здесь. |
| PASS | Любая | Сайт содержит минимум один аллель, который удовлетворяет всем фильтрам | |
| position | Соматическая | FilterMutectCalls | Медианное расстояние альтернативных мутаций от конца прочтения |
| possible_numt | Соматическая | FilterMutectCalls | Глубина аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| QD | Герминальная | QualByDepth | Достоверность варианта (значение из поля QUAL), нормализованная по нефильтрованнной глубине аллеля (AD) варианта в выборке вариантов с генотипом, отличным от hom-ref (homozygous/reference; генотип, при котором оба аллеля являются референсными) |
| QUAL | Герминальная | Качество варианта - вероятность существования (по шкале Phred) варианта в этом сайте с учетом данных секвенирования | |
| READ_POS_RANK_SUM | Герминальная | ReadPosRank- SumTest | Тест суммы рангов для относительных позиций аллелей REF и ALT в прочтениях. ReadPosRankSum на уровне варианта проверяет, различаются ли положения референсного и альтернативного аллеля в прочтениях, которые поддерживают вариант. Нахождение аллеля только вблизи концов прочтений указывает на ошибку, потому что именно там секвенаторы склонны делать больше всего ошибок. Обнаружение статистически значимой разницы в относительном положении в любом случае указывает на то, что процесс секвенирования мог быть искажен артефактом. |
| SITE | Соматическая | FilterMutectCalls | Ошибка сайта |
| slippage | Соматическая | FilterMutectCalls | Фильтр ошибок проскальзывания полимеразы в ходе ДНК-репликации (предсказывается по количеству и длине коротких тандемных повторов (STR)) |
| SOR | Герминальная | StrandOddsRatio | Смещение цепей, оцененное с помощью симметричного теста отношения шансов. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. StrandOddsRatio оценивает наличие смещения между прямыми и обратными цепями для референсного или альтернативного аллеля (аллелей). |
| strand_bias | Соматическая | FilterMutectCalls | Фильтр смещения цепей - смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим (артефакт секвенирования) |
| strict_strand | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля не представлены в обоих направлениях прочтения |
| weak_evidence | Соматическая | FilterMutectCalls | Мутация не соответствует порогу вероятности |
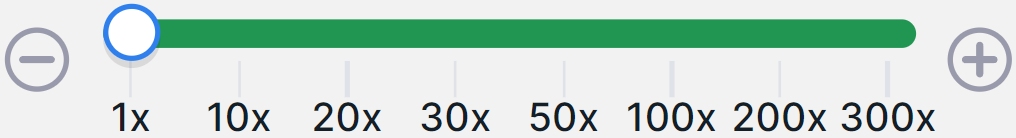
- Минимальный уровень глубины - шкала фильтрации вариантов по минимальному уровеню глубины секвенирования. Добавлена по умолчанию.
- Трио GT - фильтр вариантов по наследованию: De novo варианты, Новые гомозиготы (гомозиготы по локусам у пробанда, которые гетерозиготны у родителей) или Наследованные от одного родителя. Добавлен по умолчанию, доступен только в семейном групповом анализе в случае Трио (Пробанд, Отец и Мать).
- gnomAD 3 AF - фильтр вариантов по частоте аллеля по данным базы gnomAD (версия 3):
- <0.05 (граница между распространёнными и редкими вариантами) или пусто (нет данных о частоте в gnomAD);
- <0.02 (условная граница гипоморфов) или пусто;
- <0.01 (расчётная граница аутосомно-рецессивного наследования) или пусто;
- <0.005 (расчётная граница X-сцепленного наследования) или пусто;
- <0.0001 (расчётная граница аутосомно-доминантного наследования) или пусто.
- Регион - фильтр вариантов по позиции на геноме (хромосома, стартовая и конечная позиции).
Предпочитаемая модель транскриптов генов#
На вкладке "Gene" панели детальной информации о варианте доступны транскрипты гена из двух баз данных: Ensembl и RefSeq. По умолчанию показана таблица с транскриптами из Ensembl.
В данном разделе можно настроить, какая модель транскриптов генов будет показана по умолчанию:

Условия расширенного поиска#
В режиме расширенного поиска сверху таблицы SNV Viewer отображаются условия запроса к искомым вариантам. Когда условий в запросе очень много, их удобно свернуть, чтобы они не занимали много места на странице.
В данном разделе можно настроить, будут ли условия запроса в развёрнутом (по умолчанию) или свёрнутом состоянии сразу после применения запроса фильтрации:

SNV & CNV Viewer#
SNV Viewer - это встроенный модуль для просмотра и анализа вариантов. Подробно описан
в соответствующем разделе.
CNV Viewer - это встроенный модуль для просмотра и анализа вариации числа копий. Подробно описан
в соответствующем разделе.
Нижняя панель с деталями выбранного варианта#
Панель детальной информации о варианте содержит основную информацию о выбранном варианте. По умолчанию она находится в развёрнутом состоянии внизу таблицы SNV или CNV Viewer. Если вы не пользуетесь панелью на постоянной основе, то её можно скрыть из SNV и CNV Viewer, выбрав опцию "Свернуть":

Возвращение к настройкам по умолчанию#
Если вы внесли изменения в настройки выбора языка, панели навигации, страницы пациента,
SNV Viewer или CNV Viewer, вы можете вернуться к настройкам по умолчанию,
нажав на кнопку  .
Обратите внимание, что действие приведет к сбросу сразу всех изменений, которые вы совершили
на странице (кроме изменений email, логина и пароля).
.
Обратите внимание, что действие приведет к сбросу сразу всех изменений, которые вы совершили
на странице (кроме изменений email, логина и пароля).