Пользовательские аннотации
Если вы хотите проаннотировать найденные варианты с использованием дополнительных данных, загрузите пользовательскую аннотацию, содержащую эти данные. В том числе можно добавить в систему базу патогенности вариантов, аннотацию из базы COSMIC, базу частот вариантов и аннотацию из базы RUSeq.
Чтобы открыть раздел с пользовательскими аннотациями, перейдите на страницу "Пользовательские аннотации" из блока страниц "Настройки" через панель навигации слева:
Добавление пользовательской аннотации#
Для перехода к форме добавления пользовательской аннотации
нажмите на .
На открывшейся странице можно задать имя и ввести описание загружаемой аннотации, нажав на "-"
(по умолчанию именем аннотации станет название файла):
1. Загрузка файла#
Загрузите файл аннотации в формате VCF, CSV или TSV. Для этого перетащите файл в окно или нажмите на него, чтобы открыть диспетчер файлов:
Укажите версию референсного генома аннотации (hg19 или hg38) для правильного аннотирования вариантов:
Сразу после загрузки вам будет доступен предпросмотр первых семи строк аннотации.
2. Определение типа пользовательской аннотации#
Для загрузки доступны пользовательские аннотации следующих типов:
- База патогенности - аннотация, которая содержит колонку с классами патогенности вариантов. Для колонки поддерживаются следующие числовые или текстовые значения:
- "1" или "Benign" = доброкачественный вариант;
- "2" или "Likely_benign" = вероятно доброкачественный вариант;
- "3" или "Uncertain_significance" = вариант неопределенной значимости;
- "4" или "Likely_pathogenic" = вероятно патогенный вариант;
- "5" или "Pathogenic" = патогенный вариант.
Если в загруженной аннотации есть колонка с подобными значениями, система предложит вам определить эту аннотацию как базу патогенности. Чтобы это сделать, нажмите на переключатель:
Результат аннотации вариантов образца по базе патогенности будет представлен в колонке "Патогенность" в SNV Viewer.
На заметку
В качестве базы патогенности можно определить сколько угодно пользовательских аннотаций - все они будут использоваться при аннотации, если не пересекаются по вариантам. Если вариант есть в нескольких базах патогенности, то он будет аннотироваться той из них, которая была загружена последней.
- База COSMIC - аннотация в формате VCF, которая содержит колонку с идентификаторами вариантов из базы COSMIC (COSV********). Если в загруженной аннотации есть колонка с подобными значениями, система предложит вам определить эту аннотацию как базу COSMIC. Чтобы это сделать, нажмите на переключатель:

Результат аннотации вариантов образца по базе COSMIC будет представлен
в колонке "Внешние ссылки" в SNV Viewer: .
На заметку
Только одна пользовательская аннотация может быть определена как база COSMIC. При определении новой аннотации как базы COSMIC определение предыдущей базы удаляется.
- База частот - аннотация в формате TSV или CSV, которая содержит колонку "Samples_with_SNP" с количеством образцов, в которых был обнаружен вариант, и колонку "Number_of_samples" с общим количеством исследованных образцов (значения обеих колонок - натруальные числа). Если в загруженной аннотации есть колонки с подобными значениями, система предложит вам определить эту аннотацию как базу частот. Чтобы это сделать, нажмите на переключатель:
Результат аннотации вариантов образца по базе частот будет использоваться при расчёте значения достоверности выявления варианта при построении отчёта на основе фенотипа ClinVar.
На заметку
Только одна пользовательская аннотация может быть определена как база частот. При определении новой аннотации как базы частот определение предыдущей базы удаляется.
- База RUSeq - аннотация в формате VCF, которая содержит следующие колонки из базы RUSeq: "AC", "AF", "AN", "BaseQRankSum", "CSQ", "ExcessHet", "FS", "InbreedingCoeff", "MLEAC", "MLEAF", "MQ", "MQRankSum", "QD", "ReadPosRankSum", "SOR", "VQSLOD", "culprit", "MEAN_DP", "MIN_DP", "MEAN_GQ", "AC_diseased", "AF_diseased", "AN_diseased", "n_hom_diseased", "AC_healthy", "AF_healthy", "AN_healthy", "n_hom_healthy", "AC_clust1", "AF_clust1", "AN_clust1", "n_hom_clust1", "AC_clust2", "AF_clust2", "AN_clust2", "n_hom_clust2", "AC_clust3", "AF_clust3", "AN_clust3", "n_hom_clust3".
Если в загруженной аннотации есть все перечисленные выше колонки, система предложит вам определить эту аннотацию как базу RUSeq. Чтобы это сделать, нажмите на переключатель:

В результате аннотации вариантов по базе RUSeq в SNV Viewer можно увидеть:
- ссылку на вариант в RUSeq
в колонке "Внешние ссылки"
или на вкладке "Common" панели детальной информации о варианте:
;
- частоту альтернативного аллеля варианта из RUSeq в отдельной колонке "RUSeq AF" (не добавлена по умолчанию).
На заметку
Только одна пользовательская аннотация может быть определена как база RUSeq. При определении новой аннотации как базы RUSeq определение предыдущей базы удаляется.
- Аннотация с любой другой информацией о варианте - в этом случае тип аннотации определять не нужно. Результат аннотации вариантов образца по всем пользовательским аннотациям представлены на вкладке "Custom annotation sources" панели детальной информации о варианте:

3. Сопоставление колонок#
Если вы загрузили файл аннотации в формате VCF, то колонки, необходимые для аннотации, (CHROM, POS, REF, ALT) определятся автоматически.
Если вы загрузили файл аннотации в формате CSV или TSV, то нужно определить колонки, необходимые для аннотации:

- CHROMOSOME - колонка с названиями хромосом, в которых расположены варианты.
- POSITION (1-BASED) - колонка со стартовыми позициями вариантов.
- REFERENCE - колонка с референсными аллелями вариантов.
- ALTERNATIVE - колонка с альтернативными аллелями вариантов.
Для каждой колонки выберите соответствующую колонку из загруженной вами аннотации. Например, в данном случае колонке "CHROMOSOME" соответствует колонка "chr" из аннотации:
Если вы определили аннотацию как базу патогенности, то для любого формата файла для аннотации необходимо определить:
- колонку с названием гена:
- колонку с классами патогенности вариантов (колонка будет определена автоматически - убедитесь, что она была выбрана правильно):
Если вы определили аннотацию как базу COSMIC, то для аннотации автоматически определится колонка с идентификатором варианта из базы COSMIC (COSV********):
Если вы определили аннотацию как базу частот, то для аннотации автоматически определятся следующие колонки:
- колонка с количеством образцов, в которых был обнаружен вариант:

- колонка с общим количеством исследованных образцов:
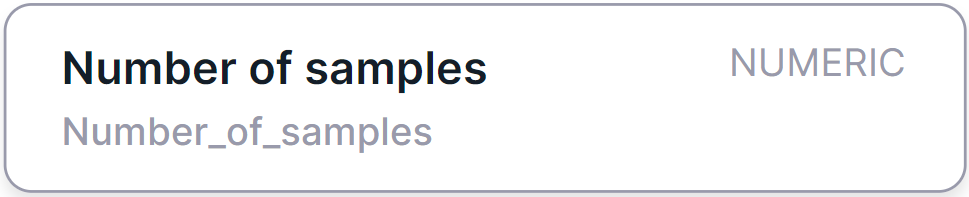
Если вы определили аннотацию как базу RUSeq, то для аннотации автоматически определится колонка с частотами альтернативных аллелей вариантов из базы RUSeq ("AF"):

4. Добавление колонки аннотации#
- Недоступно, если вы определили аннотацию как базу патогенности.
- Доступно, но не обязательно к заполнению, если вы определили аннотацию как базу COSMIC.
В загруженной аннотации выберите колонки с данными, которыми вы хотите аннотировать варианты:
- Нажмите на
.
- В открывшемся окне задайте имя колонке:
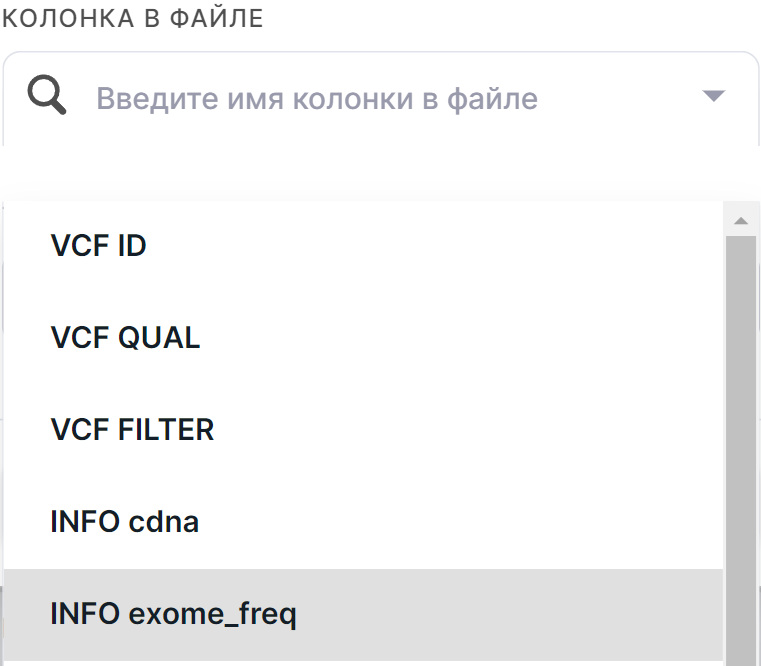
- Укажите выбранную колонку в файле. Скажем, у нас есть данные с частотами вариантов в экзоме и мы хотим аннотировать найденные варианты с использованием этой информации. Для этого выберем соответствующую колонку в файле:
- Определите тип колонки: текстовый или числовой:
- Нажмите
.
Колонка аннотации будет добавлена:
Чтобы отредактировать колонку, наведите на
неё курсор и нажмите на .
Чтобы удалить колонку, наведите на на
неё курсор и нажмите на .
Таким же образом можно добавить несколько колонок аннотации.
5. Запуск процесса обработки#
Чтобы процесс обработки аннотации начался, нажмите
на .
Внимание!
Если вы пропустили один из обязательных шагов создания аннотации
(загрузка файла, определение сборки референса файла,
сопоставление колонок, необходимых для идентификации варианта, или добавление хотя бы одной колонки
для аннотаций, отличных от баз патогенности, COSMIC, частот и RUSeq),
то
кнопка будет неактивна.
По файлу, не прошедшему обработку, аннотирование вариантов происходить не будет.
Пока обработка аннотации находится в процессе, статус будет "IN_PROGRESS":
Успешно обработанная аннотация будет иметь статус "SUCCESS":
или "PROBLEMS", если во время обработки возникли некритичные проблемы (подробнее о проблеме можно узнать ниже в разделе "Warnings"):
Обработка аннотации, подготовленной неправильно, завершится неудачно со статусом "FAILED" (об источнике ошибки можно узнать ниже в разделе "Errors"):
В такой ситуации скорректируйте один из шагов подготовки аннотации (выберите верную сборку референса, укажите правильный тип колонки аннотации) или выберите другой файл аннотации.
Чтобы вернуться к списку всех пользовательских аннотаций,
нажмите .
Значения колонок, выбранных для аннотации вариантов, будут находиться на странице SNV Viewer и в файле с вариантами, который можно скачать с главной страницы образца.
Редактирование пользовательской аннотации#
- Если вы хотите отредактировать уже подготовленную аннотацию (добавить, удалить или отредактировать колонки аннотации,
поменять сборку рефересного генома или поменять выбор обязательных колонок), вы можете это сделать на её странице.
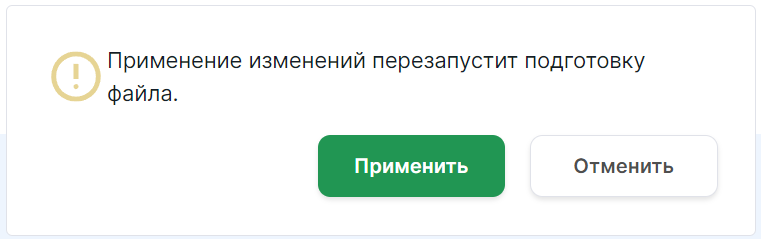
Для этого внесите необходимые изменения и нажмите
. Чтобы отменить внесенные изменения, нажмите
или просто покиньте страницу - изменения не сохранятся.
- Если вы хотите прекратить определение уже подготовленной аннотации как базы патогенности, базы COSMIC, базы частот или базы RUSeq, выключите соответствующий переключатель на странице аннотации. Сохранять изменения в таком случае не нужно.
Совет
Если вы хотите, чтобы только что созданная или отредактированная аннотация аннотировала варианты образца, для которого уже завершена стадия "Аннотация SNVs/Indels", перезапустите эту стадию или процессинг образца.
Удаление пользовательской аннотации#
Чтобы удалить ненужную пользовательскую аннотацию, выполните следующие действия:
Наведите курсор на строку аннотации на странице "Пользовательские аннотации";
Нажмите на
;
В появившемся окне подтвердите свои действия, нажав на
.
На заметку
Данные удаленной аннотации сохранятся с пометкой "deleted" для вариантов, которые были аннотированы с её помощью до её удаления:
