SNV Viewer
Знакомство с SNV Viewer#
SNV Viewer - это встроенный модуль для просмотра и анализа однонуклеотидных вариантов (single-nucleotide variants; SNVs) и коротких инсерций/делеций (Indels), выявленных в образце. Доступен на главной странице образца (вкладка Главное).
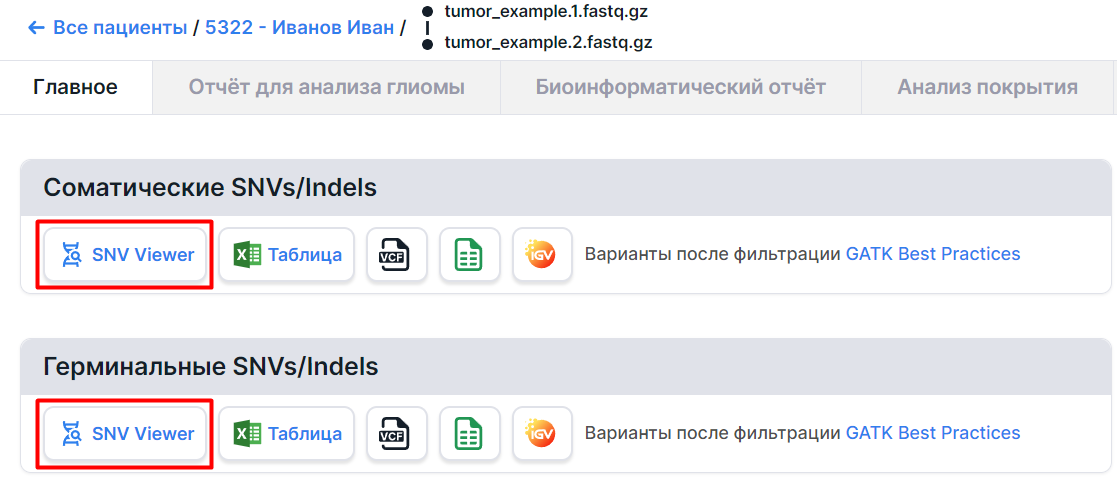
На странице образца опухолевой ткани вы найдете два SNV Viewer: с соматическими SNVs/Indels, выявленными у этого образца, и с герминальными SNVs/Indels, которые были выявлены в контрольном образце в случае пары образцов опухоль/контроль или в самом опухолевом образце, если он анализировался без контрольного образца. В SNV Viewer с соматическими мутациями можно также увидеть и SNVs/Indels из SNV Viewer с герминальными мутациями, если поменять значение фильтра по типу мутации с Somatic на Germline или сбросить фильтрацию по нему.
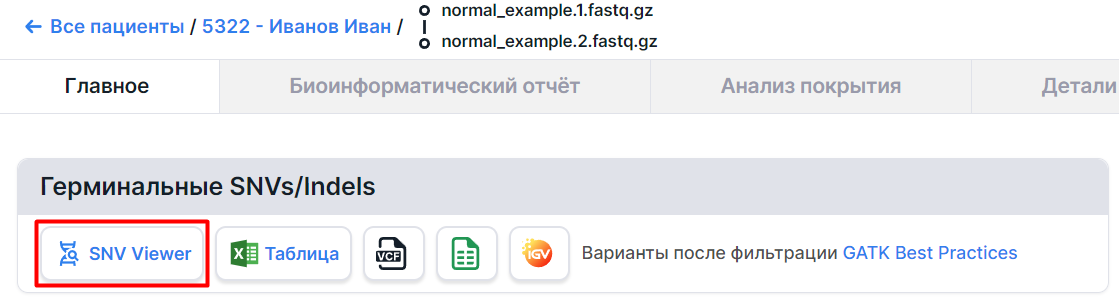
На странице образца неопухолевой ткани представлен SNV Viewer только с герминальными SNVs/Indels, выявленными у этого образца:
Основная страница#
Нажав на кнопку  ,
вы попадете на основную страницу модуля:
,
вы попадете на основную страницу модуля:
На заметку
Вид некоторых функций SNV Viewer может быть изменён на странице "Настройки пользователя".
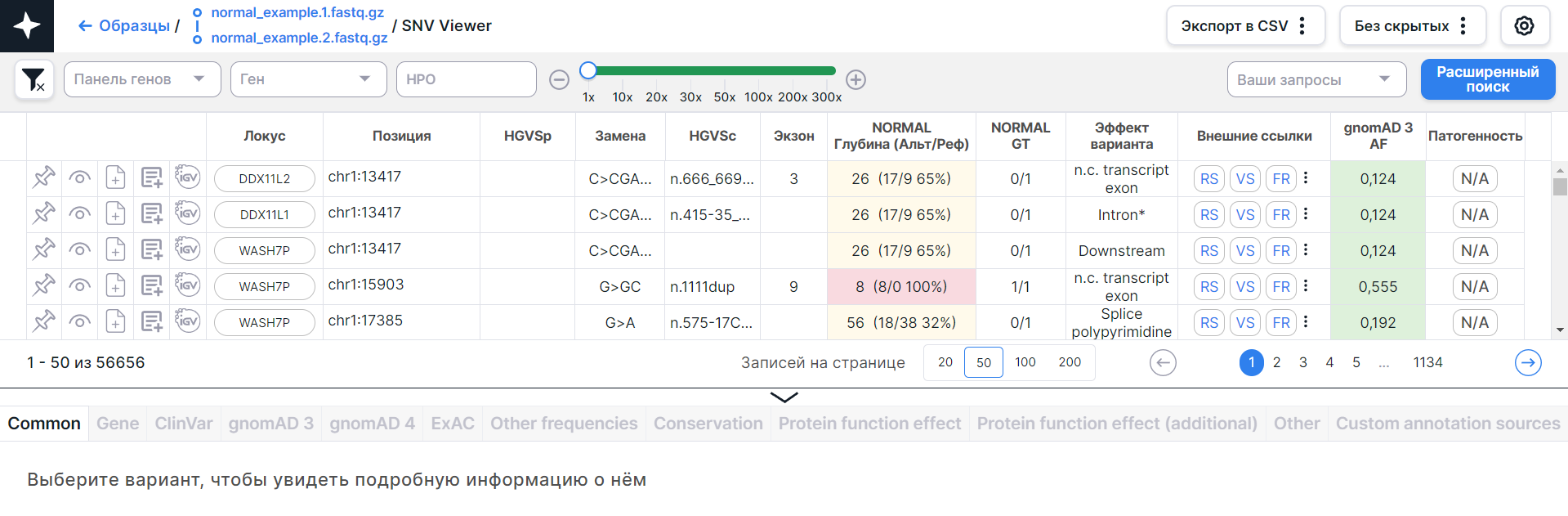
Основным объектом SNV Viewer является таблица с вариантами - каждый вариант на новой строчке:
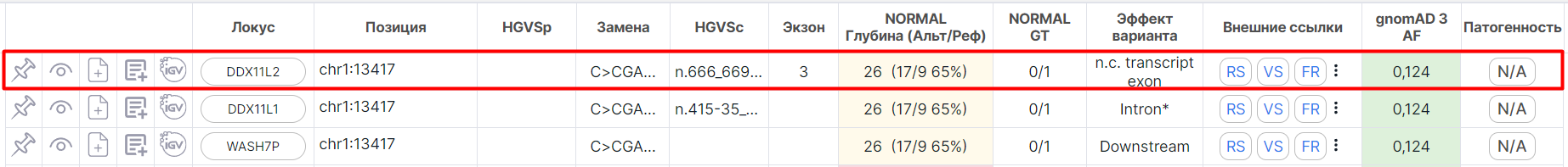
Под таблицей, над панелью детальной информации о варианте находятся:
- Информация о количестве вариантов в SNV Viewer: порядок вариантов, отображённых на странице, (например, если максимальное количество вариантов, показываемых на странице, - 50, то порядок будет таким: 1-50, 51-100 и т.д.) и суммарное количество вариантов (закреплённые варианты в подсчёте не учитываются). Количество вариантов изменяется при поиске вариантов, при котором отображается количество вариантов, подходящих под заданные условия.
- Компонент с выбором количества вариантов, отображаемых на
странице (без учёта закреплённых
вариантов):. По умолчанию отображается 50 вариантов на странице. Чтобы изменить количество отображаемых на странице вариантов на 20, 100 или 200, нажмите на соответствующую опцию. Ваш выбор будет запомнен и будет действовать для SNV Viewer и CNV Viewer всех образцов. Обратите внимание, что чем больше вариантов отображается на странице, тем дольше она будет загружаться.
- Компонент пагинации:
, который позволяет перемещаться между страницами с вариантами. Номер открытой страницы выделен синим кружком. Если нажать на этот номер, то страница перезагрузится. Чтобы переместиться на страницу с конкретным номером, либо нажмите на этот номер, либо нажмите на
, введите нужный номер и нажмите клавишу ввода или кликните вне области заполнения значения. Чтобы переместиться на предыдущую по номеру страницу, нажмите на
, а чтобы открыть следующую - нажмите на
.
Информация о варианте#
Помимо представленной в таблице информации (см. Описание колонок таблицы), для каждого варианта есть несколько встроенных модулей с дополнительной информацией:
- Панель детальной информации о варианте. Чтобы увидеть панель, нажмите на строку варианта:

Подробнее с панелью и её вкладками можно ознакомиться в соответствующем разделе.
- Страница детальной информации о варианте. Для открытия страницы
нажмите на
в строке варианта. На странице можно добавить интерпретацию для варианта, определить его патогенность и найти дополнительные сведения о варианте. Страница может включать следующие разделы:
- Подробную аннотацию варианта.
- Информацию об образце и пациенте.
- Информацию о встречаемости варианта в других образцах.
- ACMG классификацию - включает критерии
для интерпретации вариантов, разработанные ACMG.
Подробнее страница "ACMG классификация" описана
в соответствующем разделе.
На страницу можно
попасть напрямую, нажав на кнопку патогенности (
, если класс патогенности этого варианта ранее не определялся) в строке варианта (колонка "Патогенность") и в появившемся диалоге нажав на "ACMG Классификация":

- Научные публикации, ассоциированные с вариантом.
- Клинические рекомендации. Страница доступна,
если вариант является онкозначимым. На страницу можно попасть
напрямую, нажав на
в строке варианта (колонка "Онкозначимость" - может быть добавлена в режиме редактирования колонок).
- Отчёт о варианте.
- Модуль визуализации варианта в геноме (IGV). Чтобы открыть встроенный модуль,
нажмите на
в строке варианта.
Закрепление вариантов#
Нужные варианты можно закрепить. На закрепленные варианты не влияют фильтрация и сортировка всей таблицы вариантов, они всегда остаются вверху таблицы. На видимость закрепленных вариантов в таблице может повлиять только фильтр по типу мутации (например, закрепленные герминальные варианты не будут отображаться в таблице, отфильтрованной по соматическому типу мутации). Между собой закрепленные варианты отсортированы по стартовой позиции варианта (колонка "Позиция").
Чтобы закрепить вариант,
нажмите на в строке варианта.
Закрепленный вариант окажется вверху таблицы (строка подсвечена зелёным):

Чтобы открепить вариант, нажмите на ту же кнопку
в строке варианта.
Закрепить или открепить вариант можно также с помощью горячих клавиш клавиатуры. Для этого выберите строку варианта (выбранная строка подсвечена синим) и нажмите P (латинскую) на клавиатуре.
На заметку
Закрепление варианта сохраняется при выходе со страницы SNV Viewer.
Скрытие вариантов#
Варианты можно скрыть из таблицы SNV Viewer. Чтобы это сделать,
нажмите на в строке варианта.
Скрытые варианты будут доступны только при применении особого условия фильтрации.
Фильтрация по видимости вариантов: в правом верхнем углу страницы SNV Viewer (независимо от режима
поиска вариантов) находится кнопка фильтра по видимости
вариантов:  (состояние по умолчанию):
(состояние по умолчанию):
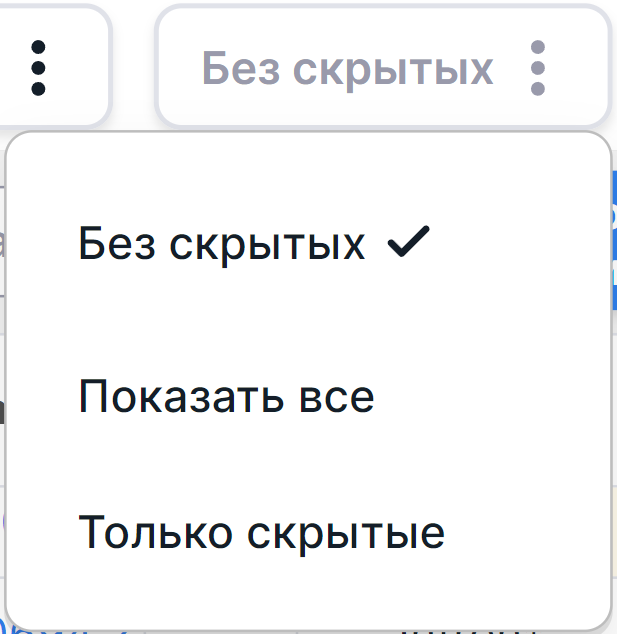
- По умолчанию в таблице SNV Viewer показываются только видимые (не скрытые) варианты (опция фильтра "Без скрытых").
- Чтобы увидеть в таблице все варианты, выявленные и аннотированные в образце,
нажмите на кнопку фильтра и выберите опцию "Показать все". Тогда в таблице отобразятся все варианты,
включая скрытые. У видимых вариантов в строке есть кнопка для
скрытия:
, а у скрытых - кнопка для показа:
.
- Чтобы увидеть только скрытые варианты, нажмите на кнопку фильтра и выберите опцию "Только скрытые".
На заметку
Закрепленные варианты - всегда видимые. При этом они отображаются в таблице при любом условии фильтрации по видимости варианта.
Чтобы сделать скрытый вариант обратно видимым,
нажмите на в его строке.
Скрыть или показать вариант можно также с помощью горячих клавиш клавиатуры. Для этого выберите строку варианта (выбранная строка подсвечена синим) и нажмите H (латинскую) на клавиатуре.
На заметку
При закреплении скрытого варианта он не только становится закрепленным в таблице SNV Viewer, но также и видимым.
Включение вариантов в отчёт#
Включенные в отчёт варианты будут входить в блоки отчётов с типом
"Выбранные пользователем SNVs/Indels для добавления в отчёт".
Для включения варианта в отчёт нажмите на в строке варианта. Для исключения варианта из отчёта нажмите на ту же кнопку
в строке варианта.
Также включенные в отчёт варианты можно удалить из отчёта на странице самого отчёта.
Включить вариант в отчёт или исключить его из отчёта можно также с помощью горячих клавиш клавиатуры. Для этого выберите строку варианта (выбранная строка подсвечена синим) и нажмите R на клавиатуре.
На заметку
При определении варианта как патогенного или вероятно патогенного он автоматически добавляется в отчёт.
Метка значимости варианта#
Вариант можно интерпретировать по его значимости (клинической или для вашего исследования и т.п.)
Для этого добавьте колонку "Метка"
в режиме редактирования колонок (не входит в число
колонок по умолчанию). Колонка
содержит кнопку метки
("Значимость не определена", если значение метки не было определено).
Чтобы интерпретировать вариант по значимости, нажмите
на кнопку метки и в появившемся списке
выберите нужное значение:

В дальнейшем значимость можно будет переопределить тем же способом или очистить, нажав на кнопку метки и выбрав опцию "Очистить":

В результате интерпретации варианта по значимости кнопка метки поменяет цвет:
- Высокая значимость:
- Средняя значимость:
- Низкая значимость:
- Ошибка секвенирования:
Варианты можно сортировать по значимости: от высокой значимости к ошибке секвенирования, нажав на название колонки "Метка" один раз, и наоборот, нажав на название два раза. Кроме того, варианты можно фильтровать по значимости:
- фильтр "Label" в режиме расширенного поиска;
- дополнительный фильтр "Метка" в режиме базового поиска (можно добавить на странице "Профиль").
Поиск и сортировка вариантов#
Поиск вариантов в SNV Viewer описан в соответствующих разделах Базовый поиск вариантов и Расширенный поиск вариантов.
По умолчанию таблица отсортирована по стартовой позиции варианта (колонка "Позиция"). Таблицу можно сортировать по возрастанию или убыванию значений следующих колонок:
- колонки по умолчанию: "Локус", "Тип мутации", "Позиция", "Экзон", "Глубина (Альт/Реф)", "Эффект варианта", "gnomAD 3 AF", "Патогенность";
- все колонки, не добавленные по умолчанию.
Для сортировки таблицы по значениям колонки нажмите на её название (нажмите один раз, чтобы отсортировать
по возрастанию, или два раза, чтобы отсортировать по убыванию). После этого у названия колонки появится
стрелка (при сортировке по возрастанию) или
(при сортировке по убыванию).
Таблицу можно отсортировать по 1-3 колонкам одновременно, чтобы сгруппировать данные с одинаковыми значениями в одной колонке, а затем отсортировать эти группы с одинаковыми значениями по другой колонке. Порядок сортировки будет зависеть от порядка нажатия на названия соответствующих колонок. Стрелочка сортировки будет у той колонки, по которой сортировали последней.
На заметку
Сортировка и фильтрация таблицы сохраняются при выходе со страницы SNV Viewer.
Для сброса сортировки возпользуйтесь кнопкой очистки всех
фильтров: . Обратите внимание, что
при этом сбросится и фильтрация вариантов.
Основным принципом сортировки является сортировка по алфавиту для колонок с текстовыми значениями (например, колонка "Локус") или сортировка от минимального к максимальному и наоборот для колонок с числовыми значениями (например, колонка "gnomAD 3 AF"). Однако для некоторых колонок сортировка особенная:
- Колонка "Эффект варианта": сортировка от наиболее значимого эффекта к наименее значимому: Transcript ablation, Splice acceptor, Splice donor, Stop gained, Frameshift, Stop lost, Start lost, Transcript amplification, Inframe insertion, Inframe deletion, Missense, Protein altering, Splice region, Incomplete terminal codon, Start retained, Stop retained, Synonymous, Coding sequence, Зрелая миРНК, 5'-НТО, 3'-НТО, Non coding transcript exon, Intron, NMD transcript, Non coding transcript, Upstream, Downstream, TFBS ablation, TFBS amplification, TF binding site, Regulatory region ablation, Regulatory region amplification, Feature elongation, Regulatory region variant, Feature truncation, Intergenic variant, Splice donor 5th base variant, Splice donor region variant, Splice polypyrimidine tract variant, Unknown. Составные значения (включающие несколько эффектов) сортируются по наименее значимому значению из списка.
- Колонка "Патогенность": сортировка от патогенного к доброкачественному варианту: Патогенный, Вероятно патогенный, Конфликт: противоречивые интерпретации патогенности, Неопределенная значимость, Вероятно доброкачественный, Доброкачественный.
- Колонка "Значение эффекта" (не добавлена по умолчанию, значения описаны здесь): сортировка от высокого до незначительного эффекта: HIGH, MODERATE, LOW, MODIFIER.
- Колонка "Genome position" (не добавлена по умолчанию): сортировка по значимости расположения варианта в геноме: Exonic (в экзоне), Intronic (в интроне), Intergenic (в межгенной области).
- Колонка "Метка" (не добавлена по умолчанию, описана здесь):
сортировка от значимых вариантов к незначимым:
(высокая значимость),
(средняя значимость),
(низкая значимость),
(ошибка секвнирования).
- Колонка "Фильтры детекции" (не добавлена по умолчанию, описана здесь): сортировка от не прошедших фильтрацию вариантов до удовлетворяющих всех фильтрам: FAIL, N/A, PASS.
- Колонка "ClinVar" (не добавлена по умолчанию, описана здесь): сортировка от безусловно значимых вариантов к незначимым:
- Патогенный;
- Патогенный с низкой пенетрантностью;
- Вероятно патогенный;
- Вероятно патогенный с низкой пенетрантностью;
- Составные значения, включающие "Патогенный" или "Патогенный с низкой пенетрантностью";
- Составные значения, включающие "Вероятно патогенный" или "Вероятно патогенный с низкой пенетрантностью";
- Неопределенная значимость;
- Конфликт (Противоречивые интерпретации патогенности или Противоречивые данные от отправителей);
- Аллель установленного риска;
- Аллель вероятного риска;
- Аллель неопределенного риска;
- Фактор риска;
- Влияет;
- Лекарственная реакция;
- Ассоциация;
- Даёт чувствительность;
- Защитный;
- Вероятно доброкачественный;
- Ассоциация не найдена;
- Доброкачественный;
- Другое;
- Не предоставлена.
Составные значения (кроме тех, которые включают значения "Патогенный", "Патогенный с низкой пенетрантностью", "Вероятно патогенный" или "Вероятно патогенный с низкой пенетрантностью") сортируются по наиболее значимому значению из списка.
- Колонка "ENIGMA" (не добавлена по умолчанию, описана здесь): сортировка от значимых вариантов к незначимым: Патогенный, Вероятно патогенный, Вероятно доброкачественный, Доброкачественный, Не предоставлена. Составные значения сортируются по наиболее значимому значению из списка.
Горячие клавиши в SNV Viewer#
В SNV Viewer можно взаимодействовать с вариантами с помощью горячих клавиш клавиатуры. Чтобы воспользоваться горячими клавишами, нажмите на строку одного из вариантов в таблице или нажмите на одну из клавиш S, W, ⬇️ или ⬆️, чтобы выбрать первый вариант в таблице.
Доступные горячие клавиши
| Нажмите эту клавишу | Для этого |
| S или ⬇️ | Перемещение к строке следующего варианта (или первого варианта в таблице, если ни один вариант не был выбран) |
| W или ⬆️ | Перемещение к строке предыдущего варианта (или первого варианта в таблице, если ни один вариант не был выбран) |
| P | Закрепление/открепление варианта |
| H | Скрытие/показ варианта |
| R | Включение варианта в отчёт или исключение варианта из отчёта |
Экспорт вариантов#
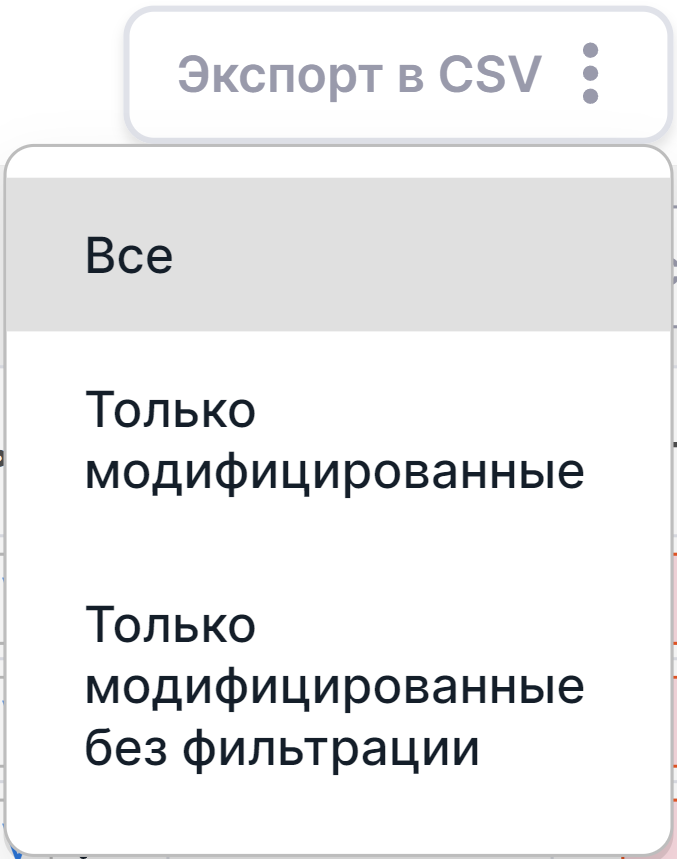
SNV Viewer позволяет экспортировать варианты в формате CSV:
- Экспорт всех вариантов: нажмите на
 и выберите опцию "Все":
и выберите опцию "Все":
- Экспорт вариантов, подходящих под определенное условие: отфильтруйте нужные варианты с помощью
Базового или Расширенного
поиска, а затем нажмите на
 и выберите
опцию "Все":
и выберите
опцию "Все":

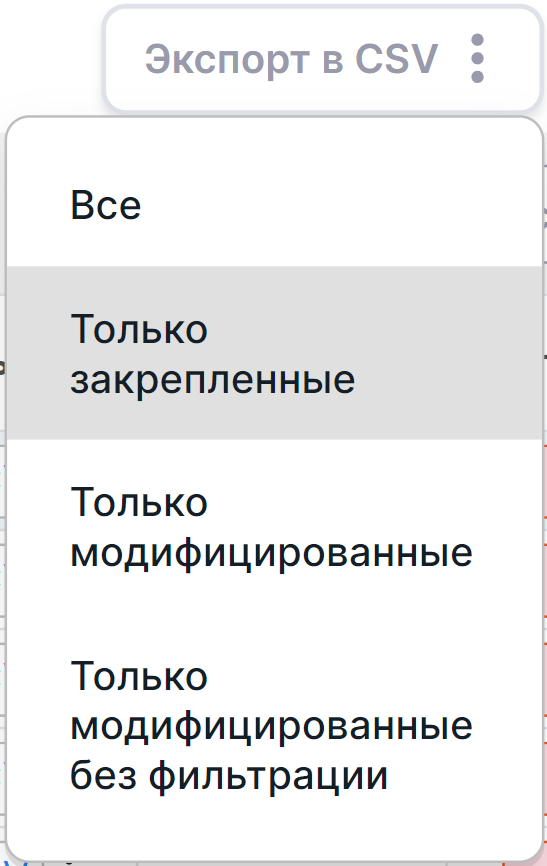
- Экспорт только закрепленных вариантов: закрепите
нужные варианты, а затем нажмите на
 и выберите опцию "Только закрепленные":
и выберите опцию "Только закрепленные":
На заметку
Если ни один вариант не был закреплен в SNV Viewer, то опции "Только закрепленные" не будет в списке опций экспорта.
- Экспорт только модифицированных вариантов: нажмите
на
 и выберите опцию
"Только модифицированные":
и выберите опцию
"Только модифицированные":

Модифицированные варианты - это варианты, для которых было сделано любое из следующих действий:
- вариант закреплен в таблице SNV Viewer;
- вариант включен в отчёт;
- для варианта добавлена интерпретация;
- для варианта определена патогенность;
- вариант интерпретирован по значимости.
На заметку
Если таблица с вариантами отфильтрована по какому-либо условию, то в таблицу экспортируемых модифицированных вариантов войдут только те модифицированные варианты, которые подходят под это условие. Исключение составляют закрепленные варианты, на видимость в таблице которых может повлиять только фильтр по типу мутации.
Чтобы экспортировать все модифицированные варианты без учёта фильтрации, нажмите
на  и выберите опцию
"Только модифицированные без фильтрации":
и выберите опцию
"Только модифицированные без фильтрации":

Описание колонок таблицы#
По умолчанию таблица включает следующие колонки (набор и порядок колонок можно изменить - то, как это сделать, описано здесь):
- Локус - название гена, в котором располагается вариант. Если ген неизвестен, локус обозначается
следующим образом:
. Название гена можно скопировать в буфер обмена. Для этого наведите курсор на значение колонки и нажмите на
.
- Тип мутации (соматическая или герминальная).
На заметку
Колонка "Тип мутации" отсутствует в SNV Viewer с герминальными мутациями и в SNV Viewer с соматическими мутациями, если включен фильтр "Origin является SOMATIC".
- Позиция - координаты варианта в геноме (хромосома + начальная позиция).
Позицию варианта можно скопировать в буфер обмена. Для этого наведите курсор на значение
колонки и нажмите на
.
- HGVSp - аминокислотная замена в номенклатуре HGVS (префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены). Аминокислоты записаны в однобуквенной кодировке. При наведении курсора на значение колонки "HGVSp" для варианта можно увидеть транскрипт, для которого была предсказана замена, и HGVSp с трёхбуквенным обозначением аминокислот:
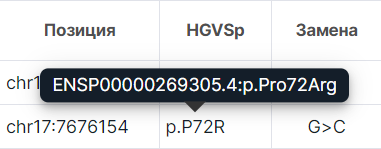
На заметку
Если вам удобнее пользоваться трёхбуквенной кодировкой аминокислот, добавьте колонку "HGVSp (long)" в окне выбора колонок и скройте колонку "HGVSp (short)".
- Замена - нуклеотидная замена (референсный аллель > альтернативный аллель).
- HGVSc - нуклеотидная замена в номенклатуре HGVS (геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель). Начинается с префикса “c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности). При наведении курсора на значение колонки "HGVSс" для варианта можно увидеть, для какого транскрипта предсказана замена (приведены идентификаторы транскриптов из Ensembl (ENSTxxxxxxxxxxx) и RefSeq (NM_)):

- Экзон - номер экзона в транскрипте, который вариант затронул.
- Глубина (Альт/Реф) - глубина секвенирования; общее количество прочтений последовательности, перекрывающих позицию варианта, для этого образца. Альт - количество раз, когда считывался альтернативный нуклеотид, Реф - количество раз, когда считывался референсный нуклеотид. Также приведен процент доли количества прочтений альтернативного аллеля среди всех прочтений (Альт/Глубина).
Ячейки колонки раскрашены в зависимости от значения глубины:
- зелёным, если глубина > 60;
- жёлтым, если 20 ≤ глубина ≤ 60;
- красным, если глубина < 20.
- GT (генотип) - значения аллелей (0 - референсный аллель, 1 - первый альтернативный аллель, 2 - второй альтернативный аллель и т.д.), разделенные ”/” (для нефазированного генотипа) или “|” (для фазированного генотипа).
- Эффект варианта - последствия варианта для генов.
Эффекты варианта в аннотации#
| Эффект варианта | Определение | Значение эффекта* |
| 3'-НТО | Вариант попадает в 3′-нетранслируемую область | MODIFIER |
| 5'-НТО | Вариант попадает в 5′-нетранслируемую область | MODIFIER |
| Зрелая миРНК | Вариант попадает в транскрипт, расположенный в последовательности зрелой миРНК | MODIFIER |
| Coding sequence | Вариант располагается в кодирующей последовательности | MODIFIER |
| Downstream | Вариант расположен после гена | MODIFIER |
| Feature elongation | Вариант вызывает удлинение геномной области | MODIFIER |
| Feature truncation | Вариант вызывает уменьшение геномной области | MODIFIER |
| Frameshift | Инсерция или делеция, вызывающая сдвиг рамки считывания | HIGH |
| Incomplete terminal codon | Вариант, в котором изменено по крайней мере одно основание стоп-кодона не полностью аннотированного транскрипта | LOW |
| Inframe deletion | Делеция одного или нескольких кодонов | MODERATE |
| Inframe insertion | Вставка одного или нескольких кодонов | MODERATE |
| Intergenic variant | Вариант попадает в межгенную область | MODIFIER |
| Intron | Вариант расположен в интроне | MODIFIER |
| Missense | Вариант приводит к появлению кодона, который кодирует другую аминокислоту | MODERATE |
| NMD transcript | Вариант попадает в транскрипт, восприимчивый к нонсенс-опосредованному распаду | MODIFIER |
| Non coding transcript | Вариант расположен в некодирующем транскрипте | MODIFIER |
| Non coding transcript exon | Вариант изменяет последовательность некодирующего экзона в некодирующем транскрипте | MODIFIER |
| Protein altering | Вариант, для которого предсказано, что он изменяет кодируемый белок | MODIFIER |
| Regulatory region ablation | Делеция регуляторной области | HIGH |
| Regulatory region amplification | Амплификация регуляторной области | HIGH |
| Regulatory region variant | Вариант попадает в регуляторную область | MODIFIER |
| Splice acceptor | Вариант попадает в акцепторный сайт сплайсинга | HIGH |
| Splice donor | Вариант попадает в донорный сайт сплайсинга | HIGH |
| Splice donor 5th base variant | Вариант приводит к изменению ориентации транскрипта в 5-й паре оснований от начала интрона | LOW |
| Splice donor region variant | Вариант попадает в область между 3-й и 6-й парами оснований после сайта сплайсинга (5'-конец интрона) | LOW |
| Splice polypyrimidine tract variant | Вариант находится в полипиримидиновом тракте на 3'-конце интрона между 17-й и 3-й парами оснований от конца (от -3 акцептора до -17 акцептора) | LOW |
| Splice region | Вариант, в котором произошло изменение в области сайта сплайсинга либо в пределах 1-3 оснований экзона, либо 3-8 оснований интрона | LOW |
| Start lost | Вариант вызывает мутацию старт-кодона в non-start кодон | HIGH |
| Start retained | Вариант вызывает мутацию старт-кодона в другой старт-кодон | LOW |
| Stop gained | Вариант приводит к появлению стоп-кодона | HIGH |
| Stop lost | Вариант вызывает мутацию стоп-кодона в non-stop кодон | HIGH |
| Stop retained | Вариант вызывает мутацию стоп-кодона в другой стоп-кодон | LOW |
| Synonymous | Вариант приводит к появлению кодона, который кодирует ту же аминокислоту | LOW |
| TF binding site | Вариант попадает в сайт связывания транскрипционного фактора (ССТФ) | MODIFIER |
| TFBS ablation | Делеция ССТФ | HIGH |
| TFBS amplification | Амплификация ССТФ | HIGH |
| Transcript ablation | Делеция транскрипта | HIGH |
| Transcript amplification | Амплификация области, содержащей транскрипт | HIGH |
| Upstream | Вариант расположен перед геном | MODIFIER |
Ячейки колонки "Эффект варианта" раскрашены в зависимости от значения эффекта*:
- красным, если значение эффекта HIGH;
- жёлтым, если значение эффекта MODERATE;
- без цвета, если значение эффекта LOW или MODIFIER.
Значения, включающие несколько эффектов варианта, обозначены звездочкой. При наведении курсора на ячейку можно увидеть полный список эффектов:
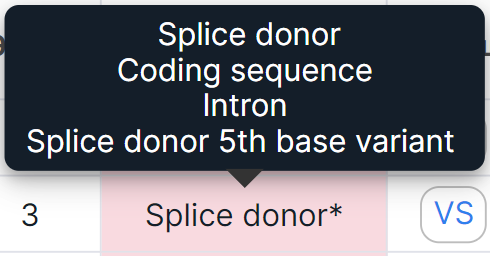
*Значение эффекта - предсказанное воздействие варианта на белок. Колонка не добавлена в таблицу по умолчанию, её можно добавить в окне выбора колонок.
Значения эффектов варианта в аннотации#
| Значение эффекта | Определение |
| HIGH | Вариант разрушающе влияет на белок, предположительно вызывая укорочение белка, потерю его функции или нонсенс-опосредованный распад мРНК |
| MODERATE | Вариант изменяет эффективность белка |
| LOW | Вариант безвреден для белка или не меняет его поведение |
| MODIFIER | Предсказание влияния варианта на белок затруднено или нет доказательства влияния варианта (обычно для вариантов, попадающих в некодирующие последовательности) |
Ячейки колонки "Значение эффекта" раскрашены соответственно:
- красным, если значение эффекта HIGH;
- жёлтым, если значение эффекта MODERATE;
- без цвета, если значение эффекта LOW или MODIFIER.
- Внешние ссылки - ссылки на вариант в базах
данных dbSNP:
, VarSome:
, ClinVar:
, COSMIC:
(если база была загружена в виде пользовательской аннотации), Franklin:
, UCSC:
, gnomAD 3:
,
RUSeq:(если база была загружена в виде пользовательской аннотации). В ячейке может быть до трёх кнопок-ссылок максимум. Если вариант найден более, чем в трёх базах, то в ячейке появляются три точки
, нажав на которые можно увидеть все остальные ссылки:

- gnomAD 3 AF - частота аллеля по данным базы gnomAD (версия 3).
Ячейки колонки раскрашены в зависимости от значения частоты:
- зелёным, если AF ≥ 0.015;
- жёлтым, если 0.01 ≤ AF < 0.015;
- красным, если AF < 0.01.
- Патогенность - патогенность варианта, которая может быть определена
либо самостоятельно,
либо по критериям ACMG,
либо по базе патогенности.
Если вы не загрузили базу патогенности
или не определили патогенность варианта, то
значение колонки будет
(Нет интерпретации).
Описание дополнительных колонок таблицы#
В дополнение к описанным выше колонкам в таблицу SNV Viewer можно добавить другие колонки аннотации, как описано здесь. Ниже описаны колонки с "особенностями":
- Колонка "Метка", в которой можно определить значимость варианта, описана выше.
- Колонка "ФГ" содержит информацию о фазовой группе варианта.
В форме добавления колонок она
называется "Статус фазовой группы" (раздел "Common").
Сама фазовая группа (объединение гомозиготных вариантов, расположенных в пределах определенного геномного
интервала) обозначена
. Варианты, являющиеся частью фазовой группы с совпадающей аминокислотной заменой, обозначены
, а с несовпадающей -
.
- Колонка "Онкозначимость" содержит
кнопку
, если вариант является онкозначимым, т.е. ассоциирован с развитием онкологических заболеваний. Кнопка открывает вкладку "Клинические рекомендации" страницы детальной информации о варианте.
- Колонка "Фильтры детекции" содержит информацию о качестве варианта:
1. PASS - сайт содержит минимум один аллель, который удовлетворяет всем фильтрам. Ячейка раскрашена зелёным.
2. N/A - значение колонки неизвестно (точка в VCF-файле). Ячейка раскрашена жёлтым.
3. FAIL - вариант не удовлетворяет одному или нескольким фильтрам GATK. Наведя курсор мыши на ячейку, можно увидеть эти фильтры. Ячейка раскрашена красным. Возможные фильтры перечислены ниже.
Нажмите, чтобы посмотреть определения фильтров детекции
| Значение фильтра детекции | Тип мутации | Инструмент GATK | Описание фильтра |
| alignment | Соматическая | FilterMutectCalls | Фильтр артефактов выравнивания |
| base_qual | Соматическая | FilterMutectCalls | Среднее качество оснований прочтений, поддерживающих альтернативный аллель |
| clustered_events | Соматическая | FilterMutectCalls | Кластерные события, наблюдаемые в опухоли, - соматические мутации, которые совместно встречаются в одном и том же интервале сборки |
| CNN_2D | Герминальная | CNNScoreVariants | Фильтрация на основе оценок предварительно обученной сверточной нейронной сети (convolutional neural network; CNN) 2D |
| contamination | Соматическая | FilterMutectCalls | Фильтр контаминации данных секвенирования |
| duplicate | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля чрезмерно представлены очевидными дубликатами |
| FAIL | Соматическая | FilterMutectCalls | Ошибка сайта, если все аллели не проходят по разным причинам |
| fragment | Соматическая | FilterMutectCalls | Разница медианных длин фрагментов прочтений, поддерживающих альтернативный и референсный аллели |
| FS | Герминальная | FisherStrand | Вероятность смещения цепей на сайте, оцененная с помощью точного критерия Фишера. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. FisherStrand определяет, существует ли смещение цепи между прямой и обратной цепями для референсного или альтернативного аллеля (то есть встречается ли альтернативный аллель чаще или реже на прямой или обратной цепи, чем референсный аллель). FisherStrand хуже учитывает большие объемы данных в ситуациях с высоким покрытием по сравнению с StrandOddsRatio. Кроме того, FisherStrand имеет тенденцию "штрафовать" варианты, которые встречаются на концах экзонов. Варианты на концах экзонов, как правило, покрываются прочтениями только в одном направлении цепи, и FisherStrand дает таким вариантам плохую оценку, тогда как StrandOddsRatio учитывает соотношение прочтений, охватывающих оба аллеля. |
| germline | Соматическая | FilterMutectCalls | Доказательства указывают на то, что этот сайт является герминальным, а не соматическим |
| haplotype | Соматическая | FilterMutectCalls | Вариант расположен рядом с отфильтрованным вариантом того же гаплотипа |
| low_allele_frac | Соматическая | FilterMutectCalls | Доля аллеля ниже установленного порога |
| LowQual | Соматическая | FilterMutectCalls | Низкое качество |
| map_qual | Соматическая | FilterMutectCalls | Среднее качество картирования прочтений, поддерживающих альтернативный аллель |
| MQ | Герминальная | RMMSMapping- Quality | Среднеквадратичное значение качества сопоставления прочтений по всем образцам. Представляет собой оценку общего качества картирования прочтений, покрывающих сайт варианта, усредненную по всем образцам в когорте, т.е. квадратный корень среднего квадрата качества картирования сайта варианта. Таким образом, оценка учитывает стандартное отклонение качества картирования. |
| MQ_RANK_SUM | Герминальная | MappingQuality- RankSumTest | Тест суммы рангов для сопоставления качества картирования прочтения REF и качества картирования прочтения ALT. MQRankSum на уровне варианта сравнивает качество картирования прочтений, поддерживающих референсный аллель, с прочтениями, поддерживающими альтернативный аллель. Обнаружение любой статистически значимой разницы в качестве предполагает, что процесс секвенирования и/или картирования мог быть искажен артефактом. |
| mt_many_low_hets | Соматическая | FilterMutectCalls | Фильтр сайтов с низкой гетероплазмией |
| multiallelic | Соматическая | FilterMutectCalls | Мутация располагается в мультиаллельном сайте: слишком много альтернативных аллелей удовлетворяют LOD score (вероятный признак артефакта) |
| n_ratio | Соматическая | FilterMutectCalls | Отношение количества неизвестных нуклеотидов N к количеству альтернативных аллелей превышает установленный порог |
| normal_artifact | Соматическая | FilterMutectCalls | Нормальный артефакт: вариант, наблюдаемый в опухоли, также присутствует как артефакт в контрольном образце |
| numt_chimera | Соматическая | FilterMutectCalls | Вариант, расположенный в ядерной копии гена мтДНК (NuMT), со слишком большим количеством прочтений, поддерживающих альтернативный аллель, происходящих из аутосомы |
| numt_novel | Соматическая | FilterMutectCalls | Глубина альтернативного аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| orientation | Соматическая | FilterMutectCalls | Фильтр смещения ориентации - смещения, возникающего из-за химического изменения нуклеотида во время подготовки библиотеки, которое приводит к неправильному спариванию основанию (G с A, T c C и т.д.) (артефакт секвенирования) |
| OTHER | Любая | Значение колонки не распознано | |
| panel_of_normals | Соматическая | FilterMutectCalls | Сайт находится в "черном списке" нормальной панели (PON). Как выбрать панель, описано здесь. |
| position | Соматическая | FilterMutectCalls | Медианное расстояние альтернативных мутаций от конца прочтения |
| possible_numt | Соматическая | FilterMutectCalls | Глубина аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| QD | Герминальная | QualByDepth | Достоверность варианта (значение из поля QUAL), нормализованная по нефильтрованнной глубине аллеля (AD) варианта в выборке вариантов с генотипом, отличным от hom-ref (homozygous/reference; генотип, при котором оба аллеля являются референсными) |
| QUAL | Герминальная | Качество варианта - вероятность существования (по шкале Phred) варианта в этом сайте с учетом данных секвенирования | |
| READ_POS_RANK_SUM | Герминальная | ReadPosRank- SumTest | Тест суммы рангов для относительных позиций аллелей REF и ALT в прочтениях. ReadPosRankSum на уровне варианта проверяет, различаются ли положения референсного и альтернативного аллеля в прочтениях, которые поддерживают вариант. Нахождение аллеля только вблизи концов прочтений указывает на ошибку, потому что именно там секвенаторы склонны делать больше всего ошибок. Обнаружение статистически значимой разницы в относительном положении в любом случае указывает на то, что процесс секвенирования мог быть искажен артефактом. |
| SITE | Соматическая | FilterMutectCalls | Ошибка сайта |
| slippage | Соматическая | FilterMutectCalls | Фильтр ошибок проскальзывания полимеразы в ходе ДНК-репликации (предсказывается по количеству и длине коротких тандемных повторов (STR)) |
| SOR | Герминальная | StrandOddsRatio | Смещение цепей, оцененное с помощью симметричного теста отношения шансов. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. StrandOddsRatio оценивает наличие смещения между прямыми и обратными цепями для референсного или альтернативного аллеля (аллелей). |
| strand_bias | Соматическая | FilterMutectCalls | Фильтр смещения цепей - смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим (артефакт секвенирования) |
| strict_strand | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля не представлены в обоих направлениях прочтения |
| weak_evidence | Соматическая | FilterMutectCalls | Мутация не соответствует порогу вероятности |
- Колонка "ClinVar" содержит информацию о клинической значимости варианта в
базе ClinVar.
В форме добавления колонок она
называется "Clinical significance" (раздел "ClinVar"). Значения колонки представлены в виде
значков:
- Аллель вероятного риска;
- Ассоциация не найдена;
- Аллель неопределенного риска;
- Аллель установленного риска;
- Ассоциация;
- Вероятно доброкачественный;
- Влияет;
- Вероятно патогенный или Вероятно патогенный с низкой пенетрантностью;
- Доброкачественный;
- Другое;
- Даёт чувствительность;
- Защитный;
- Конфликт: Противоречивые интерпретации патогенности или Противоречивые данные от отправителей;
- Лекарственная реакция;
- Неопределенная значимость;
- Не предоставлена;
- Патогенный или Патогенный с низкой пенетрантностью;
- Фактор риска.
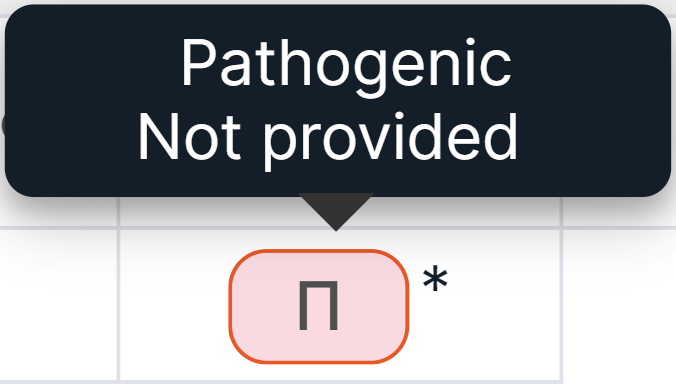
При наведении курсора на значок можно посмотреть полное значение. Значения, состоящие из нескольких значимостей, обозначены звездочкой, а значком в таком случае обозначена первая значимость из списка:

- Колонка "ENIGMA" содержит информацию о клинической значимости варианта, если он расположен в известном или
предполагаемом гене предрасположенности к раку молочной железы и/или яичников (BRCA1, BRCA2 и др.),
выявленной экспертами из консорциума ENIGMA.
В форме добавления колонок она
называется "ENIGMA clinical significance" (раздел "Other"). Значения колонки представлены
в виде значков:
- Патогенный;
- Вероятно патогенный;
- Вероятно доброкачественный;
- Доброкачественный;
- Не предоставлена.
При наведении курсора на значок можно посмотреть полное значение. Значения, состоящие из нескольких значимостей, обозначены звездочкой, а значком в таком случае обозначена первая значимость из списка: