Патогенность варианта
Патогенность варианта можно определить либо самостоятельно, либо по критериям ACMG, либо по базе патогенности,
загруженной в виде пользовательской аннотации.
Информация о патогенности варианта находится в колонке "Патогенность" в SNV Viewer.
Если вы не определили патогенность варианта или не загрузили базу патогенности, то
значение колонки будет (нет интерпретации).
Самостоятельное определение патогенности#
Чтобы определить патогенность варианта самостоятельно,
нажмите на кнопку патогенности
в строке варианта (колонка "Патогенность") и в появившемся диалоге выберите нужную опцию:

Все варианты с определённой патогенностью автоматически добавляются в результаты интерпретации образца. Если вы хотите определить патогенность варианта, но не добавлять его в результаты интерпретации, отключите опцию "Включить в результаты интерпретации" в том же диалоге.
На заметку
Если вариант был определен как Патогенный или Вероятно патогенный, он автоматически добавляется в отчёт (если патогенность была подтверждена).
Патогенность можно переопределить в том же диалоговом окне.
Вы также можете определить патогенность на вкладке "ACMG классификация", воспользовавшись дополнительной информацией о критериях ACMG, как описано здесь.
На заметку
Класс патогенности варианта, определенный самостоятельно, сохраняется в системе и автоматически определится для такого же варианта в образцах, проаннотированных позднее, с предупреждением о необходимости подтвердить интерпретацию.
ACMG классификация#
"ACMG классификация" - это вкладка
на странице детальной информации о варианте, на которой
представлены критерии для интерпретации вариантов,
разработанные American College of Medical Genetics and Genomics (ACMG). Чтобы попасть
на страницу, нажмите на кнопку патогенности
в строке варианта (колонка "Патогенность") и в появившемся диалоге нажмите на строку "ACMG Классификация":

Откроется вкладка "ACMG классификация" на странице детальной информации о варианте (на вкладку можно также попасть и с других вкладок страницы).
Слева на вкладке - панель навигации критериев ACMG, сгруппированных по категориям. При нажатии на название определенной группы критериев она отобразится справа на странице, а при нажатии на определенный критерий будет осуществлён переход к соответствующему критерию группы. Серым обозначены критерии, которые не были определены для варианта; чёрным - критерии, которые варианту не подходят; зелёным - доброкачественные критерии, подходящие варианту; оранжевым - патогенные критерии, подходящие варианту.
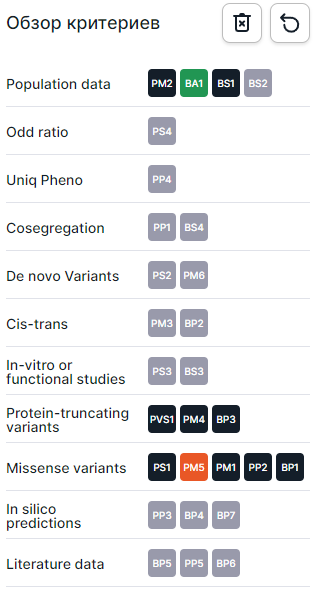
Справа на вкладке приведена подробная информация о каждом критерии по группам. У каждого критерия есть:
- Код и значимость критерия, представленные в графической форме. Палитра цветов соответствует цветам на панели навигации слева: серый - критерий не определен для варианта; чёрный - критерий не подходит варианту; зелёный - доброкачественный критерий, который подходит варианту; оранжевый - патогенный критерий, который подходит варианту.
- Решение: Automatic (состояние критерия выставлено автоматически), UNDEFINED (состояние критерия не определено) или
MANUALLY CHANGED (состояние критерия было определено пользователем; для возврата к автоматическому определению
состояния и уровня значимости критерия нажмите
);
- Состояние критерия: UNDEFINED - критерий не определен для варианта; UNMET - критерий не подходит варианту; MET - критерий подходит варианту. Выбранное состояние обозначено синим. Состояние критерия можно поменять, кликнув на кнопку нужного состояния.
- Критерий можно развернуть, нажав на
. Здесь можно добавить комментарий к критерию и поменять уровень значимости критерия:
- SUPPORTING - вспомогательный критерий;
- MODERATE (если нет опции "Stand alone") - критерий средней значимости;
- STRONG - критерий высокой значимости;
- VERY STRONG (если нет опции "Stand alone") - критерий очень высокой значимости;
- STAND ALONE (если нет опций "Moderate" и "Very strong") - независимый критерий очень высокой значимости.
Здесь же будет находиться раздел "Литература" со списком публикаций, связанных с критерием, если такие связи были добавлены на вкладке "Литература".
1. Популяционные данные (Population data)#
Сверху раздела приведены:
- gnomAD 3 AF - суммарная частота аллеля в базе gnomAD (версия 3);
- ExAC AF - суммарная частота аллеля в базе ExAC;
- 1000G AF - суммарная частота аллеля из проекта 1000 Genomes;
- Max AF - максимальная частота аллеля среди всех частот в разных популяцях по данным разных баз;
- Min AF - минимальная частота аллеля среди всех частот в разных популяцях по данным разных баз.
Критерии раздела "Популяционные данные"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Pathogenic Moderate 2 (PM2) | Средний | Патогенный | Отсутствует в контрольной выборке (или встречается с очень низкой частотой, если рецессивный: ≤0.01% для заболеваний с аутосомно-доминантным типом наследования или ≤0.5% для заболеваний с аутосомно-рецессивным типом наследования) в gnomAD, 1000 Genomes или ExAC. Должен быть покрыт до достаточной глубины/качества прочтения (>100) в базах данных. |
| Stand-alone evidence of benign impact 1 (BA1) | Очень высокий; независимый критерий | Доброка- чественный | Частота альтернативных аллелей >5% в глобальной популяции в gnomAD, 1000 Genomes и ExAC и вариант не входит в список исключений ClinGen SVI. |
| Strong evidence of benign 1 (BS1) | Высокий | Доброка- чественный | Частота альтернативных аллелей >1%, должна быть выше ожидаемой для заболевания: 1. Варианты, известные как патогенные для заболеваний с доминантным типом наследования, должны иметь частоты альтернативных аллелей в глобальной популяции ниже частоты заболевания (распространённости). 2. Варианты, известные как патогенные для заболеваний с рецессивным типом наследования, должны иметь гетерозиготные частоты, соответствующие частоте заболевания (распространённости). - Для заболевания с аутосомно-доминантным типом наследования и высокой пенетрантностью допустимо использовать этот критерий как независимое доказательство того, что вариант вероятно доброкачественный. - Будьте осторожны, используя критерий, основываясь на низкой частоте аллелей в gnomAD, для заболеваний с поздним началом или переменной пенетрантностью/экспрессивностью. - Для избегания дублирования отмечается как UNMET, если вариант отвечает критериям BA1 или PM2. |
| Strong evidence of benign 2 (BS2) | Высокий | Доброка- чественный | Наблюдается у здорового взрослого человека при заболеваниях с полной пенетрантностью, манифестирующих в детском возрасте, (1) в гомозиготном состоянии с рецессивным типом наследования; (2) в гетерозиготном состоянии с доминантным типом наследования или (3) в гемизиготном состоянии с Х-сцепленным типом наследования. - Используйте критерий, если есть основания полагать, что люди, у которых встретился вариант по данным баз, действительно были здоровы. Доказательства можно получить из gnomAD, публикаций или ваших собственных данных. Например, gnomAD показывает возрастное распределение носителей гетерозиготного или гомозиготного варианта. - Будьте осторожны, используя критерий, если в gnomAD приведены данные для небольшой выборки людей для заболеваний с поздним началом или переменной пенетрантностью/экспрессивностью. |
2. Отношение шансов (Odds ratio)#
Критерий раздела "Отношение шансов"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию | Дополнительные опции |
| Pathogenic Strong 4 (PS4) | Высокий | Патогенный | Распространенность варианта у больных индивидуумов значительно выше, чем у контрольной группы. Отношение шансов, полученное из исследований случай-контроль, составляет > 5.0, а доверительный интервал вокруг оценки не включает 1.0. - Поскольку данные исследований случай-контроль редко доступны и не достигают показателей статистической значимости для очень редких вариантов и редких заболеваний, используйте критерий: 1. на среднем уровне значимости (MODERATE), если этот вариант был предварительно идентифицирован у нескольких (двух или более) не связанных родством пациентов с одинаковым фенотипом и отсутствует в контрольной группе в gnomAD (или встречается с очень низкой частотой при аутосомно-рецессивным типе наследования); 2. на вспомогательном уровне (SUPPORTING), если этот вариант был предварительно идентифицирован у одного больного индивидуума и отсутствует в контрольной группе в gnomAD (или встречается с очень низкой частотой при аутосомно-рецессивным типе наследования); 3. на вспомогательном уровне (SUPPORTING), если этот вариант был предварительно идентифицирован у нескольких не связанных родством пациентов, но присутствует в контрольной группе. - При этом фенотип пациента должен соответствовать известному спектру заболеваний. - На практике это наиболее применимо к аутосомно-доминантным заболеваниям (для аутосомно-рецессивных заболеваний используйте PM3). - Критерии PS4 и PM2 можно использовать вместе. | Чтобы открыть вариант в каталоге полногеномного поиска ассоциаций человека GWAS Catalog, разверните описание критерия, нажав на |
3. Специфичный фенотип (Uniq Pheno)#
Критерий раздела "Специфичный фенотип"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Supporting evidence of pathogenicity 4 (PP4) | Вспомогательный критерий | Патогенный | Фенотип пациента и/или семейный анамнез высоко специфичны для заболевания с данной генетической этиологией. - Перед использованием критерия важно убедиться, что: 1. все известные гены, связанные с заболеванием, были проанализированы с помощью высокочувствительного метода, подходящего для известных типов вариантов; 2. варианты в этих известных генах объясняют большинство случаев с этим клиническим диагнозом. |
4. Косегрегация (Cosegregation)#
Сверху раздела можно вычислить отношение правдоподобия шансов (LOD-оценку) для определенного типа сегрегации. LOD-оценки, рассчитанные для разных семей с одним и тем же вариантом, могут быть добавлены для расчета окончательного показателя. Окончательную LOD-оценку можно затем использовать для определения уровня значимости критериев. Для расчёта LOD выполните следующее:
- Выберите тип сегрегации: Доминантная/X-связанная или Рецессивная;
- Введите значения количеств затронутых сегрегаций и незатронутых сегрегаций (если был выбран рецессивный тип сегрегации) (натуральные числа);
- Кликните вне области заполнения значений.
Значение показателя LOD для доминантной/X-связанной сегрегации рассчитывается по формуле:
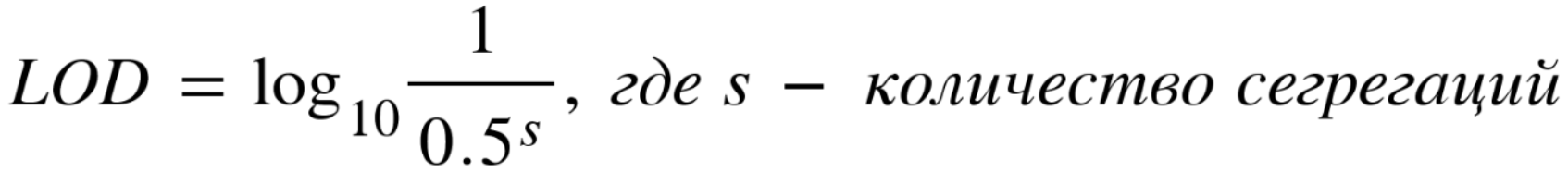
Значение показателя LOD для рецессивной сегрегации рассчитывается по формуле:

Критерии раздела "Косегрегация"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Supporting evidence of pathogenicity 1 (PP1) | Вспомо- гательный критерий | Патогенный | Находится в гене, который, как известно, вызывает заболевание, и для которого точно установлена связь с косегрегацией с заболеванием у нескольких пораженных членов семьи. - Перед использованием критерия определите сегрегацию варианта с фенотипами заболевания в родословных больных пациентов. Используйте критерий с соответствующим уровнем значимости, если было обнаружено, что вариант косегрегирует с несколькими пораженными членами семьи. Примените BS4, если сегрегация варианта с пораженными членами семьи не была найдена. - The Hearing Loss Working Group рекомендует использовать критерий: 1. на вспомогательном уровне (SUPPORTING), если LOD = 0.6; 2. на среднем уровне значимости (MODERATE), если LOD = 1.2; 3. на высоком уровне значимости (STRONG), если LOD = 1.5. |
| Strong evidence of benign 4 (BS4) | Высокий | Добро- качественный | Находится в гене, для которого точно установлено отсутствие косегрегации с заболеванием у пораженных членов семьи. - Перед использованием критерия определите сегрегацию варианта с фенотипами заболевания в родословных больных пациентов. Примените критерий, если сегрегация варианта с пораженными членами семьи не была найдена. Используйте PP1 с соответствующим уровнем значимости, если было обнаружено, что вариант косегрегирует с несколькими пораженными членами семьи. |
5. Варианты de novo (De novo Variants)#
The Sequence Variant Interpretation Working Group предлагает балльную систему для определения уровня значимости критериев для вариантов de novo (критерии PS2 и PM6) на основе трех параметров:
- выберите PS2, если статус de novo подтвержден, и PM6, если нет;
- фенотипическая согласованность;
- количество наблюдений de novo.
Критерии раздела "Варианты de novo"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Pathogenic Strong 2 (PS2) | Высокий | Патогенный | De novo у пациента с заболеванием и отсутствием семейного анамнеза. Обязательно подтверждение отцовства и материнства. |
| Pathogenic Moderate 6 (PM6) | Средний | Патогенный | Предполагаемый de novo, но без подтверждения отцовства и материнства. |
Для критериев раздела можно добавить дополнительных неродственных пациентов (пробандов) с тем же фенотипом:
- Разверните описание критерия, нажав на
;
- Для пробанда 1 (название можно поменять, нажав на нынешнее и введя новое) нажмите на окно поиска и выберите соответствующую согласованность между фенотипом и геном;
- Чтобы добавить ещё пациента, нажмите на
.
После добавления всех пациентов нажмите на появившуюся кнопку для применения уровня значимости критерия, соответствующего фенотипам пациентов:
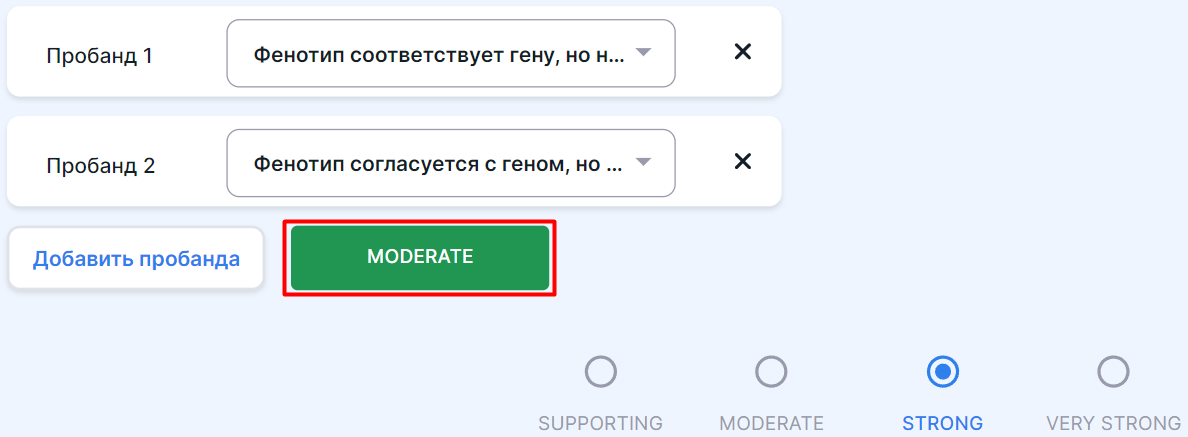
После этого состояние критерия сменится на MET (критерий подходит варианту) и уровень значимости критерия будет соответствовать фенотипам добавленных пациентов.
Для удаления пациента нажмите на .
6. Цис/транс-положение (Cis-trans)#
Критерии раздела "Цис/транс-положение"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию | Дополнительные опции |
| Pathogenic Moderate 3 (PM3) | Средний | Патогенный | Находится в транс-положении с описанным патогенным вариантом при рецессивных заболеваниях. - The Sequence Variant Interpretation (SVI) Working Group предлагает балльную систему для определения уровня значимости критерия PM3 для вариантов в транс-положении на основе фазирования вариантов и классификации варианта, встречающегося на другом аллеле. - SVI рекомендует использовать новое определение критерия: "вариант, находящийся в транс-положении по отношению к известному патогенному или вероятно патогенному варианту, при рецессивных заболеваниях у индивидуума, подверженного патологии". | Можно добавить дополнительных неродственных пациентов (пробандов), у которых был обнаружен тот же вариант: 1. Разверните описание критерия, нажав на 2. Для пробанда 1 (название можно поменять, нажав на нынешнее и введя новое) нажмите на активное окно поиска и выберите соответствующую фазу варианта; 3. В активированном окне поиска выберите классификацию варианта; 4. Чтобы добавить ещё пациента, нажмите на После добавления всех пациентов нажмите на появившуюся кнопку для применения уровня значимости критерия, соответствующего пациентам. После этого состояние критерия сменится на MET (критерий подходит варианту) и уровень значимости критерия будет соответствовать классификации варианта у добавленных пациентов. Для удаления пациента нажмите на |
| Supporting evidence of benign impact 2 (BP2) | Вспомо- гательный критерий | Добро- качественный | Находится либо в транс-положении с (вероятно) патогенным вариантом, соответствующим фенотипу, для заболевания с полной пенетрантностью и доминантным типом наследования, либо в цис-положении с (вероятно) патогенным вариантом, соответствующим фенотипу, для заболевания с полной пенетрантностью и любым типом наследования. |
7. Функциональные исследования (In vitro or functional studies)#
Функциональные исследования (in vitro или in vivo), рассматриваемые при применении критериев раздела, должны быть валидируемы, воспроизводимы и проведены лабораториями с хорошей репутацией. Не используйте исследования in silico, включая моделирование белков.
Критерии раздела "Функциональные исследования"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Pathogenic Strong 3 (PS3) | Высокий | Патогенный | Функциональные исследования подтверждают патогенный эффект варианта на ген или продукт гена. - Уровень значимости критерия зависит от силы доказательств (например, если доступно только одно исследование, используйте критерий с более низкой значимостью). Как правило, исследования in vivo дают более убедительные доказательства, чем исследования in vitro, а исследования, восстанавливающие фенотип, дают более убедительные доказательства, чем исследования, манипулирующие им. - Если исследования дают доказательства на уровне гена, а не варианта (например, биохимический анализ), это должно быть включено в критерий фенотипической специфичности PP4. |
| Strong evidence of benign 3 (BS3) | Высокий | Доброкачественный | Функциональные исследования подтверждают отсутствие патогенного эффекта варианта на ген или продукт гена. |
8. Варианты с укорочением белка (Protein-truncating variants)#
Критерии раздела "Варианты с укорочением белка"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию | Дополнительные опции |
| Pathogenic Very Strong 1 (PVS1) | Очень высокий | Патогенный | LoF-варианты в генах, где данный тип варианта является известной причиной развития заболевания. LoF-варианты - варианты, приводящие к прекращению синтеза белка: нонсенс-мутации; мутации со сдвигом рамки считывания; изменения канонических ±1 или ±2 нуклеотидов сайта сплайсинга; варианты, приводящие к исчезновению стоп-кодона; делеции/дупликации одного или нескольких экзонов. - Не используйте в сочетании с PM4 или PP3. | Чтобы воспользоваться деревом решений для определения LoF-эффекта варианта в контексте структуры гена и молекулярной этиологии, такой как альтернативный сплайсинг или нонсенс-опосредованный распад мРНК, нажмите на |
| Pathogenic Moderate 4 (PM4) | Средний | Патогенный | Вариант, приводящий к синтезу белка с изменённой длиной из-за делеций/инсерций в рамках считывания или потери стоп-кодона в неповторяющейся области гена. - Не используйте в сочетании с PVS1 или PP3. - Используйте критерий на вспомогательном уровне (SUPPORTING) для делеции/инсерции одной аминокислоты внутри рамки считывания. | |
| Supporting evidence of benign impact 3 (BP3) | Вспомо- гательный критерий | Добро- качественный | Делеции/инсерции с сохранением рамки считывания в повторяющейся области гена. Без известной функции. - Не используйте в сочетании с BP4. |
9. Варианты с заменой аминокислоты (Missense variants)#
Критерии раздела "Варианты с заменой аминокислоты"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Pathogenic Strong 1 (PS1) | Высокий | Патогенный | Приводит к замене на ту же аминокислоту в том же положении, которая была описана ранее как патогенная для этого заболевания. - Как правило, используется для миссенс-вариантов, но также можно использовать на среднем уровне значимости (MODERATE) для вариантов в старт-кодоне. - Используйте критерий на вспомогательном уровне (SUPPORTING) для различных интронных изменений с таким же или более серьезным эффектом сплайсинга по сравнению с прогнозируемым значением эффекта на мРНК/белок. - Будьте осторожны с мутациями, которые влияют не только на белковый/аминокислотный уровень, но и на сплайсинг. |
| Pathogenic Moderate 5 (PM5) | Средний | Патогенный | Новые миссенс-варианты в аминокислотном остатке, где другие миссенс-варианты ранее были установлены как патогенные. - Используйте критерий на высоком уровне значимости (STRONG), если было найдено несколько разных патогенных вариантов в одном и том же остатке. - Используйте критерий на вспомогательном уровне (SUPPORTING), если вариант классифицируется как вероятно патогенный и сообщается только об одном случае. - Будьте осторожны с мутациями, которые влияют не только на белковый/аминокислотный уровень, но и на сплайсинг. |
| Pathogenic Moderate 1 (PM1) | Средний | Патогенный | Расположен в "горячей" точке и/или в важном и хорошо исследованном функциональном домене (например, в активном сайте фермента), в котором нет доброкачественных изменений. Точку можно считать "горячей", если патогенные варианты наблюдаются с высокой частотой в одном или нескольких близлежащих остатках. В качестве подтверждающих доказательств могут быть включены данные моделирования белков in silico. - Используйте критерий на высоком уровне значимости (STRONG) для очень специфических остатков, которые имеют решающее значение для структуры или функции белка. |
| Supporting evidence of pathogenicity 2 (PP2) | Вспомо- гательный критерий | Патогенный | Миссенс-вариант в гене, который имеет низкий уровень доброкачественных миссенс-мутаций и в котором миссенс-варианты являются распространенным механизмом возникновения заболевания. - Значимы Z-оценки ≥3.09 (отмечены оранжевым цветом в gnomAD), но важно учитывать ограничение для области, охватывающей вариант, а не только для всего гена. Z-оценка рассчитывается путем сравнения ожидаемого и наблюдаемого количества миссенс-вариантов. Чем выше Z-оценка, тем выше ограничение или нетолерантность к вариациям. |
| Supporting evidence of benign impact 1 (BP1) | Вспомо- гательный критерий | Добро- качественный | Миссенс-вариант в гене, для которого известной причиной заболевания являются мутации, приводящие к изменению длины белка. - Используйте критерий, если все или почти все (например, >90%) вызывающие заболевание варианты приводят к укорочению длины белка. |
10. Предсказания in silico (In silico predictions)#
Сверху раздела приведены результаты предсказания эффекта аминокислотной замены на функцию белка:
- DBNSFP DANN score - оценка предсказания эффекта, сделанная с помощью DANN;
- DBNSFP MetaLR prediction - значение предсказания эффекта с помощью Meta LR;
- DBNSFP GERP++ RS - оценка (rankscore) предсказания эффекта, сделанная с помощью GERP++.
Критерии раздела "Предсказания in silico"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Supporting evidence of pathogenicity 3 (PP3) | Вспомо- гательный критерий | Патогенный | Результаты не менее трёх программ предсказания подтверждают патогенное воздействие варианта на ген или продукт гена. Предупреждение: поскольку многие программы предсказания используют одни и те же или очень похожие расчёты и входные данные, результаты нескольких программ нельзя рассматривать как независимые критерии и должны оцениваться в сочетании. - Критерий можно использовать для оценки варианта только один раз. - Не используйте в сочетании с PVS1 или PM4. - Используйте PP3, если 2/3 программы предсказывают патогенный эффект, и BP4, если 2/3 программы предсказывают доброкачественный эффект и нет консервативности или 3/3 программы предсказывают доброкачественный эффект. |
| Supporting evidence of benign impact 4 (BP4) | Вспомо- гательный критерий | Добро- качественный | Результаты не менее трёх программ предсказания подтверждают отсутствие воздействия варианта на ген или продукт гена. Предупреждение: поскольку многие программы предсказания используют одни и те же или очень похожие расчёты и входные данные, результаты нескольких программ нельзя рассматривать как независимые критерии и должны оцениваться в сочетании. - Критерий можно использовать для оценки варианта только один раз. - Не используйте в сочетании с BS3. - Используйте PP3, если 2/3 программы предсказывают патогенный эффект, и BP4, если 2/3 программы предсказывают доброкачественный эффект и нет консервативности или 3/3 программы предсказывают доброкачественный эффект. - Используйте в сочетании с BP7 для синонимичных вариантов, затрагивающих слабоконсервативные нуклеотиды, которые не влияют на сплайсинг. |
| Supporting evidence of benign impact 7 (BP7) | Вспомо- гательный критерий | Добро- качественный | Синонимичный ("молчащий") вариант, для которого алгоритмы предсказания сплайсинга показывают отсутствие его влияния на консенсусную последовательность сплайс-сайтов (не создается новый сайт сплайсинга), и нуклеотид не является высококонсервативным в эволюции. - Используйте в сочетании с BP4 для синонимичных вариантов, затрагивающих слабоконсервативные нуклеотиды, которые не влияют на сплайсинг. |
11. Данные литературы (Literature data)#
Критерии раздела "Данные литературы"#
| Критерий | Уровень значимости критерия | Патогенность критерия | Описание варианта, который отвечает критерию |
| Supporting evidence of benign impact 5 (BP5) | Вспомо- гательный критерий | Добро- качественный | Обнаружен в случае с альтернативной молекулярной причиной заболевания (причиной, которая может объяснить фенотип пациента). - Как правило, критерий более применим к доминантным заболеваниям с очевидным альтернативным механизмом. - При рецессивных заболеваниях случайные носители могут иметь патогенные варианты, не связанные с фенотипом заболевания. |
| Supporting evidence of pathogenicity 5 (PP5) | Вспомо- гательный критерий | Патогенный | Источники с хорошей репутацией показали патогенность варианта, но независимая оценка не проводилась. - The Sequence Variant Interpretation Working Group рекомендует не использовать критерии PP5 и BP6. |
| Supporting evidence of benign impact 6 (BP6) | Вспомо- гательный критерий | Добро- качественный | Источники с хорошей репутацией показали отсутствие патогенности варианта, но независимая оценка не проводилась. - The Sequence Variant Interpretation Working Group рекомендует не использовать критерии PP5 и BP6. |
Определение патогенности на вкладке "ACMG классификация"#
- При первом открытии вкладки "ACMG классификация" состояние критериев будет определено для варианта автоматически.
- Вы можете менять состояние и уровень значимости критериев и добавлять к ним комментарии.
- Для удаления всех результатов интерпретации варианта
нажмите на
.
На заметку
Если патогенность была определена с помощью комбинирования критериев ACMG, то после удаления результатов интерпретации вариант автоматически исключается из результатов интерпретации образца.
- Для возврата к автоматической интерпретации варианта
нажмите на
.
В результате комбинирования критериев вариант классифицируется по патогенности (Доброкачественный, Вероятно доброкачественный, Неопределенной значимости, Конфликт: противоречивые интерпретации патогенности, Вероятно патогенный, Патогенный) и результат классификации выставляется на шкале патогенности:
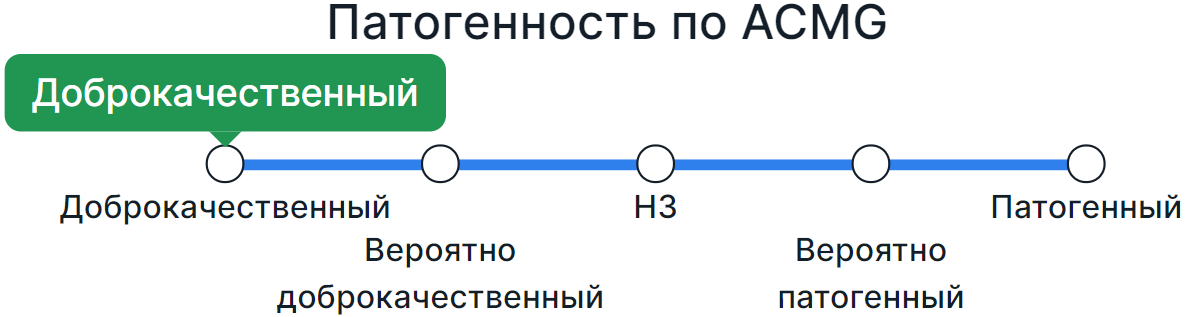
Если класс патогенности варианта был определён по результатам автоматической интерпретации, появится предупреждение о необходимости подтвердить интерпретацию.
- Чтобы самостоятельно классифицировать вариант по патогенности, нажмите на название или точку с нужным значением на шкале:

На заметку
Класс патогенности варианта, определенный самостоятельно, сохраняется в системе и автоматически определится для такого же варианта в образцах, проаннотированных позднее, с предупреждением о необходимости подтвердить интерпретацию.
На заметку
Если вариант был определен как Патогенный или Вероятно патогенный, он автоматически добавляется в отчёт (если патогенность была подтверждена).
Включение в результаты интерпретации#
Все варианты с определённой патогенностью автоматически добавляются в результаты интерпретации образца. Если вы хотите исключить вариант с определённой патогенностью из результатов интерпретации, отключите опцию "Включить в результаты интерпретации" в правом верхнем углу страницы.
Определение патогенности по базе#
Если вы загрузили в систему базу патогенности, как описано здесь, то база будет аннотировать варианты образцов. Таким образом, для вариантов, присутствующих в базе, патогенность будет соответствовать их классу патогенности в базе (с пометкой "определено пользователем"), однако такая классификация требует подтверждения.
Определённая патогенность варианта#
После завершения классификации варианта по патогенности (по ACMG или самостоятельно) или после аннотации вариантов по базе патогенности кнопка патогенности сменится согласно полученному значению:
- Патогенный вариант:
- Вероятно патогенный вариант:
- Вариант неопределенной значимости:
- Конфликт: вариант с противоречивыми интерпретациями
патогенности:
- Вероятно доброкачественный вариант:
- Доброкачественный вариант:
На заметку
Все варианты с определённой патогенностью автоматически добавляются в результаты интерпретации образца. Если вы хотите исключить вариант с определённой патогенностью из результатов интерпретации, отключите опцию "Включить в результаты интерпретации" в диалоге определения патогенности или на странице детальной информации о варианте.
На заметку
Если вариант был определен как Патогенный или Вероятно патогенный, он автоматически добавляется в отчёт (если патогенность была подтверждена).
Способ получения результата классификации варианта ("по ACMG" или "определено пользователем") можно узнать во всплывающей подсказке, наведя курсор на кнопку патогенности.
Варианты можно отсортировать от патогенного к доброкачественному, нажав на название колонки "Патогенность" один раз, и от доброкачественного к патогенному, нажав на название колонки дважды.
Варианты можно отфильтровать по патогенности:
- фильтр "Pathogenicity" в режиме расширенного поиска;
- дополнительный фильтр "Патогенность" в режиме базового поиска (можно добавить на странице "Профиль").
Очистка патогенности#
Результат классификации варианта по патогенности можно удалить, нажав на кнопку патогенности в строке варианта и в появившемся диалоге выбрав опцию "Очистить":
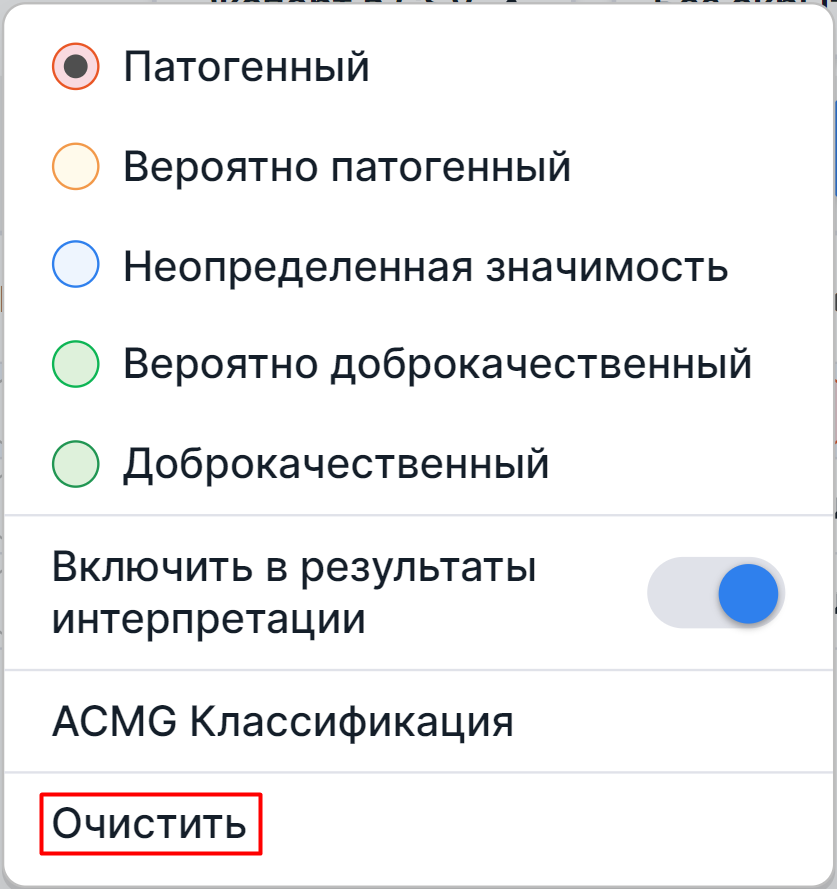
На заметку
После очистки патогенности вариант автоматически исключается из результатов интерпретации образца.
Подтверждение патогенности#
Подтверждение класса патогенности варианта может понадобиться в следующих случаях:
- Была восстановлена автоматическая интерпретация патогенности варианта по ACMG после каких-либо манипуляций с критерями на вкладке "ACMG классификация";
- Была перезапущена аннотация вариантов или более ранние стадии анализа образца;
- Класс патогенности варианта был получен из базы патогенности.
- Класс патогенности варианта был сохранен в системе при самостоятельно определении патогенности.
На странице SNV Viewer варианты, для которых требуется подтверждение патогенности, обозначены
значком рядом с кнопкой патогенности.
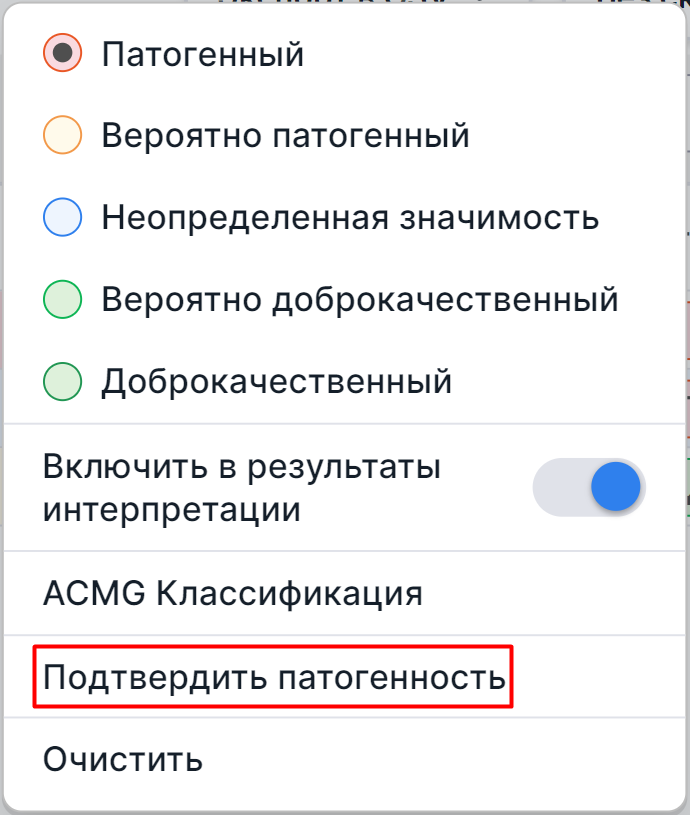
Если вы согласны с классификацией варианта, нажмите на колокольчик или кнопку патогенности и в появившемся
диалоге выберите опцию "Подтвердить патогенность":
На вкладке "ACMG классификация" предупреждение о том, что класс патогенности требуется проверить и подтвердить, выглядит так:

Если вы согласны с классификацией варианта,
нажмите на  .
.
На заметку
После подтверждения патогенности вариант автоматически добавляется в результаты интерпретации образца. Если вы хотите исключить вариант из результатов интерпретации, отключите опцию "Включить в результаты интерпретации" в диалоге определения патогенности или на странице детальной информации о варианте.
На заметку
Вариант можно добавить в результаты интерпретации образца и без подтверждения патогенности варианта - для этого включите опцию "Включить в результаты интерпретации" на странице детальной информации о варианте.