CNV Viewer
Знакомство с CNV Viewer#
CNV Viewer - это встроенный модуль для просмотра и анализа вариаций числа копий (copy number variations; CNVs), выявленных в образце. Доступен на главной странице образца (вкладка Главное), если для него успешно завершилась стадия "Выявление вариации числа копий".
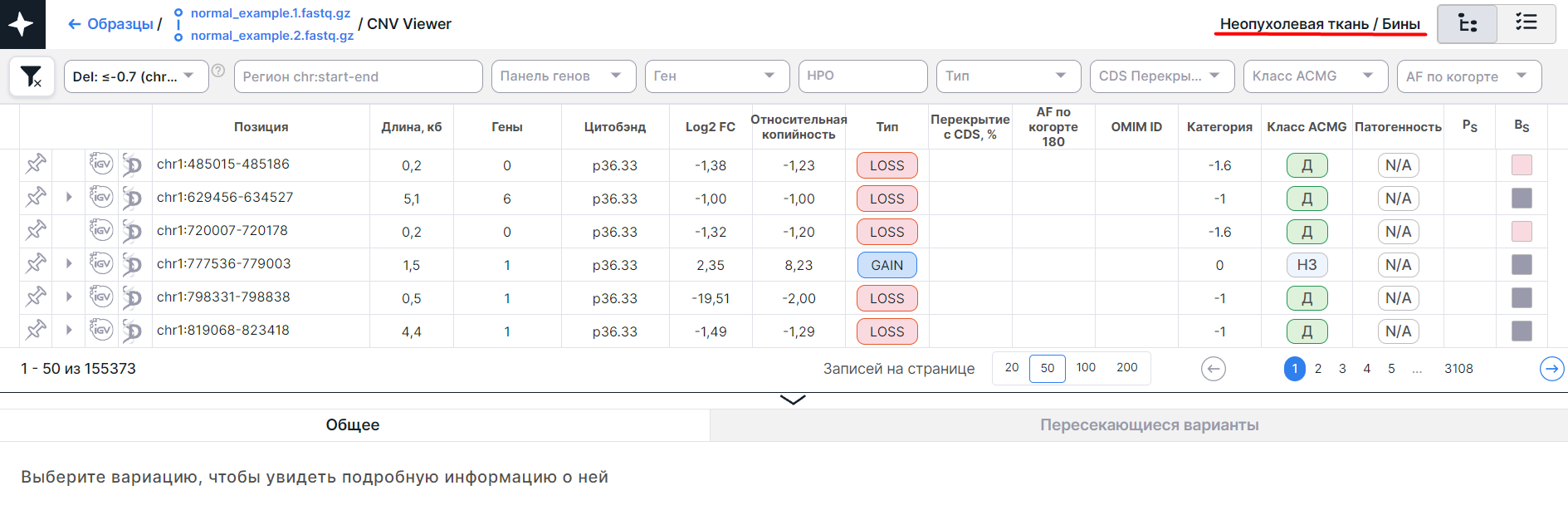
На странице образца неопухолевой ткани представлен CNV Viewer с герминальными вариациями числа копий, выявленными у этого образца:
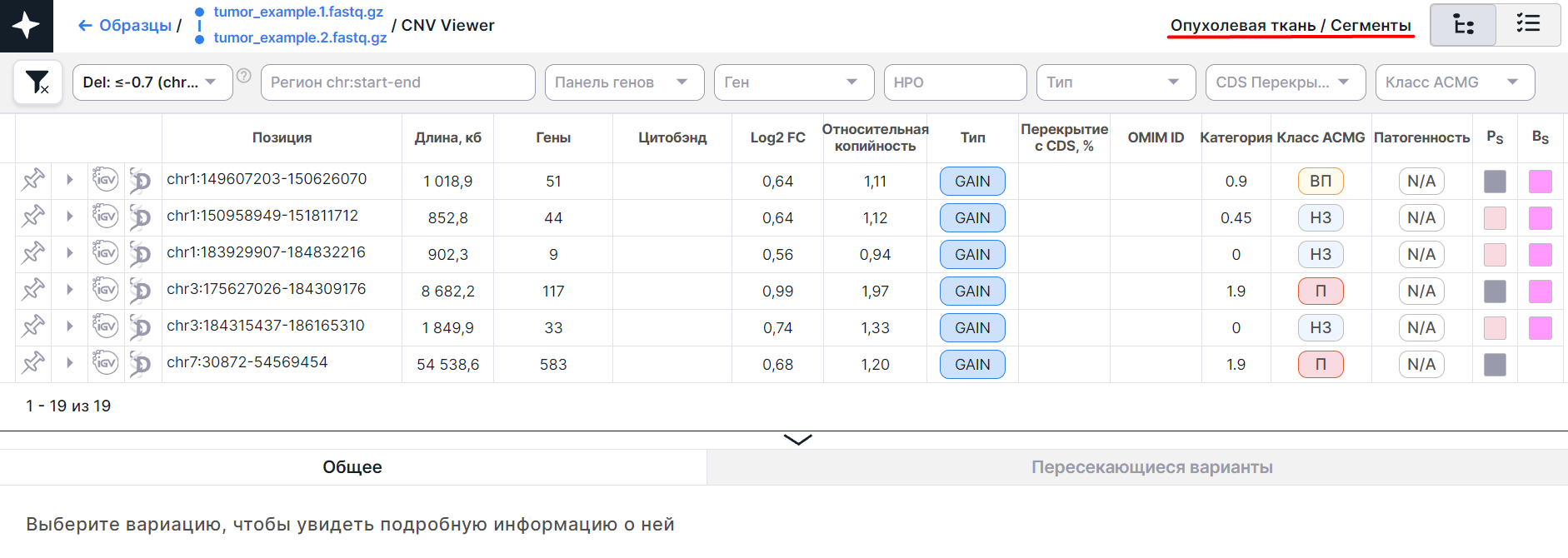
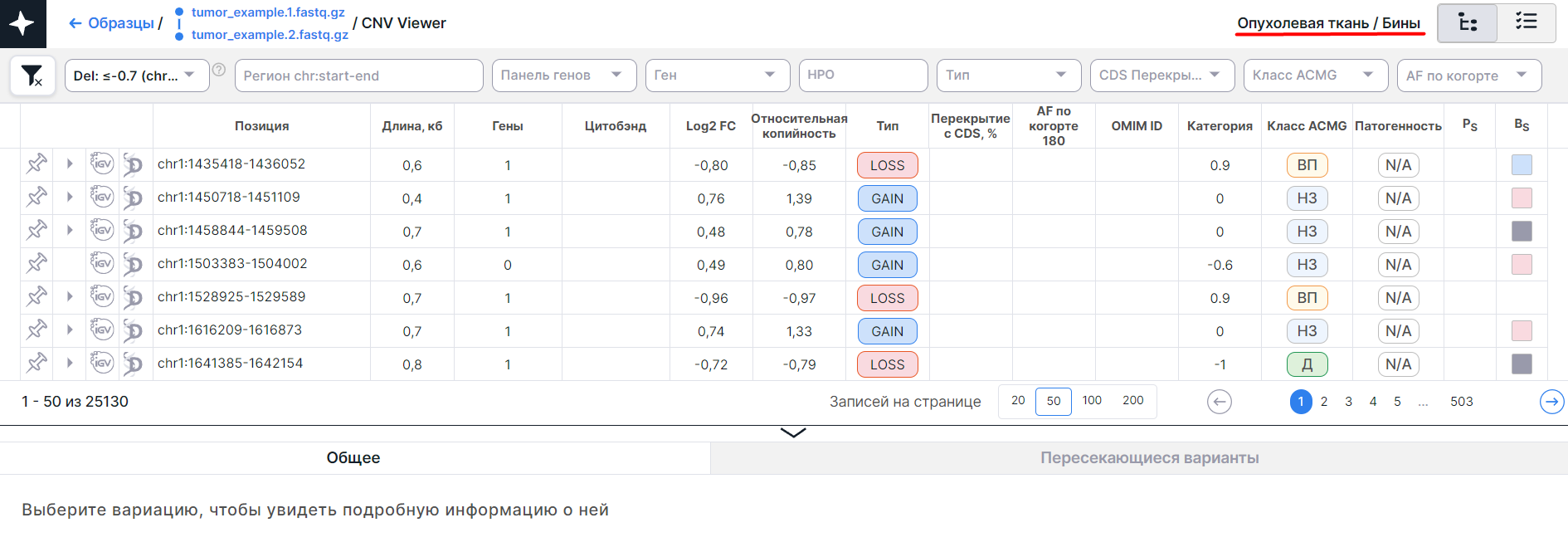
На странице образца опухолевой ткани из пары образцов опухоль/контроль может быть два CNV Viewer: с соматическими вариациями числа копий, выявленными у этого образца, и с герминальными вариациями числа копий, выявленными в контрольном образце:
На странице образца опухолевой ткани, анализировавшегося без контроля, представлен CNV Viewer c соматическими и герминальными вариациями числа копий, выявленными у этого образца:
Сегменты и бины#
Нажав на кнопку  ,
вы увидите две опции: "Сегменты" и "Бины", которые открывают CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно:
,
вы увидите две опции: "Сегменты" и "Бины", которые открывают CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно:

На заметку
Сегменты и бины - это интервалы, в которых проводится подсчёт количества прочтений.
Бины - это небольшие интервалы, размер которых
регулируется соответствующей настройкой для WGS
и которые создаются в результате разделения референсного генома на интервалы равного размера.
Размер бина определяет разрешение выявления вариации числа копий.
Сегменты - это крупные области генома, которые получаются в результате объединения соседних бинов
со схожей копийностью.
Бины обеспечивают мелко раздробленный сигнал числа копий по всему геному без потери информации и дают
некоторый шум. Сегментация пытается удалить этот шум и определить местоположение дискретных вариаций
числа копий, т.е. отдельных областей, которые были продублированы или утеряны. Поэтому в первую очередь
рекомендуется анализировать сегменты, а прибегать к анализу бинов - для отображения уровня поддержки
каждого сегмента, а также для отслеживания потенциальных артефактов, таких как особенно зашумленных
областей генома. Однако стоит учесть, что некоторые выпадающие бины, которые отметаются сегментацией
как зашумлённость, могут оказаться реальными вариациями числа копий, равными по размеру этим бинам.
Основная страница#
Выбрав одну из опций кнопки  :
"Сегменты" или "Бины", вы попадете на основную страницу CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно.
Вверху справа будет отмечено, с какими вариациями открыта страница CNV Viewer:
:
"Сегменты" или "Бины", вы попадете на основную страницу CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно.
Вверху справа будет отмечено, с какими вариациями открыта страница CNV Viewer:
- CNV Viewer с вариациями числа копий, выявленными в сегментах образца неопухолевой ткани:
- CNV Viewer с вариациями числа копий, выявленными в бинах образца неопухолевой ткани:
- CNV Viewer с вариациями числа копий, выявленными в сегментах образца опухолевой ткани:
- CNV Viewer с вариациями числа копий, выявленными в бинах образца опухолевой ткани:
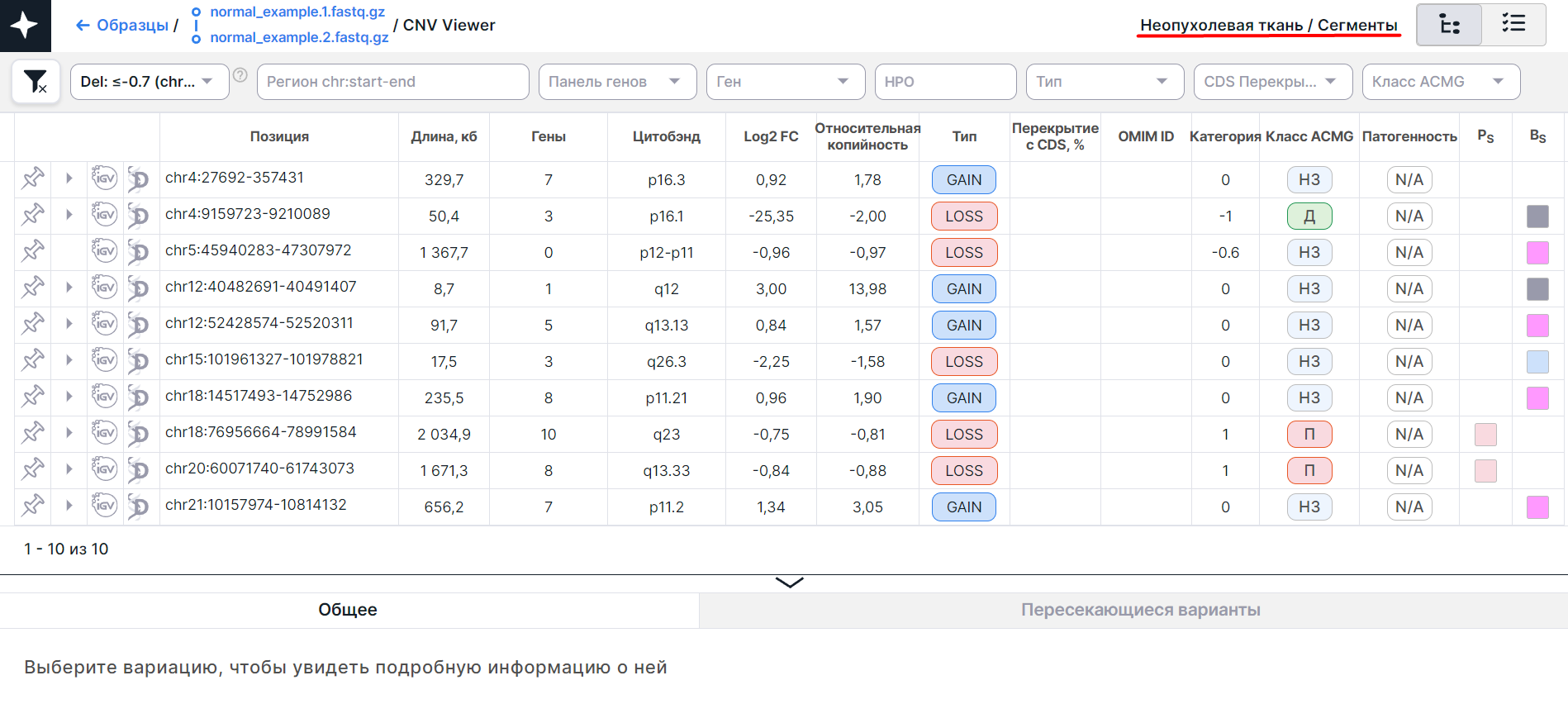
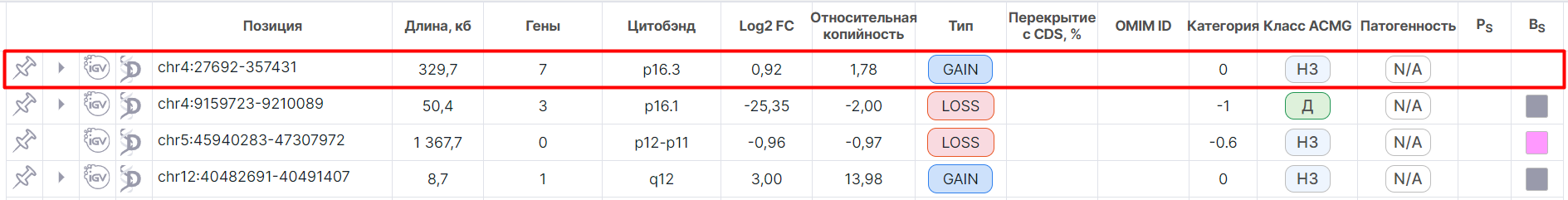
Основным объектом CNV Viewer является таблица с вариациями числа копий - каждая вариация на новой строчке:
Под таблицей, над панелью детальной информации о вариации находятся:
- Информация о количестве вариаций в CNV Viewer: порядок вариаций, отображённых на странице, (например, если максимальное количество вариаций, показываемых на странице, - 50, то порядок будет таким: 1-50, 51-100 и т.д.) и суммарное количество вариаций (закреплённые вариации в подсчёте не учитываются). Количество вариаций изменяется при поиске вариаций, при котором отображается количество вариаций, подходящих под заданные условия. В древовидном режиме отображается количество общих строк, либо, если одна из общих строк была раскрыта - количество детальных строк, соответствующих этой общей строке. В режиме плоского списка отображается количество как общих, так и детальных строк.
- Компонент с выбором количества вариаций, отображаемых на
странице (без учёта закреплённых
вариаций):. По умолчанию отображается 50 вариаций на странице. Чтобы изменить количество отображаемых на странице вариаций на 20, 100 или 200, нажмите на соответствующую опцию. Ваш выбор будет запомнен и будет действовать для CNV Viewer и SNV Viewer всех образцов. Обратите внимание, что чем больше вариаций отображается на странице, тем дольше она будет загружаться.
- Компонент пагинации:
, который позволяет перемещаться между страницами с вариациями. Номер открытой страницы выделен синим кружком. Если нажать на этот номер, то страница перезагрузится. Чтобы переместиться на страницу с конкретным номером, либо нажмите на этот номер, либо нажмите на
, введите нужный номер и нажмите клавишу ввода или кликните вне области заполнения значения. Чтобы переместиться на предыдущую по номеру страницу, нажмите на
, а чтобы открыть следующую - нажмите на
.
Общие и детальные строки#
Для вариации числа копий есть два типа записи: full - участок целиком, и split - участок, разбитый по генам. Каждому full-участку могут соответствовать несколько split-участков. Записи full отображаются в CNV Viewer в виде общих строк, а записи split - в виде детальных строк по генам. Вид строк зависит от режима представления таблицы CNV Viewer.
Режимы CNV Viewer#
Для представления таблицы CNV Viewer существует два
режима: древовидный режим и
режим плоского списка.
Режимы сменяются кнопками переключателя в правом верхнем углу CNV
Viewer: .
На заметку
Выбранный режим и раскрытие общей строки в древовидном режиме (см. далее) сохраняются при выходе со страницы CNV Viewer.
Древовидный режим#
По умолчанию CNV Viewer открывается в древовидном режиме. С этим режимом таблица грузится быстрее, поскольку в нём детальные строки не показываются вместе с общими:
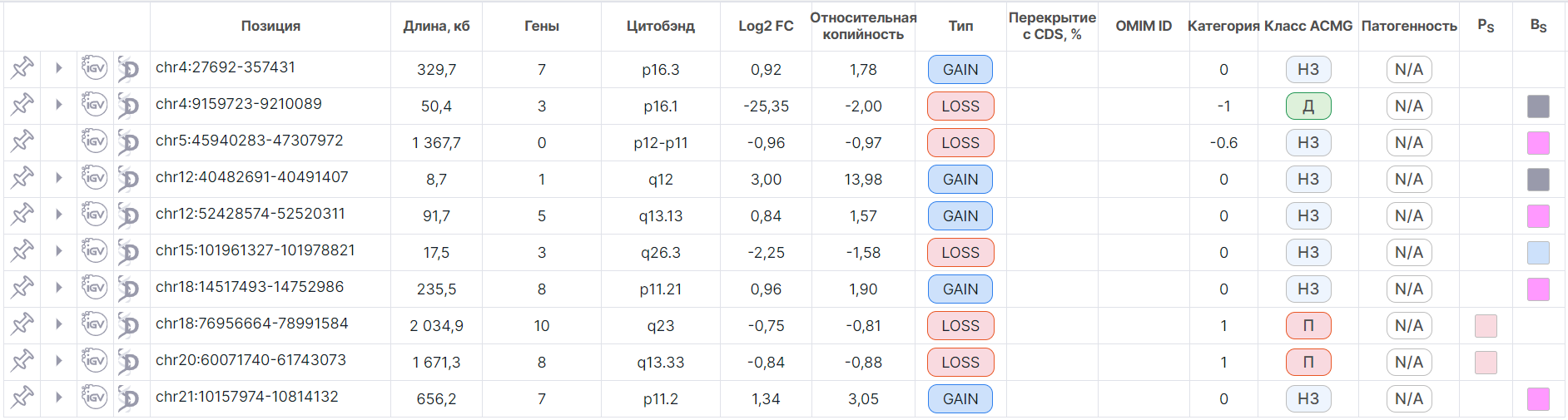
Общую строку можно раскрыть, чтобы увидеть соответствующие детальные строки в этой позиции,
нажав на в общей строке
вариации. В строках, для которых нет соответствующих записей об участках, разбитых по генам, такой
кнопки нет. Раскрытая общая строка с её детальными строками выглядит следующим образом:
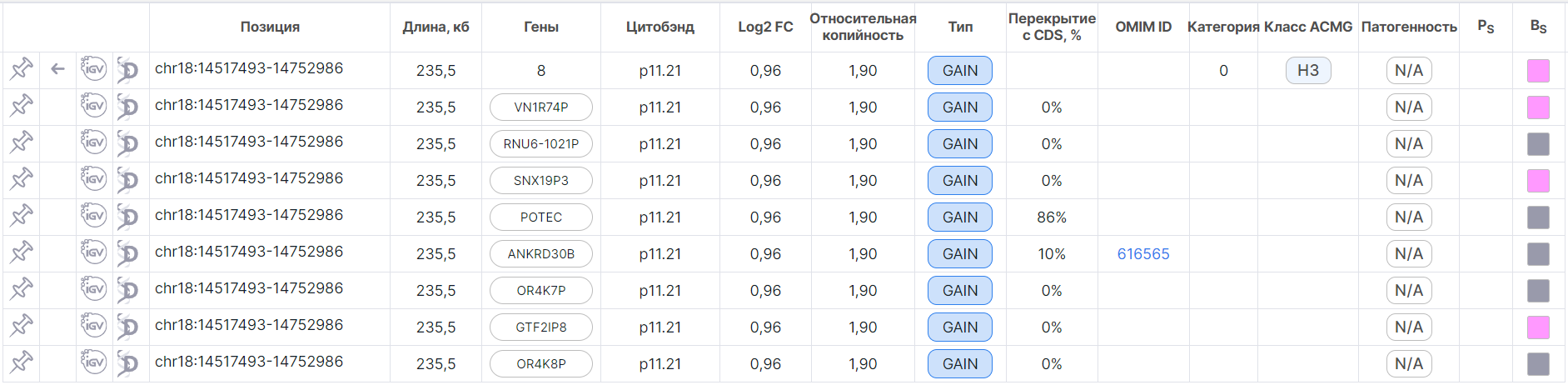
Здесь первая строка - общая, а ниже идут соответствующие детальные строки. Для возврата к таблице со
всеми общими строками без детальных
нажмите на в общей строке вариации.
Для перехода в древовидный режим
из режима плоского списка нажмите
на кнопку переключателя вверху страницы.
Режим плоского списка#
Для перехода в режим плоского списка из древовидного
нажмите на кнопку переключателя вверху страницы.
CNV Viewer работает медленнее в этом режиме, так как в нём отображаются не только общие строки,
но и детальные. При этом общие строки раскрашены серым, а
детальные - белым и записаны под соответствующей общей строкой:
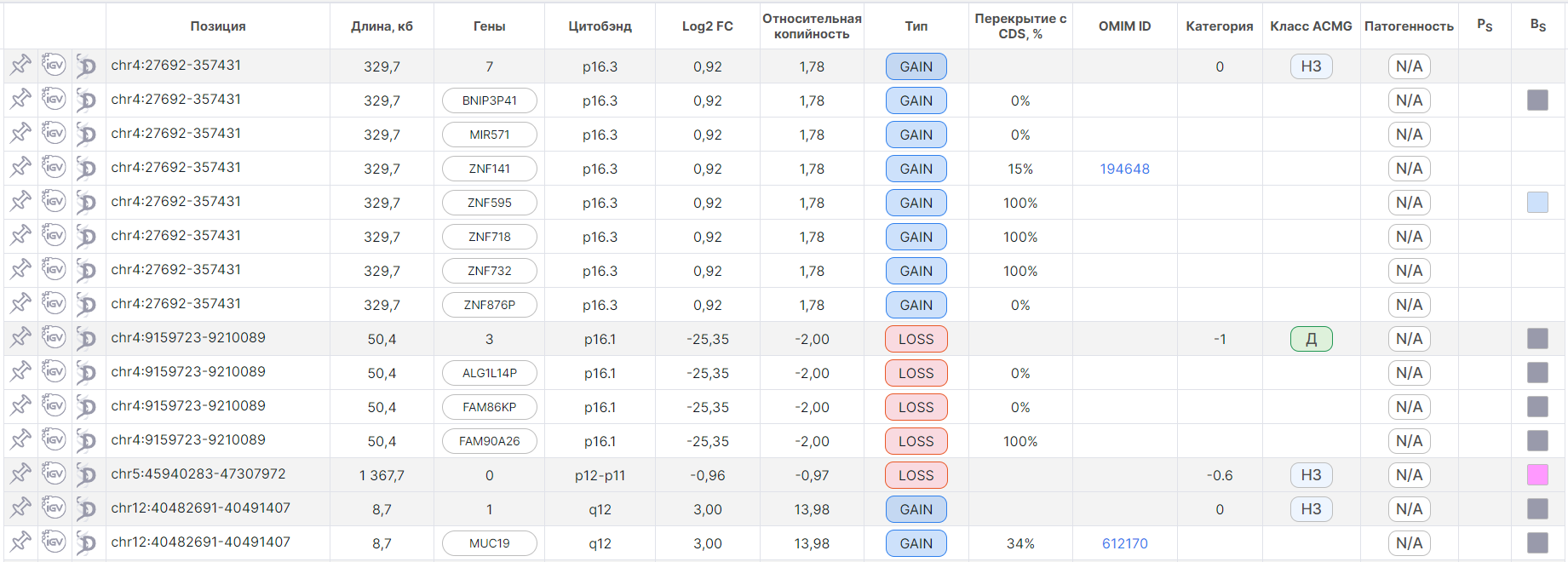
Закрепление вариаций#
Нужные вариации можно закрепить. На закрепленные вариации не влияют фильтрация и сортировка всей таблицы вариаций, они всегда остаются вверху таблицы. Между собой закрепленные вариации отсортированы по позиции.
Чтобы закрепить вариацию,
нажмите на в строке вариации.
В древовидном режиме закреплённая общая строка окажется вверху таблицы над остальными незакреплёнными общими строками (строка подсвечена зелёным):
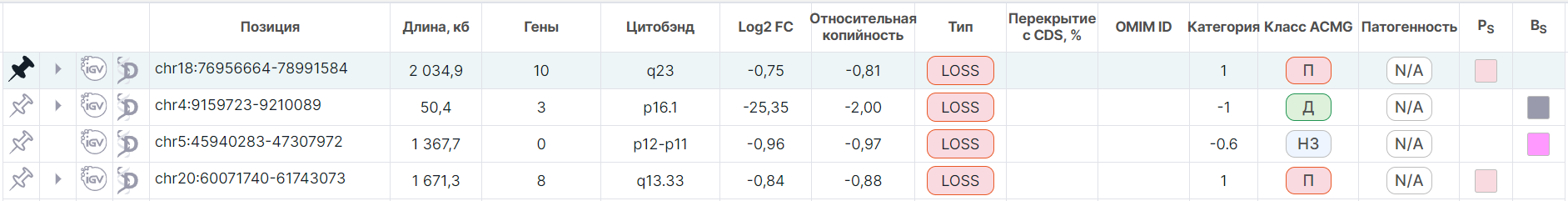
При закреплении детальной строки происходит автоматическое закрепление и соответствующей общей строки. При этом наверху таблицы сохраняется общая строка (она остаётся белой), а закреплённая детальная становится над остальными незакреплёнными детальными строками. Между собой закреплённые детальные строки отсортированы по названию гена.
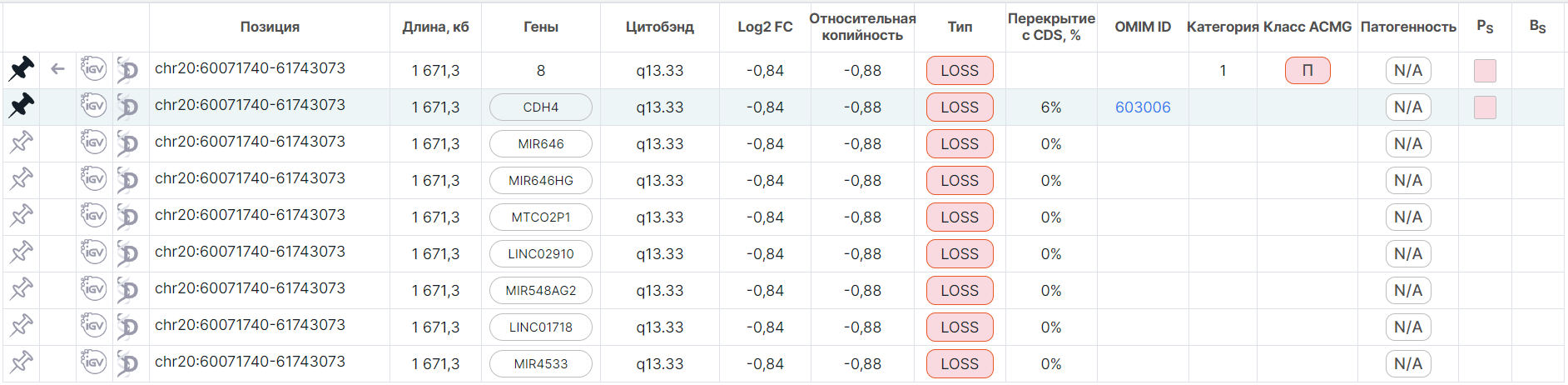
В режиме плоского списка закреплённая общая строка также становится наверх таблицы (строка подсвечена зелёным).
При этом, если для общей строки есть соответствующие записи об участках, разбитых по генам, то её запись
дублируется в таблице над соответствующими детальными строками (дублирующаяся строка подсвечена серым, как остальные
общие строки, а кнопка закрепления имеет вид ).
При выделении одной из этих общих строк: закреплённой или дублирующей, обе строки подсветятся синим.
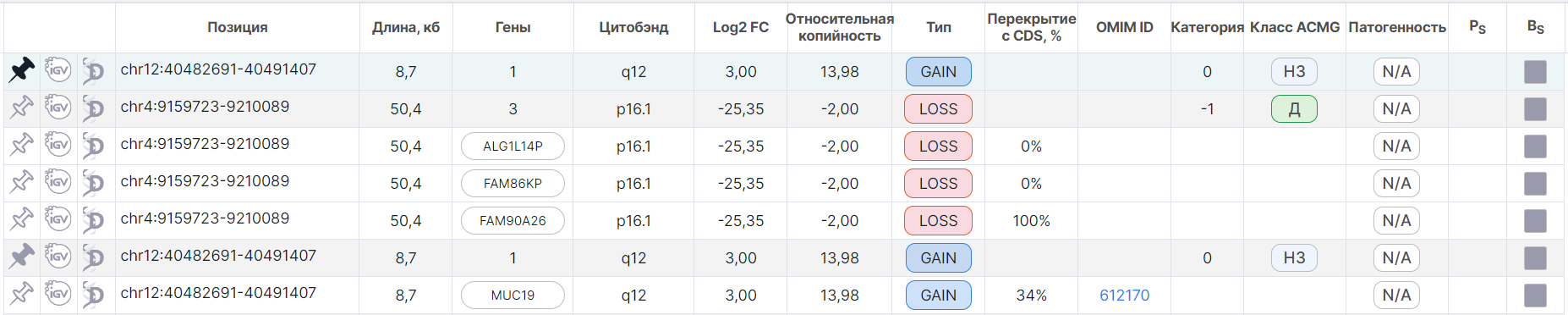
При закреплении детальной строки происходит автоматическое закрепление и соответствующей общей строки. Обе строки перемещаются наверх таблицы и подсвечиваются зелёным. Если у этой общей строки остались ещё незакреплённые соответствующие детальные строки, то она дублируется в таблице, как это описано выше. Если же все детальные строки общей строки закреплены, то дублирующая строка не создаётся.
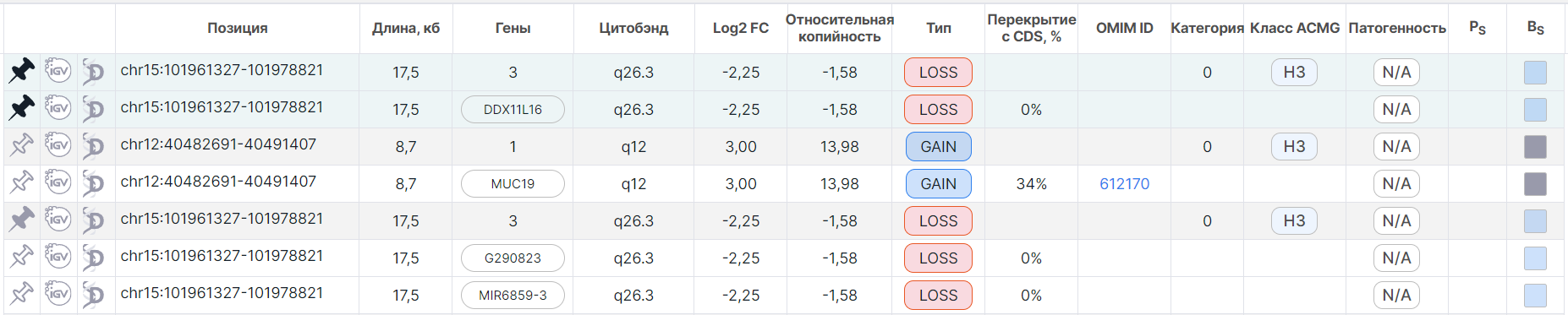
Чтобы открепить вариацию, нажмите на кнопку в строке вариации (или на
в дублирующей общей строке).
На заметку
Закрепление вариации сохраняется при выходе со страницы CNV Viewer.
Информация о вариации#
Помимо представленной в таблице информации (см. Описание колонок таблицы), для каждой вариации есть несколько встроенных модулей с дополнительной информацией:
- Панель детальной информации о вариации. Чтобы увидеть панель, нажмите на строку вариации:
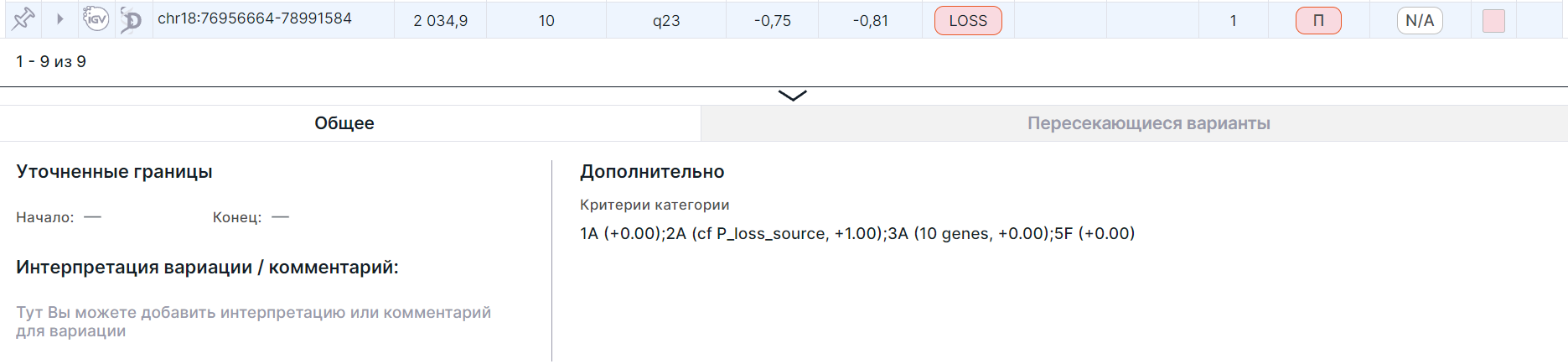
На заметку
По умолчанию панель находится в развёрнутом состоянии внизу таблицы CNV Viewer. Если вы не пользуетесь панелью на постоянной основе, то её можно скрыть из CNV Viewer на странице "Настройки пользователя".
Подробнее с панелью и её вкладками можно ознакомиться в соответствующем разделе.
- Модуль визуализации вариации на геноме (IGV). Чтобы открыть встроенный модуль,
нажмите на
в строке вариации. Чтобы открыть одновременно два региона по границам достаточно крупной перестройки в IGV, нажмите на
, а чтобы вернуться к визуализации всей вариации, нажмите на
. Если вы хотите перейти на страницу позиции, открытой в IGV, в геномном браузере Decipher, нажмите на
. Из IGV, открытого из CNV Viewer, можно вернуться обратно в CNV Viewer с фильтром по позиции, которая открыта в IGV, нажав на кнопку
.
- Ссылка на позицию вариации в геномном браузере Decipher.
Чтобы перейти в браузер, нажмите на
в строке вариации.
Описание колонок таблицы#
Таблица CNV Viewer может включать следующие колонки:
- Позиция - координаты вариации числа копий в геноме (хромосома + начальная позиция + конечная позиция).
Позицию вариации можно скопировать в буфер обмена. Для этого наведите курсор на значение
колонки и нажмите на
.
- Длина, кб - длина участка вариации числа копий в килобазах.
- Гены - для общих строк это количество генов, расположенных на затронутом участке вариации
числа копий, а для детальных строк это название расположенного на участке гена.
Название гена можно скопировать в буфер обмена. Для этого наведите курсор на значение
колонки и нажмите на
.
- Цитобэнд - сокращённая запись положения вариации числа копий на хромосоме. Это положение указывается относительно полос (участков хромосом, чётко отличающихся от соседних), которые образуются на хромосомах в результате применения дифференциальных методов окрашивания. Эти полосы называются бэндами или сегментами. Район - это участок плеча хромосомы между двумя ближайшими маркерными бэндами (самыми заметными и чёткими светлыми или тёмными бэндами). Суббэнды, или субсегменты - это участки хромосом, получающиеся в результате деления бэндов при анализе хромосом с высоким разрешением. В дальнейшем суббэнды могут быть также разделены на части. Принцип обозначение бэндов (полос) на хромосоме следующий: плечо хромосомы (короткое плечо p или длинное плечо q), район, бэнд, суббэнд (может также дополнительно делиться на части). Например, запись "p.36.33" означает, что вариация находится в 3-й части 3-го суббэнда 6-го бэнда 3-го района короткого плеча хромосомы.
- Log2 FC - логарифмическое соотношение выявленного числа копий к нормальному числу копий (равно двум для аутосом и X-хромосомы в случае генотипа XX либо единице для половых хромосом в случае генотипа XY).
- Относительная копийность - количество копий некоторого участка на гаплоидный геном.
- Тип - тип изменения числа копий:
- делеция,
- дупликация.
- Перекрытие с CDS, % - доля длины кодирующей последовательности (CoDing Sequence; CDS), которая перекрывается с вариацией (в процентах). Приводится только для детальных строк.
- AF по когорте 180 - частота вариации в когорте, состоящей из 188 нормальных образцов. Значения частот есть только для герминальных вариаций числа копий, выявленных в образцах полногеномного секвенирования (WGS или low-pass WGS), для нормализации которых использовались панели референсных образцов "BGI WGS" с размером бина 3000 или 5000 или "BGI WGS v2" с размером бина 3000. Колонка отображается только в CNV Viewer с варициями числа копий, выявленными в бинах. Ячейки колонки раскрашены в зависимости от значения частоты:
- зелёным, если AF ≥ 0.015;
- жёлтым, если 0.01 ≤ AF < 0.015;
- красным, если AF < 0.01.
- OMIM ID - идентификатор гена, расположенного на затронутом участке вариации, в базе OMIM. Пишется только для детальных строк.
- Категория - рейтинговая оценка патогенности вариации, предоставляемая AnnotSV и основанная на рекомендациях ACMG и ClinGen1. Согласно рейтинговой оценке выставляется класс ACMG:
- Вариациям с рейтинговой оценкой ≥ 0.99 выставляется класс 5 - патогенные;
- Вариациям с рейтинговой оценкой от 0.90 до 0.98 выставляется класс 4 - вероятно патогенные;
- Вариациям с рейтинговой оценкой от 0.89 до −0.89 выставляется класс 3 - неопределенная значимость;
- Вариациям с рейтинговой оценкой от −0.90 до −0.98 выставляется класс 2 - вероятно доброкачественные;
- Вариациям с рейтинговой оценкой ≤ −0.99 выставляется класс 1 - доброкачественные.
- Класс ACMG - патогенность вариации, выставляемая соответственно категории, определённой
AnnotSV (см. условия выше).
Значения колонки представлены
в виде значков:
- Патогенная;
- Вероятно патогенная;
- Неопределенная значимость;
- Вероятно доброкачественная;
- Доброкачественная.
При наведении курсора на значок можно посмотреть полное значение. - Патогенность - патогенность вариации, определяемая пользователем. Чтобы определить патогенность вариации,
нажмите на кнопку патогенности
в строке вариации и в появившемся диалоге выберите нужную опцию:
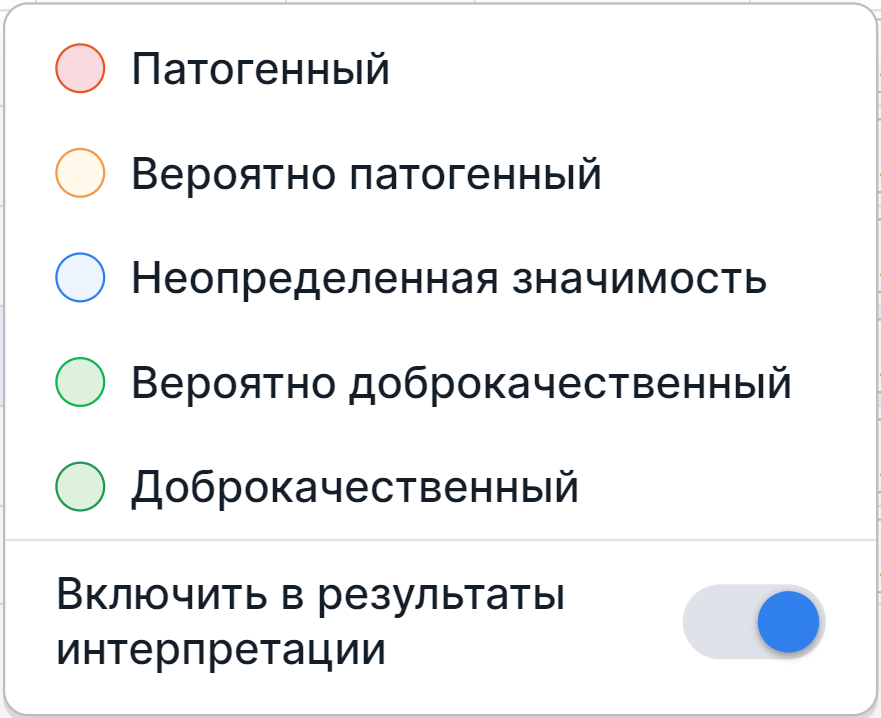
На заметку
Все вариации с определённой патогенностью автоматически добавляются в результаты интерпретации образца. Если вы хотите исключить вариацию с определённой патогенностью из результатов интерпретации, отключите опцию "Включить в результаты интерпретации" в диалоге определения патогенности.
Возможные значения колонки представлены
в виде значков: - Нет интерпретации (до определения патогенности вариации);
- Патогенная;
- Вероятно патогенная;
- Неопределенная значимость;
- Вероятно доброкачественная;
-
Доброкачественная.
При наведении курсора на значок можно посмотреть полное значение.
Патогенность можно переопределить в том же диалоговом окне или очистить, нажав на кнопку патогенности в строке
вариации и в появившемся диалоге выбрав опцию "Очистить":
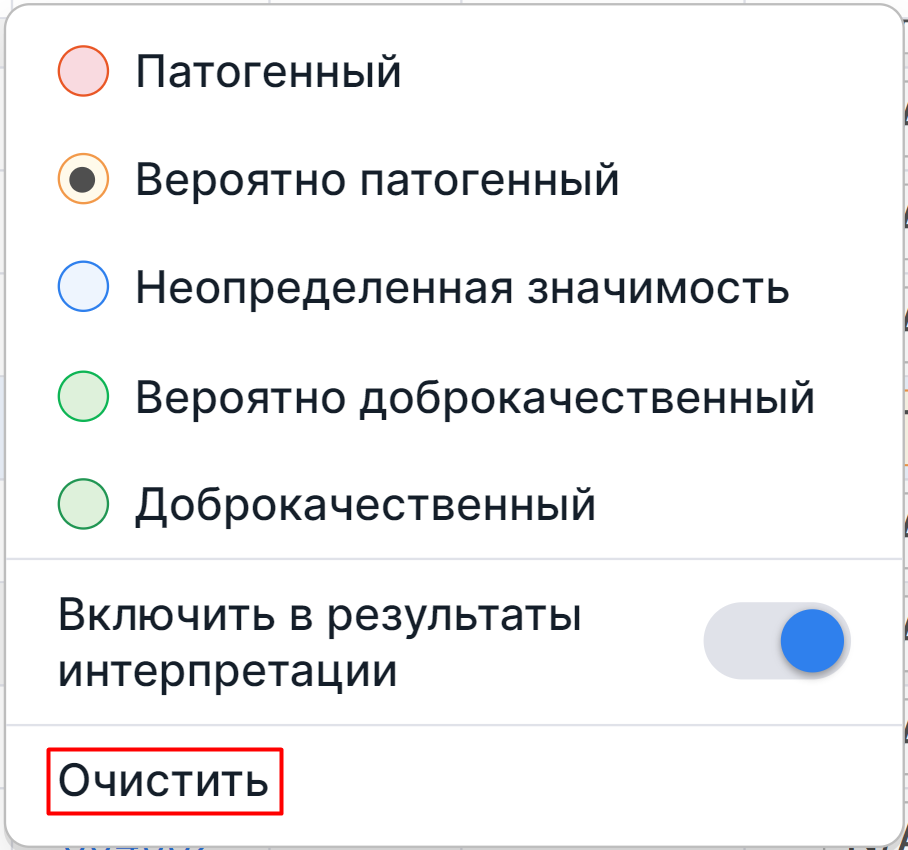
При перезапуске стадии выявления вариации числа копий или более ранних стадий анализа образца
определенный класс патогенности вариации сохраняется, но требует подтверждения.
Кнопка патогенности, которую нужно подтвердить, обозначена
значком .
Если вы согласны с классификацией вариации, нажмите на колокольчик или кнопку патогенности и в появившемся
диалоге выберите опцию "Подтвердить патогенность":
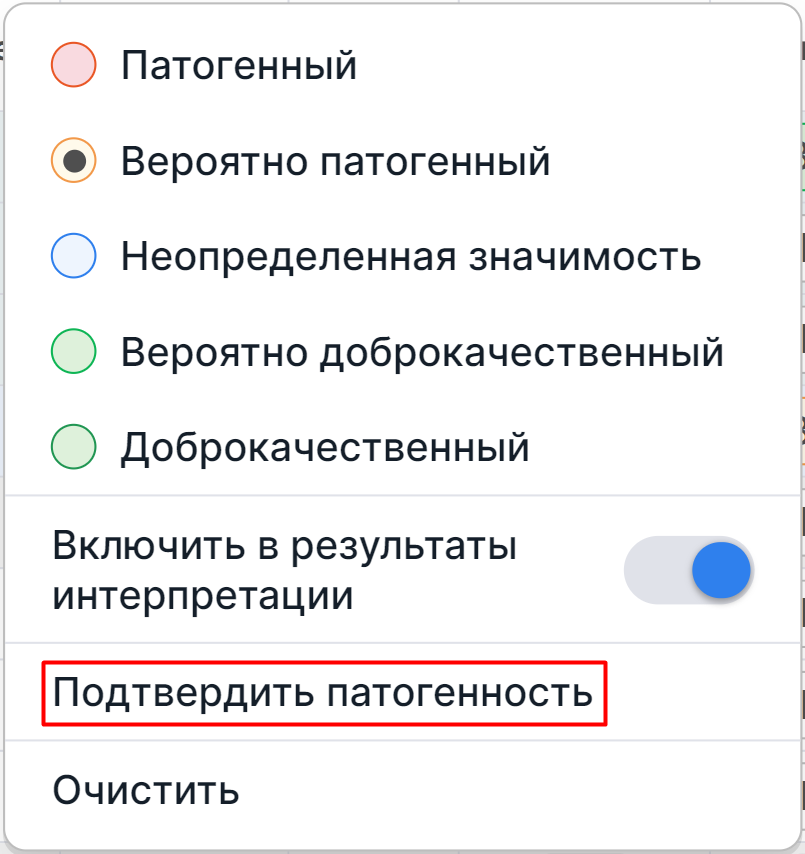
- Ps - патогенные пересекающиеся варианты: установленные патогенные гены или геномные области, полностью перекрывающиеся вариацией. Значения колонки представлены в виде значков:
- Дупликация (Gain);
- Делеция (Loss);
- Инсерция или смешанное значение.
При наведении курсора на значок можно посмотреть полное значение. - Bs - доброкачественные пересекающиеся варианты: установленные доброкачественные геномные области, полностью перекрывающиеся вариацией. Значения колонки представлены в виде значков:
- Дупликация (Gain);
- Делеция (Loss);
- Инверсия;
- Инсерция или смешанное значение.
При наведении курсора на значок можно посмотреть полное значение.
Поиск и сортировка вариаций#
Поиск вариаций в CNV Viewer описан в соответствующем разделе.
По умолчанию таблица отсортирована по позиции вариации. Таблицу можно сортировать по возрастанию или убыванию значений следующих колонок: "Позиция", "Длина, кб", "Гены" (при этом в древовидном режиме сортировка общих строк происходит по количеству генов и сортировка детальных строк - по названию гена, а в режиме плоского списка осуществляется только сортировка общих строк по количеству генов, а сортировка детальных строк остаётся неизменной), "Цитобэнд", "Log2 FC", "Относительная копийность", "Тип", "AF по когорте 180", "Перекрытие с CDS, %", "OMIM ID" (только в древовидном режиме для детальных строк), "Категория", "Класс ACMG", "Патогенность".
Для сортировки таблицы по значениям колонки нажмите на её название (нажмите один раз, чтобы отсортировать
по возрастанию, или два раза, чтобы отсортировать по убыванию). После этого у названия колонки появится
стрелка вверх (при сортировке по возрастанию) или вниз
(при сортировке по убыванию).
Таблицу можно отсортировать по 1-3 колонкам одновременно, чтобы сгруппировать данные с одинаковыми значениями в одной колонке, а затем отсортировать эти группы с одинаковыми значениями по другой колонке. Порядок сортировки будет зависеть от порядка нажатия на названия соответствующих колонок. Стрелочка сортировки будет у той колонки, по которой сортировали последней.
На заметку
Сортировка и фильтрация таблицы сохраняются при выходе со страницы CNV Viewer.
Для сброса сортировки возпользуйтесь кнопкой очистки всех
фильтров: . Обратите внимание, что
при этом сбросится и фильтрация вариаций.
Основным принципом сортировки является сортировка по алфавиту для колонок с текстовыми значениями (например, колонка "Тип") или сортировка от минимального к максимальному и наоборот для колонок с числовыми значениями (например, колонка "Длина, кб"). Однако для некоторых колонок сортировка особенная:
- Колонка "Позиция" - сортировка сначала по хромосоме, затем по позиции на ней.
- Колонки "Класс ACMG" и "Патогенность": сортировка от патогенной к доброкачественной вариации: Патогенная, Вероятно патогенная, Неопределенная значимость, Вероятно доброкачественная, Доброкачественная.
- Riggs E.R., Andersen E.F., Cherry A.M. et al. Technical standards for the interpretation and reporting of constitutional copy number variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics (ACMG) and the Clinical Genome Resource (ClinGen). Genet Med 22, 245-257 (2020)↩