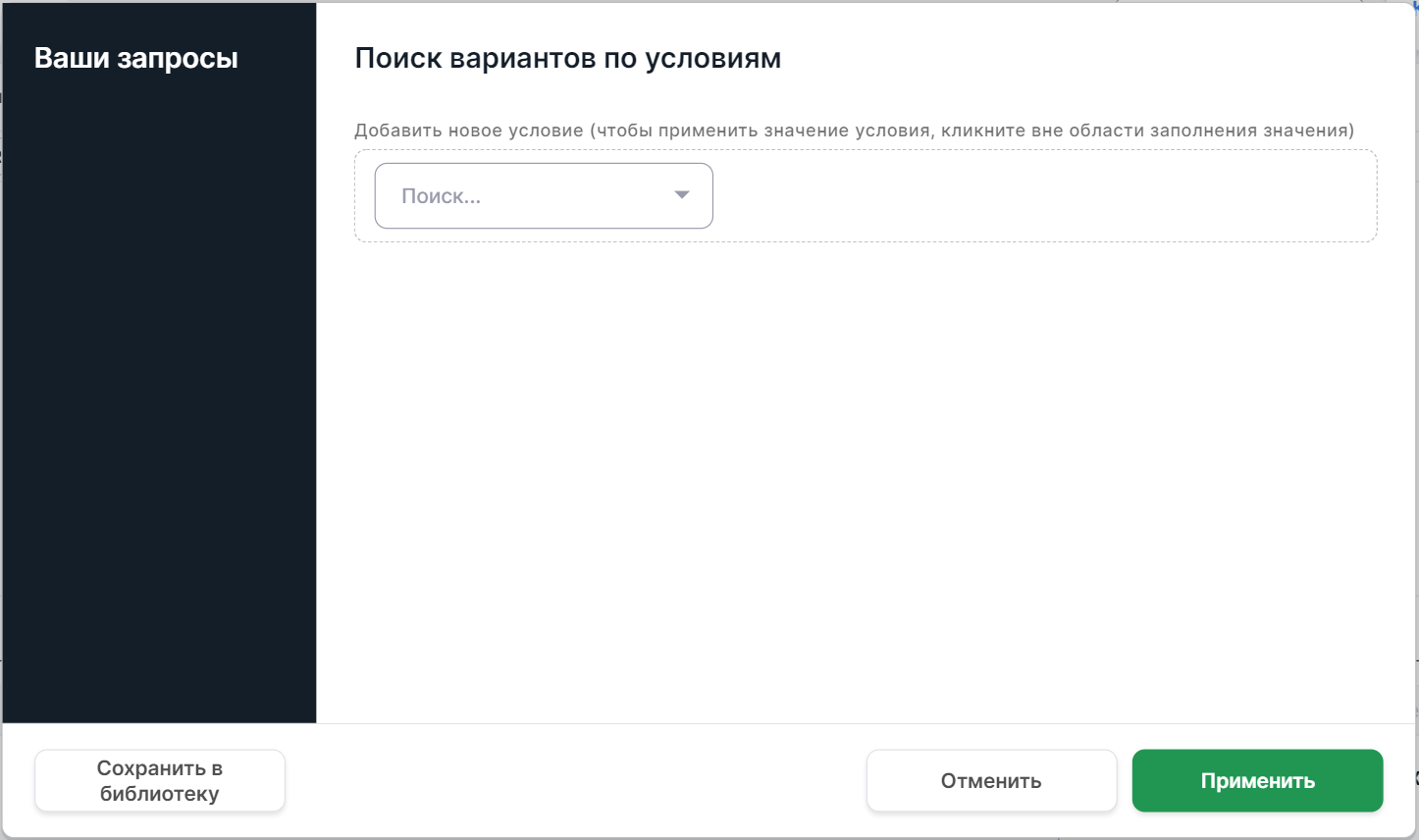
Расширенный поиск вариантов
Чтобы попасть в режим расширенного поиска вариантов, нажмите
на кнопку сверху таблицы.
Вы увидите мастер создания запросов:
На заметку
Если вы находитесь на странице SNV Viewer с соматическими мутациями, то условие фильтрации по соматическим мутациям ("Origin является Somatic") будет добавлено по умолчанию:
На заметку
На странице "Настройки пользователя" можно отрегулировать, в какой режим поиска вы будете попадать, когда впервые открываете страницу SNV Viewer образца: базовый или расширенный.
На заметку
Фильтрация таблицы сохраняется при выходе со страницы SNV Viewer.
Колонки фильтрации#
Колонки разбиты на группы:
- Common
- Per-sample data (если есть данные об образцах секвенирования)
- Gene
- Affected transcripts
- gnomAD 3
- gnomAD 4
- ClinVar
- Conservation
- ExAC
- Protein function effect
- Protein function effect (additional)
- Other frequencies
- Other
- Custom annotation sources (если хотя бы одна пользовательская аннотация была добавлена в систему до того, как варианты в образце были проаннотированы)
С группами колонок можно
взаимодействовать с помощью кнопок и
для
раскрытия и сворачивания списка колонок группы, соответственно.
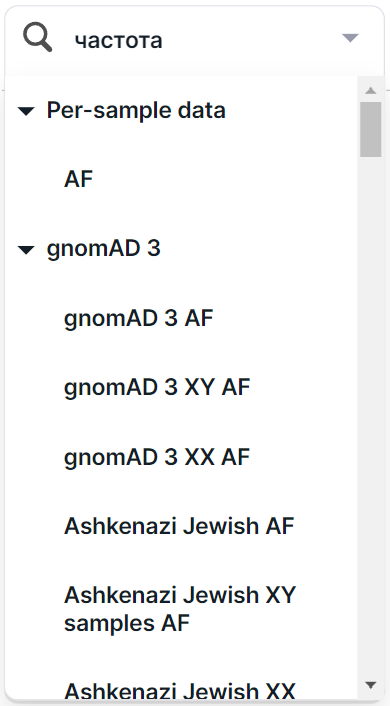
Для поиска нужной колонки фильтрации воспользуйтесь окном поиска. Поиск не зависит от регистра введенных букв. Некоторые колонки можно найти по синонимам поиска. Например, колонки с частотами альтернативного аллеля найдутся по слову "частота":
Common#
- Sample Status (базовый фильтр "Статус образца") - фильтр вариантов по статусу интерпретации образцов запуска: Завершенная интерпретация или Незавершенная интерпретация. Фильтр с выбором одного значения. Доступен только в SNV Viewer запусков.
- Interpretation - фильтр вариантов по интерпретации (комментарию), добавленной пользователем на странице или на панели детальной информации о варианте. Фильтр с вводом любого значения.
- HPO Phenotype - фильтр вариантов по терминам из словаря фенотипических аномалий
человека Human Phenotype Ontology (HPO).
Чтобы открыть форму поиска фенотипа, нажмите на поле
значения
фильтра. Поиск и выбор нужного фенотипа в этой форме описаны здесь.
- Gene panel (базовый фильтр "Панель генов") - фильтр вариантов по локализации в определенных генах из панели. Фильтр с выбором одного значения. Панель генов можно добавить на странице "Панели генов" или импортировать из библиотеки самых распространенных клинических панелей - такая опция доступна в фильтре "Панель генов" в режиме базового поиска. Если у вас нет добавленных генных панелей, список значений фильтра будет пустым.
- Region (базовый фильтр "Регион") - фильтр вариантов по
позиции на геноме (хромосома, стартовая и конечная позиции). Для фильтрации по региону нажмите на окно
ввода
 и введите позицию искомого
геномного интервала с указанием хромосомы, стартовой и конечной позиции. Возможные варианты записи позиции интервала:
и введите позицию искомого
геномного интервала с указанием хромосомы, стартовой и конечной позиции. Возможные варианты записи позиции интервала:
chr1:161,190,824-161,193,921chr1:161190824-161193921chr1:161190824_1611939211:161190824-161193921
Также можно фильтровать сразу по более, чем одному интервалу. Например,
по chr1:161190824-161193921, chrX:151110769-151113866.
Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
В результате фильтрации по региону будут найдены те варианты, чьи позиции пересекаются с введённым интервалом.
- Gene (колонка "Локус", базовый фильтр "Ген") - фильтр вариантов по локализации в транскрипте гена. Значения фильтра зависят от выбранного оператора фильтрации:
- любое значение, если выбраны операторы "начинается с", "содержит", "не содержит". Например, это полезно, если вы хотите отфильтровать варианты по генам, содержащим определённую последовательность букв в названии (к примеру, "HLA-").
- мультиселект, если выбраны операторы "является", "не является". Для фильтрации вариантов по локализации в определённых генах. В опциях значений транскрипт гена приведён в скобках рядом с официальным названием гена.
- без значения, если выбраны операторы "не пусто", "пусто".
- Impact (колонка "Значение эффекта") - фильтр по предсказанному воздействию варианта на белок: High, Moderate, Low, Modifier. Значения колонки описаны здесь. Фильтр с мультиселектом.
- Genome position - фильтр по позиции варианта в геноме: в экзоне (Exonic), в интроне (Intronic), в межгенной области (Intergenic). Фильтр с мультиселектом.
- Consequence (колонка "Эффект варианта", базовый фильтр "Эффект") - фильтр по эффекту вариантов на гены. Значения колонки описаны здесь. Фильтр с мультиселектом.
- Pathogenicity (колонка "Патогенность", базовый фильтр "Патогенность") - фильтр с мультиселектом по патогенности варианта, определенной пользователем самостоятельно, по критериям ACMG или по базе патогенности:
- Pathogenic - Патогенный;
- Likely pathogenic - Вероятно патогенный;
- Uncertain significance conflict - Конфликт: противоречивые интерпретации патогенности;
- Uncertain significance - Неопределенная значимость;
- Likely benign - Вероятно доброкачественный;
- Benign - Доброкачественный;
- Undefined - Нет интерпретации.
- Label (колонка "Метка", базовый фильтр "Метка") - фильтр с мультиселектом по значимости вариантов, определенной пользователем:
- High significance - Высокая значимость;
- Moderate significance - Средняя значимость;
- Low significance - Низкая значимость;
- Sequencing error - Ошибка секвенирования;
- Undefined - Неопределенная значимость.
- Phasing group (колонка "ФГ") - фильтр вариантов с мультиселектом по статусу фазовой группы:
- Phasing group - фазовая группа (объединение гомозиготных вариантов, расположенных в пределах определенного геномного интервала);
- Part of group consequence equal - часть фазовой группы с совпадающей аминокислотной заменой;
- Part of group consequence unequal - часть фазовой группы с несовпадающей аминокислотной заменой.
- Compound heterozygote (Genotype) - фильтр вариантов по тому, входят ли они в компаунд-гетерозиготы (т.е. гетерозиготность по двум или более вариантам одного гена; подробно все случаи описаны здесь). В отличие от режима фильтрации по компаунд-гетерозиготе, этот фильтр применяется одновременно с остальными условиями фильтрации, т.е. в результате найдутся варианты, которые входят в компаунд-гетерозиготу, но остальные варианты из этой компаунд-гетерозиготы могут отфильтроваться по другим условиям.
В SNV Viewer семейного анализа и в SNV Viewer с одним образцом секвенирования для фильтрации вариантов по компаунд-гетерозиготе достаточно выбрать сам фильтр в списке колонок фильтрации:
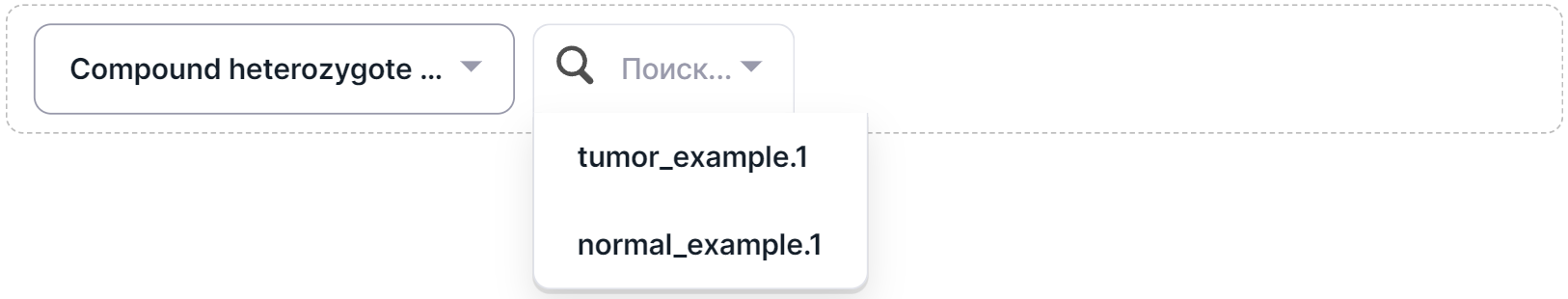
В SNV Viewer группового анализа герминальной когорты, в SNV Viewer с несколькими образцами секвенирования и в SNV Viewer запуска (если есть данные об образцах секвенирования) для фильтрации вариантов по компаунд-гетерозиготе нужно выбрать образец, в котором будут найдены все случаи, когда хотя бы две гетерозиготы (0/1, 1/0, 0|1 и т.п.) расположены в одном гене:
- Trio GT (Genotype) - фильтр вариантов по наследованию с мультиселектом: De novo варианты, Новые гомозиготы (гомозиготы по локусам у пробанда, которые гетерозиготны у родителей) или Наследованные от одного родителя. Доступен только в семейном групповом анализе в случае Трио (Пробанд, Отец и Мать).
- Chromosome (колонка "Позиция") - фильтр вариантов по локализации в хромосомах: chr1-chr22, chrX, chrY, chrM, хромосомы вида chrN_random (контиги, расположенные в хромосоме N, которые невозможно с уверенностью разместить на этой хромосоме, или последовательности альтернативных гаплотипов), хромосомы вида chrUn_ (контиги, которые нельзя с уверенностью разместить на конкретной хромосоме), chrEBV. Фильтр с мультиселектом.
- rsId (колонка "Внешние ссылки") - фильтр вариантов по идентификатору в базе dbSNP. Фильтр с вводом идентификатора. В качестве значения можно использовать как полный идентификатор в базе (например, rs1126497), так и цифры идентификатора отдельно (1126497).
- ClinVar ID (колонка "Внешние ссылки") - фильтр вариантов по идентификатору (Variation ID) в базе ClinVar (например, 183700). Фильтр с вводом идентификатора.
- Origin (колонка "Тип мутации", базовый фильтр "Тип мутации") - фильтр вариантов по типу мутации: соматическая (Somatic), герминальная (Germline). Фильтр с мультиселектом.
- Start (колонка "Позиция") - фильтр по начальной позиции варианта на хромосоме. Фильтр с вводом числового значения (натуральное число).
- End - фильтр по конечной позиции варианта на хромосоме. Фильтр с вводом числового значения (натуральное число).
- Reference allele (колонка "Замена") - фильтр по референсному аллелю варианта. Фильтр с вводом любого значения.
- Alternative allele (колонка "Замена") - фильтр по альтернативному аллелю варианта. Фильтр с вводом любого значения.
- HGVSc - фильтр вариантов по нуклеотидной замене в номенклатуре HGVS: префикс “c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности) + геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель. Фильтр с вводом любого значения.
- HGVSp (short) (колонка "HGVSp") - фильтр вариантов по аминокислотной замене в номенклатуре HGVS с однобуквенными обозначениями аминокислот (префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены). Фильтр с вводом любого значения. Пример: p.P72R.
- HGVSp (long) (колонка "HGVSp") - фильтр вариантов по аминокислотной замене в номенклатуре HGVS с трехбуквенными обозначениями аминокислот. Записывается с учетом транскрипта: идентификатор транскрипта из Ensembl + префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены. Фильтр с вводом любого значения. Пример: ENSP00000269305.4:p.Pro72Arg.
- Exon (колонка "Экзон") - фильтр по номеру экзона в транскрипте, который вариант затронул. Фильтр с вводом числового значения (натуральное число).
- Onco Relevance (колонка "Онкозначимость") - фильтр по значимости варианта для развития онкологических заболеваний: Relevant, Irrelevant. Фильтр с выбором одного значения.
- Caller Filters (колонка "Фильтры детекции", базовый фильтр "Фильтры") - фильтр вариантов по качеству с мультиселектом.
Нажмите, чтобы посмотреть определения фильтров детекции
| Значение фильтра детекции | Тип мутации | Инструмент GATK | Описание фильтра |
| alignment | Соматическая | FilterMutectCalls | Фильтр артефактов выравнивания |
| base_qual | Соматическая | FilterMutectCalls | Среднее качество оснований прочтений, поддерживающих альтернативный аллель |
| clustered_events | Соматическая | FilterMutectCalls | Кластерные события, наблюдаемые в опухоли, - соматические мутации, которые совместно встречаются в одном и том же интервале сборки |
| CNN_2D | Герминальная | CNNScoreVariants | Фильтрация на основе оценок предварительно обученной сверточной нейронной сети (convolutional neural network; CNN) 2D |
| contamination | Соматическая | FilterMutectCalls | Фильтр контаминации данных секвенирования |
| duplicate | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля чрезмерно представлены очевидными дубликатами |
| FAIL | Соматическая | FilterMutectCalls | Ошибка сайта, если все аллели не проходят по разным причинам |
| fragment | Соматическая | FilterMutectCalls | Разница медианных длин фрагментов прочтений, поддерживающих альтернативный и референсный аллели |
| FS | Герминальная | FisherStrand | Вероятность смещения цепей на сайте, оцененная с помощью точного критерия Фишера. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. FisherStrand определяет, существует ли смещение цепи между прямой и обратной цепями для референсного или альтернативного аллеля (то есть встречается ли альтернативный аллель чаще или реже на прямой или обратной цепи, чем референсный аллель). FisherStrand хуже учитывает большие объемы данных в ситуациях с высоким покрытием по сравнению с StrandOddsRatio. Кроме того, FisherStrand имеет тенденцию "штрафовать" варианты, которые встречаются на концах экзонов. Варианты на концах экзонов, как правило, покрываются прочтениями только в одном направлении цепи, и FisherStrand дает таким вариантам плохую оценку, тогда как StrandOddsRatio учитывает соотношение прочтений, охватывающих оба аллеля. |
| germline | Соматическая | FilterMutectCalls | Доказательства указывают на то, что этот сайт является герминальным, а не соматическим |
| haplotype | Соматическая | FilterMutectCalls | Вариант расположен рядом с отфильтрованным вариантом того же гаплотипа |
| low_allele_frac | Соматическая | FilterMutectCalls | Доля аллеля ниже установленного порога |
| LowQual | Соматическая | FilterMutectCalls | Низкое качество |
| map_qual | Соматическая | FilterMutectCalls | Среднее качество картирования прочтений, поддерживающих альтернативный аллель |
| MQ | Герминальная | RMMSMapping- Quality | Среднеквадратичное значение качества сопоставления прочтений по всем образцам. Представляет собой оценку общего качества картирования прочтений, покрывающих сайт варианта, усредненную по всем образцам в когорте, т.е. квадратный корень среднего квадрата качества картирования сайта варианта. Таким образом, оценка учитывает стандартное отклонение качества картирования. |
| MQ_RANK_SUM | Герминальная | MappingQuality- RankSumTest | Тест суммы рангов для сопоставления качества картирования прочтения REF и качества картирования прочтения ALT. MQRankSum на уровне варианта сравнивает качество картирования прочтений, поддерживающих референсный аллель, с прочтениями, поддерживающими альтернативный аллель. Обнаружение любой статистически значимой разницы в качестве предполагает, что процесс секвенирования и/или картирования мог быть искажен артефактом. |
| mt_many_low_hets | Соматическая | FilterMutectCalls | Фильтр сайтов с низкой гетероплазмией |
| multiallelic | Соматическая | FilterMutectCalls | Мутация располагается в мультиаллельном сайте: слишком много альтернативных аллелей удовлетворяют LOD score (вероятный признак артефакта) |
| n_ratio | Соматическая | FilterMutectCalls | Отношение количества неизвестных нуклеотидов N к количеству альтернативных аллелей превышает установленный порог |
| normal_artifact | Соматическая | FilterMutectCalls | Нормальный артефакт: вариант, наблюдаемый в опухоли, также присутствует как артефакт в контрольном образце |
| Not available | Любая | Значение колонки неизвестно (точка в VCF-файле) | |
| numt_chimera | Соматическая | FilterMutectCalls | Вариант, расположенный в ядерной копии гена мтДНК (NuMT), со слишком большим количеством прочтений, поддерживающих альтернативный аллель, происходящих из аутосомы |
| numt_novel | Соматическая | FilterMutectCalls | Глубина альтернативного аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| orientation | Соматическая | FilterMutectCalls | Фильтр смещения ориентации - смещения, возникающего из-за химического изменения нуклеотида во время подготовки библиотеки, которое приводит к неправильному спариванию основанию (G с A, T c C и т.д.) (артефакт секвенирования) |
| OTHER | Любая | Значение колонки не распознано | |
| panel_of_normals | Соматическая | FilterMutectCalls | Сайт находится в "черном списке" нормальной панели (PON). Как выбрать панель, описано здесь. |
| PASS | Любая | Сайт содержит минимум один аллель, который удовлетворяет всем фильтрам | |
| position | Соматическая | FilterMutectCalls | Медианное расстояние альтернативных мутаций от конца прочтения |
| possible_numt | Соматическая | FilterMutectCalls | Глубина аллеля ниже ожидаемого покрытия ядерной копии гена мтДНК (NuMT) в аутосоме |
| QD | Герминальная | QualByDepth | Достоверность варианта (значение из поля QUAL), нормализованная по нефильтрованнной глубине аллеля (AD) варианта в выборке вариантов с генотипом, отличным от hom-ref (homozygous/reference; генотип, при котором оба аллеля являются референсными) |
| QUAL | Герминальная | Качество варианта - вероятность существования (по шкале Phred) варианта в этом сайте с учетом данных секвенирования | |
| READ_POS_RANK_SUM | Герминальная | ReadPosRank- SumTest | Тест суммы рангов для относительных позиций аллелей REF и ALT в прочтениях. ReadPosRankSum на уровне варианта проверяет, различаются ли положения референсного и альтернативного аллеля в прочтениях, которые поддерживают вариант. Нахождение аллеля только вблизи концов прочтений указывает на ошибку, потому что именно там секвенаторы склонны делать больше всего ошибок. Обнаружение статистически значимой разницы в относительном положении в любом случае указывает на то, что процесс секвенирования мог быть искажен артефактом. |
| SITE | Соматическая | FilterMutectCalls | Ошибка сайта |
| slippage | Соматическая | FilterMutectCalls | Фильтр ошибок проскальзывания полимеразы в ходе ДНК-репликации (предсказывается по количеству и длине коротких тандемных повторов (STR)) |
| SOR | Герминальная | StrandOddsRatio | Смещение цепей, оцененное с помощью симметричного теста отношения шансов. Смещение цепей - это тип смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим. StrandOddsRatio оценивает наличие смещения между прямыми и обратными цепями для референсного или альтернативного аллеля (аллелей). |
| strand_bias | Соматическая | FilterMutectCalls | Фильтр смещения цепей - смещения последовательности, при котором предпочтение отдается одной цепи ДНК перед другой, что может привести к неправильной оценке количества доказательств, наблюдаемых для одного аллеля по сравнению с другим (артефакт секвенирования) |
| strict_strand | Соматическая | FilterMutectCalls | Доказательства наличия альтернативного аллеля не представлены в обоих направлениях прочтения |
| weak_evidence | Соматическая | FilterMutectCalls | Мутация не соответствует порогу вероятности |
- Occurrence in Run - фильтр по встречаемости варианта среди образцов запуска. Фильтр с вводом числового значения (целое число). Доступен только в SNV Viewer запусков и образцов запуска.
- Cosmic ID - фильтр по идентификатору варианта в базе COSMIC (например, COSV52665487). Фильтр с вводом идентификатора. Доступен, только если база COSMIC была загружена в виде пользовательской аннотации до завершения аннотации вариантов образца.
- RUSeq AF - фильтр по частоте альтернативного аллеля варианта из базы RUSeq. Фильтр с вводом числового значения (любое число). Доступен, только если база RUSeq была загружена в виде пользовательской аннотации до завершения аннотации вариантов образца.
Per-sample data#
- Группа колонок фильтрации представлена, только если есть данные об образцах секвенирования.
- Genotype (колонка "GT") - фильтр
вариантов по генотипу - значениям референсного и альтернативного аллелей для образца.
При фильтрации данные о фазировании генотипа не учитываются. Чтобы добавить условие фильтрации вариантов по генотипу,
сделайте следующее:
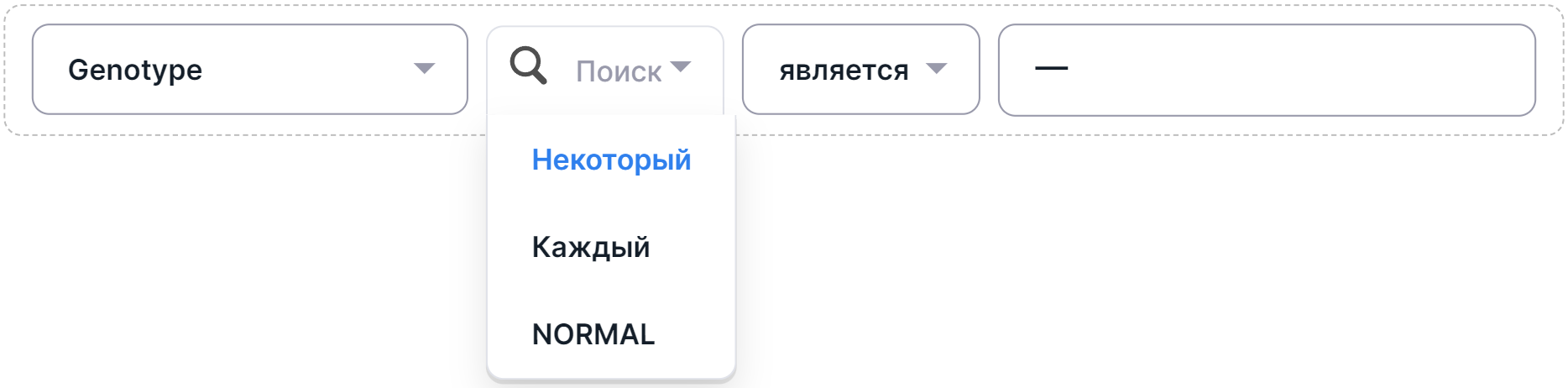
1. Выберите образец для фильтрации:
- "Некоторый" - любой образец имеет искомый генотип;
- "Каждый" - каждый образец имеет искомый генотип;
- Название образца.
- Выберите оператор фильтрации:
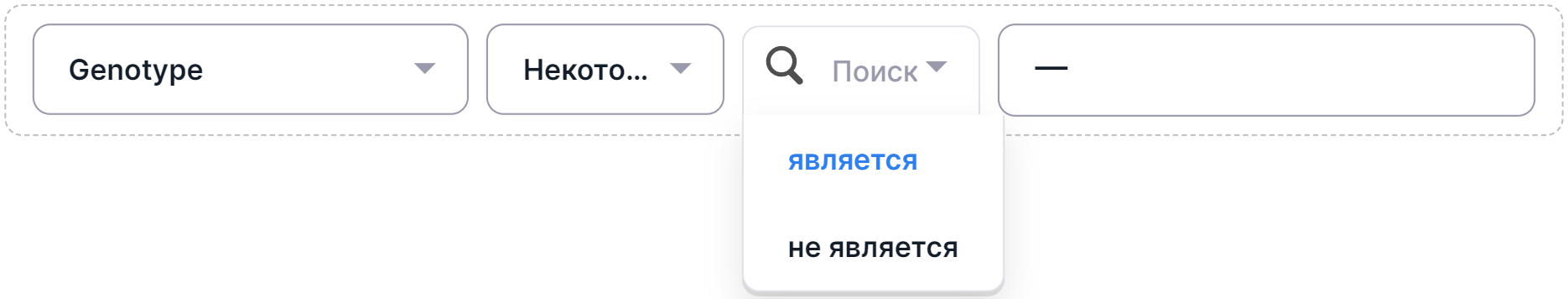
- Выберите необходимое значение фильтра, нажав на строку значения:
- "REF" - сайт содержит только референсные аллели (например,
0/0,0|0,0/0/0); - "ALT" - сайт содержит хотя бы один альтернативный аллель (например,
1/1,1|1,0/1,1/0,0|1,1|0,1/.,1|.,./1,.|1,1,0/0/1,0/2,1/2); - "HOM" - гомозиготный сайт, который содержит либо референсные, либо альтернативные аллели (например,
1|1,1/1,1/1/1,0/0,0|0); - "HET" - гетерозиготный сайт, который содержит минимум два разных аллеля (например,
0/1,1/0,0|1,1|0,2/0,0/1/2); - "HOM_ALT" - гомозиготный сайт, который содержит только альтернативные аллели (например,
1/1,1|1|1); - "MIS" - нет информации о сайте (например,
./.).
- "Read depth" (колонка "Глубина (Альт/Реф)") - фильтр вариантов по глубине секвенирования - общему количеству прочтений последовательности, перекрывающих позицию варианта, для этого образца.
- "Ref count" (Реф) - фильтр вариантов по количеству раз, когда в последовательности считывался референсный нуклеотид для данного образца.
- "Alt count" (Альт) - фильтр вариантов по количеству раз, когда в последовательности считывался альтернативный нуклеотид для данного образца.
- "AF" - фильтр вариантов по частоте альтернативного аллеля для данного образца.
Чтобы добавить условие фильтрации вариантов по глубине, реф, альт или AF, сделайте следующее:
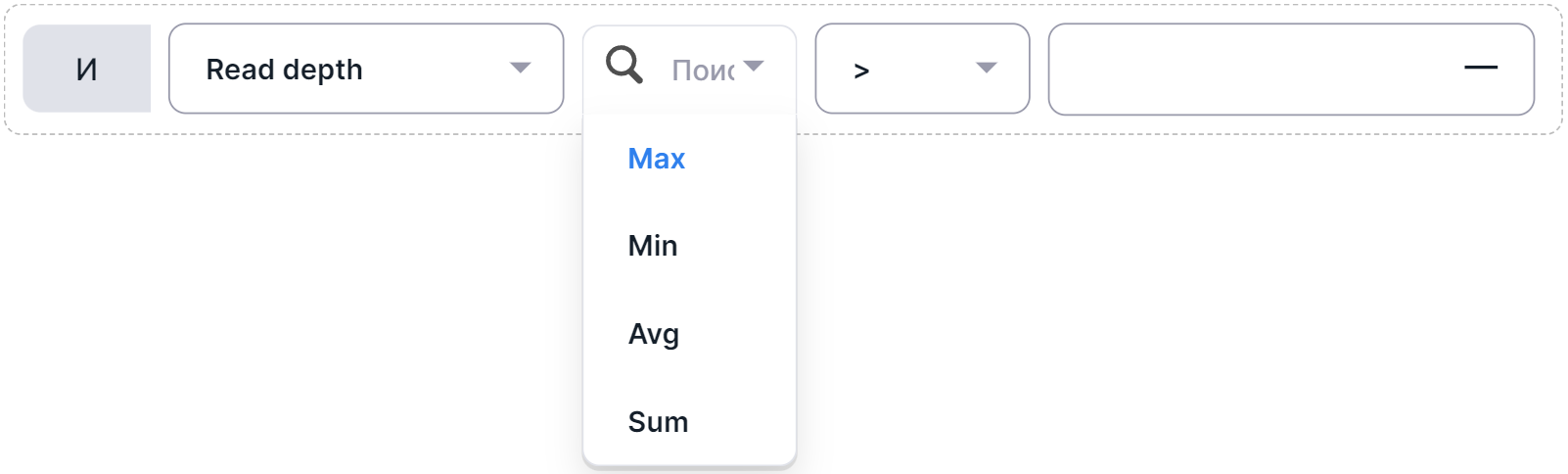
- Выберите функцию фильтра:
- "Max" - максимальное значение;
- "Min" - минимальное значение;
- "Avg" - среднее значение;
- "Sum" - суммарное значение.
- Выберите оператор фильтрации:
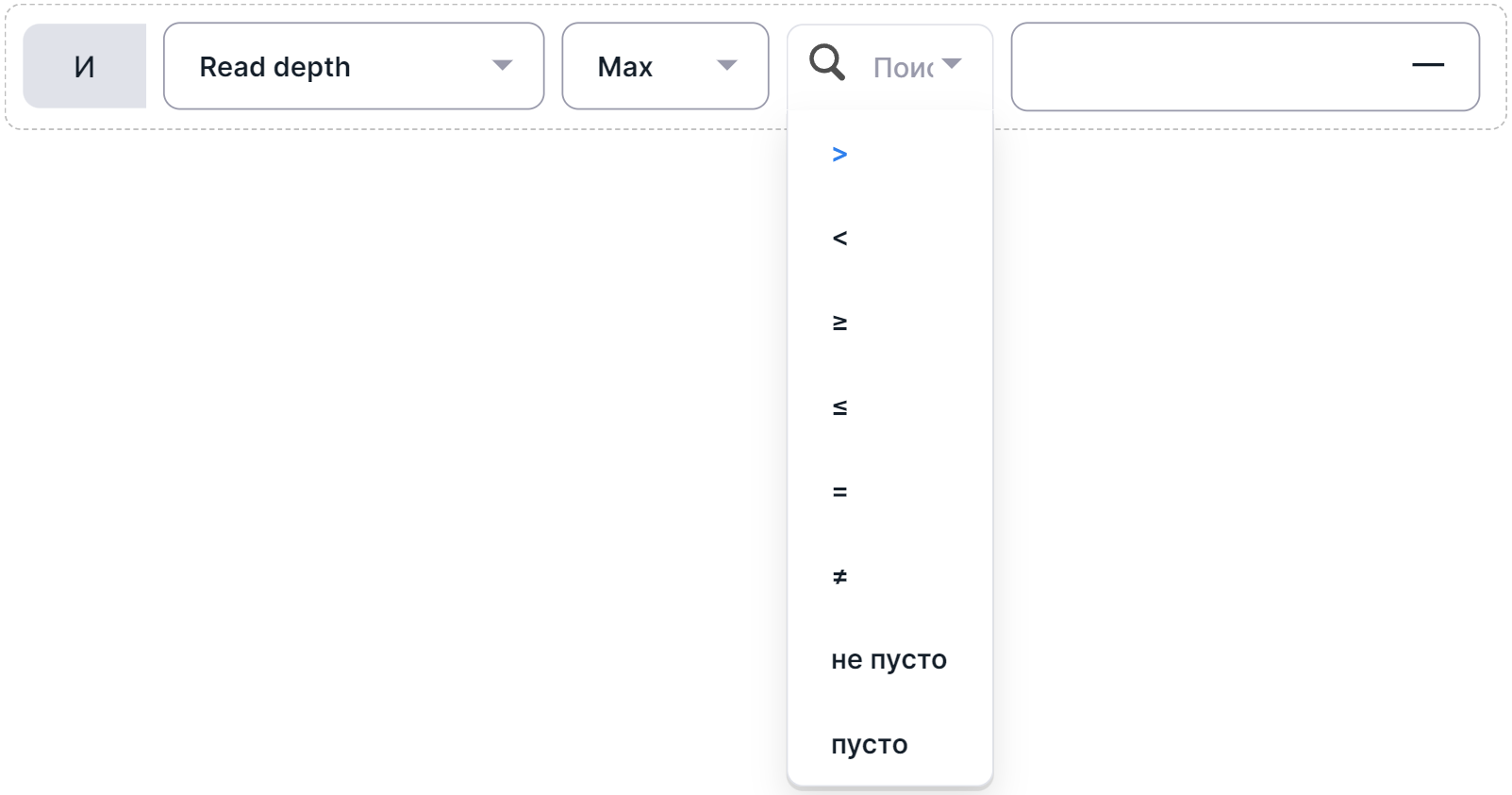
- Если в качестве оператора фильтрации был выбран математический оператор, нажмите на
окно
вводаи введите пороговое значения фильтра (натуральное число для фильтров по глубине, реф и альт и положительное рациональное число для AF). Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
Gene#
- RefSeq Transcript - фильтр вариантов по идентификатору транскрипта в базе RefSeq (например, NM_002354.3). Фильтр с вводом любого значения.
- OMIM ID - фильтр вариантов по идентификатору гена в базе OMIM (например, 185535). Фильтр с вводом идентификатора.
- OMIM Phenotypes - фильтр по значениям колонок таблицы с фенотипами OMIM, связанными с геном, в котором расположен вариант. Фильтр с вводом любого значения. Доступен, только если у вас есть доступ к данным базы OMIM внутри Genomenal, который предоставляется при наличии у пользователя лицензии на доступ к базе.
- NCBI ID - фильтр вариантов по идентификатору гена в базе NCBI Gene (например, 4072). Фильтр с вводом идентификатора.
- Swiss-Prot ID - фильтр вариантов по идентификатору гена в базе UniProtKB Swiss-Prot (например, P16422). Фильтр с вводом идентификатора.
- TrEMBL ID - фильтр вариантов по идентификатору гена в базе UniProtKB TrEMBL (например, B5MCA4). Фильтр с вводом идентификатора.
- Ensembl ID - фильтр вариантов по идентификатору гена в базе Ensembl (например, ENSG00000119888). Фильтр с вводом идентификатора.
- pLI - фильтр вариантов по pLI (Probability of Loss-of-Function Intolerance) - вероятности того, что транскрипт чувствителен к вариантам потери функции (Lof; Loss of Function). Диапазон значений: от 0 до 1. Гены с pLI >= 0.9 считаются непереносимыми к вариантам потери функции. Фильтр с вводом числового значения (положительное рациональное число).
- LOEUF - фильтр вариантов по LOEUF (Loss-of-function Observed/Expected Upper bound Fraction) - верхней границе 90% доверительного интервала отношения наблюдаемых к ожидаемым высоконадёжным вариантам потери функции (pLoF). Более низкие значения (LOEUF < 0.35) указывают на более высокую степень генетического ограничения к потерям функции. Фильтр с вводом числового значения (положительное рациональное число).
- Missense O/E ratio - фильтр вариантов по верхней границе 90% доверительного интервала отношения наблюдаемых к ожидаемым миссенс-вариантам. Чем ниже значение, тем больше ген ограничен в отношении миссенс-вариантов. Фильтр с вводом числового значения (положительное рациональное число).
- Missense Z-score - фильтр вариантов по Z-оценке для миссенс-вариантов в транскрипте. Z-оценка отражает, насколько число наблюдаемых миссенс-мутаций в данном гене отличается от ожидаемого количества, если бы мутации распределялись случайным образом по геному. Более высокие положительные значения Z-оценки (≥3.09) указывают на повышенную чувствительность гена к аминокислотным заменам, то есть на большую степень генетического ограничения к миссенс-вариантам. Более низкие или отрицательные значения Z-оценки, наоборот, указывают на то, что в гене встречается больше миссенс-мутаций, чем ожидалось, а значит, он терпим к таким изменениям. Фильтр с вводом числового значения (рациональное число).
Affected transcripts#
Группа включает фильтр Is MANE transcript, позволяющий отфильтровать варианты в зависимости от того, затрагивают ли они основной транскрипт гена согласно MANE Select, MANE plus clinical или Ensembl canonical.
Фильтр реализован в виде переключателя:
- Включите переключатель, чтобы отображались только варианты, затрагивающие основной транскрипт гена:
- Выключите переключатель, чтобы отображались только варианты, не затрагивающие основной транскрипт гена:
gnomAD 3#
Колонки фильтрации со следующими данными из базы gnomAD (версия 3):
- AF (колонка "gnomAD 3 AF") - фильтр вариантов по суммарной частоте альтернативного аллеля. Фильтр с вводом числового значения (положительное рациональное число).
- AC - фильтр вариантов по количеству альтернативного аллеля в генотипах высокого качества. Фильтр с вводом числового значения (натуральное число).
- AN - фильтр вариантов по общему количеству генотипов высокого качества. Фильтр с вводом числового значения (натуральное число).
- XY AF - фильтр вариантов по частоте аллеля в подпопуляции XY. Фильтр с вводом числового значения (положительное рациональное число).
- XX AF - фильтр вариантов по частоте аллеля в подпопуляции XX. Фильтр с вводом числового значения (положительное рациональное число).
- Number of Homozygotes - фильтр вариантов по количеству гомозигот. Фильтр с вводом числового значения (натуральное число).
- [Популяция] AF - фильтр вариантов по суммарной частоте аллеля в различных популяциях, где [Популяция]: Ashkenazi Jewish, Amish, African and Afr. Am., Finnish, South Asian, Latino, Non-finnish european, East Asian, Middle Eastern, Other. Фильтр с вводом числового значения (положительное рациональное число).
- [Популяция] XY/XX samples AF - фильтр вариантов по частотам в подпопуляциях XY или XX в различных популяциях, где [Популяция]: Ashkenazi Jewish, Amish, African and African American, Finnish, South Asian, Latino, Non-finnish european, East Asian, Middle Eastern, Other. Фильтр с вводом числового значения (положительное рациональное число).
- [Популяция] Number of Homozygotes - фильтр вариантов по количеству гомозигот в различных популяциях, где [Популяция]: Ashkenazi Jewish, Amish, African and African American, Finnish, South Asian, Latino, Non-finnish european, East Asian, Middle Eastern, Other. Фильтр с вводом числового значения (положительное рациональное число).
- Coverage - фильтр вариантов по средней глубине покрытия по основаниям: <10, 10-100, ⩾100. Фильтр с вводом любого значения.
gnomAD 4#
Колонки фильтрации со следующими данными из базы gnomAD (версия 4):
- AF - фильтр вариантов по суммарной частоте альтернативного аллеля в генотипах высокого качества в экзомных и геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- AC - фильтр вариантов по количеству альтернативного аллеля в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- AN - фильтр вариантов по общему количеству генотипов высокого качества в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- Number of Homozygotes - фильтр вариантов по количеству людей, гомозиготных по альтернативному аллелю, в популяции в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XY Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XY в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XX Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XX в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XY AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XY в экзомных и геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- XY AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XY в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XY AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XY в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XX AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XX в экзомных и геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- XX AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XX в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- XX AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XX в экзомных и геномных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes AF - фильтр вариантов по суммарной частоте альтернативного аллеля в генотипах высокого качества в экзомных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Exomes AC - фильтр вариантов по количеству альтернативного аллеля в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes AN - фильтр вариантов по общему количеству генотипов высокого качества в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes Number of Homozygotes - фильтр вариантов по количеству людей, гомозиготных по альтернативному аллелю, в популяции в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XY Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XY в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XX Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XX в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XY AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XY в экзомных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Exomes XY AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XY в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XY AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XY в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XX AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XX в экзомных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Exomes XX AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XX в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Exomes XX AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XX в экзомных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes AF - фильтр вариантов по суммарной частоте альтернативного аллеля в генотипах высокого качества в геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Genomes AC - фильтр вариантов по количеству альтернативного аллеля в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes AN - фильтр вариантов по общему количеству генотипов высокого качества в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes Number of Homozygotes - фильтр вариантов по количеству людей, гомозиготных по альтернативному аллелю, в популяции в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XY Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XY в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XX Number of Homozygotes - фильтр вариантов по количество людей, гомозиготных по альтернативному аллелю, в подпопуляции XX в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XY AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XY в геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Genomes XY AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XY в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XY AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XY в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XX AF - фильтр вариантов по суммарной частоте альтернативного аллеля в подпопуляции XX в геномных данных. Фильтр с вводом числового значения (положительное рациональное число).
- Genomes XX AC - фильтр вариантов по количеству альтернативного аллеля в подпопуляции XX в геномных данных. Фильтр с вводом числового значения (натуральное число).
- Genomes XX AN - фильтр вариантов по общему количеству генотипов высокого качества в подпопуляции XX в геномных данных. Фильтр с вводом числового значения (натуральное число).
ClinVar#
Колонки фильтрации со следующими данными из базы ClinVar:
- Clinical significance (колонка "ClinVar", базовый фильтр "ClinVar") - фильтр вариантов по клинической значимости (определение значений можно посмотреть тут). Фильтр с мультиселектом.
- Allele ID - фильтр вариантов по идентификатору аллельной замены (например, 405185). Фильтр с вводом идентификатора.
- Phenotypes - фильтр вариантов по фенотипической значимости с вводом любого значения.
- Phenotypes cross references - фильтр вариантов по ссылкам на фенотип в различных базах данных (Orphanet, MedGen, MONDO, OMIM и др.) Фильтр с вводом любого значения.
- Haplotype phenotypes - фильтр по фенотипической значимости варианта, являющегося частью гаплотипа. Фильтр с вводом любого значения.
- Haplotype phenotypes cross references - фильтр по ссылкам на фенотип варианта, являющегося частью гаплотипа, в различных базах данных (Orphanet, MedGen, MONDO, OMIM и др.) Фильтр с вводом любого значения.
- Review status - фильтр вариантов по суммарной оценке источника данных, в котором заявлена клиническая значимость фенотипа. Фильтр с вводом любого значения. Возможные варианты значений и их определения можно посмотреть тут.
- Cross references - фильтр вариантов по ссылкам в различных базах данных (ClinGen, UniProtKB, OMIM, GTR и др.) Фильтр с вводом любого значения.
- Allele origin - фильтр вариантов по происхождению аллеля с мультиселектом:
- Unknown - варианты неизвестного происхождения;
- Germline - варианты герминального происхождения;
- Somatic - варианты соматического происхождения;
- Inherited - варианты, которые наследуются;
- Paternal - варианты, унаследованные от отца;
- Maternal - варианты, унаследованные от матери;
- De novo варианты - варианты, возникшие de novo;
- Biparental - варианты, унаследованные от обоих родителей;
- Tested inconclusive - варианты, для которых нет результатов проверки происхождения;
- Other - варианты с другим типом происхождения.
- Variant Haplotype ID - фильтр по идентификатору варианта (Variation ID), являющегося частью гаплотипа. Фильтр с вводом идентификатора.
Conservation#
- Ancestral allele - фильтр вариантов по предковому аллелю, предсказанному EPO. Фильтр с вводом любого значения.
- Altai Neanderthal - фильтр вариантов по генотипу Altai Neanderthal с вводом любого значения.
- Denisova - фильтр вариантов по генотипу Denisova с вводом любого значения.
- Vindija Neanderthal - фильтр вариантов по генотипу Vindija Neanderthal с вводом любого значения.
- [Программа] [Оценка] - фильтр вариантов по оценкам консервативности, полученным с помощью предсказания различными программами, где [Программа] - программа предсказания: FitCons, GERP++, PhyloP, PhastCons, SiPhy, BStatistic, и [Оценка] - оценка консервативности: rankscore, score, neutral rate. Фильтр с вводом числового значения (положительное рациональное число).
- [Оценка] confidence value - фильтр вариантов по значению достоверности различных оценок консервативности: Highly significant; Significant; Informative; Other, где [Оценка] - оценка консервативности: Integrated, GM12878, H1-hESC, HUVEC. Фильтр с мультиселектом.
- [Распределение] - фильтр вариантов по стационарному распределению аллелей в сайте, оцененному с помощью алгоритма SiPhy, где [Распределение] - распределение аллелей A, G, C или T: pA, pG, pC, pT. Фильтр с вводом числового значения (положительное рациональное число).
ExAC#
Колонки фильтрации со следующими данными из базы ExAC:
- AF - фильтр вариантов по суммарной частоте аллеля с вводом числового значения (положительное рациональное число).
- Allele count - фильтр вариантов по количеству альтернативного аллеля с вводом числового значения (натуральное число).
- Adjusted AF - фильтр вариантов по частоте аллеля (AF) в популяции с качеством генотипа ≥20 и глубиной ≥10. Фильтр с вводом числового значения (положительное рациональное число).
- Adjusted allele count - фильтр вариантов по количеству альтернативного аллеля в популяции с качеством генотипа ≥20 и глубиной ≥10. Фильтр с вводом числового значения (натуральное число).
- [Популяция] AF - фильтр вариантов по суммарной частоте аллеля (AF) в различных популяциях, где [Популяция]: African & African American, American, East Asian, Finnish, Non-Finnish European, South Asian. Фильтр с вводом числового значения (положительное рациональное число).
- [Популяция] allele count - фильтр вариантов по количеству альтернативного аллеля в различных популяциях, где [Популяция]: African & African American, American, East Asian, Finnish, Non-Finnish European, South Asian. Фильтр с вводом числового значения (положительное рациональное число).
- [Когорта] AF - фильтр вариантов по суммарной частоте аллеля (AF) в различных когортах, где [Когорта]: Non-TCGA, Non-Psych. Фильтр с вводом числового значения (положительное рациональное число).
- [Когорта] Allele count - фильтр вариантов по количеству альтернативного аллеля в различных когортах, где [Когорта]: Non-TCGA, Non-Psych. Фильтр с вводом числового значения (натуральное число).
- [Когорта] adjusted AF - фильтр вариантов по частоте аллеля (AF) в популяции с качеством генотипа ≥20 и глубиной ≥10 в различных когортах, где [Когорта]: Non-TCGA, Non-Psych. Фильтр с вводом числового значения (положительное рациональное число).
- [Когорта] adjusted allele count - фильтр вариантов по количеству альтернативного аллеля в популяции с качеством генотипа ≥20 и глубиной ≥10в различных когортах, где [Когорта]: Non-TCGA, Non-Psych. Фильтр с вводом числового значения (натуральное число).
- [Когорта] [Популяция] AF - фильтрация по суммарной частоте аллеля (AF) в различных популяциях в различных когортах, где [Когорта]: Non-TCGA, Non-Psych, и [Популяция]: African & African American, American, East Asian, Finnish, Non-Finnish European, South Asian. Фильтр с вводом числового значения (положительное рациональное число).
- [Когорта] [Популяция] allele count - фильтр вариантов по количеству альтернативного аллеля в различных популяциях в различных когортах, где [Когорта]: Non-TCGA, Non-Psych, и [Популяция]: African & African American, American, East Asian, Finnish, Non-Finnish European, South Asian. Фильтр с вводом числового значения (натуральное число).
Protein function effect#
- [Алгоритм] [Оценка] - фильтр вариантов по оценкам предсказания эффекта аминокислотной замены на функцию белка, полученным с помощью различных алгоритмов и программ, где [Алгоритм] - алгоритм предсказания: SIFT, PolyPhen, SIFT4G, LRT, FAtHMM, Mutation Assessor, и [Оценка] - оценка предсказания: rankscore, score, omega, p-value. Фильтр с вводом числового значения (положительное рациональное число).
- [Алгоритм] prediction - фильтр вариантов по результатам предсказания эффекта аминокислотной замены на функцию белка, полученным с помощью различных алгоритмов и программ, где [Алгоритм] - алгоритм предсказания: PolyPhen, SIFT4G, LRT, FAtHMM, Mutation Assessor. Фильтр с мультиселектом.
- SpliceAI Delta score - фильтр по вероятности того, что вариант влияет на сплайсинг в любой позиции в пределах заданного интервала вокруг него (+/- 50 п.н. по умолчанию), предсказанной SpliceAI. Фильтр с вводом числового значения (любое число).
- SpliceAI Delta score (acceptor gain) - фильтр по вероятности того, что позиция в пределах заданного интервала вокруг варианта (+/- 50 п.н. по умолчанию) используется как акцептор сплайсинга, предсказанной SpliceAI. Фильтр с вводом числового значения (любое число).
- SpliceAI Delta score (acceptor loss) - фильтр по вероятности того, что позиция в пределах заданного интервала вокруг варианта (+/- 50 п.н. по умолчанию) не используется как акцептор сплайсинга, предсказанной SpliceAI. Фильтр с вводом числового значения (любое число).
- SpliceAI Delta score (donor gain) - фильтр по вероятности того, что позиция в пределах заданного интервала вокруг варианта (+/- 50 п.н. по умолчанию) используется как донор сплайсинга, предсказанной SpliceAI. Фильтр с вводом числового значения (любое число).
- SpliceAI Delta score (donor loss) - фильтр по вероятности того, что позиция в пределах заданного интервала вокруг варианта (+/- 50 п.н. по умолчанию) не используется как донор сплайсинга, предсказанной SpliceAI. Фильтр с вводом числового значения (любое число).
- SpliceAI Delta position - фильтр по позиции (в п.н.) относительно варианта, в которой изменяется сплайсинг (положительные значения — ниже позиции варианта, отрицательные значения — выше). Фильтр с вводом числового значения (целое число).
- SpliceAI Delta position (acceptor gain) - фильтр по позиции (в п.н.) относительно варианта, которая используется как акцептор сплайсинга. Фильтр с вводом числового значения (целое число).
- SpliceAI Delta position (acceptor loss) - фильтр по позиции (в п.н.) относительно варианта, которая не используется как акцептор сплайсинга. Фильтр с вводом числового значения (целое число).
- SpliceAI Delta position (donor gain) - фильтр по позиции (в п.н.) относительно варианта, которая используется как донор сплайсинга. Фильтр с вводом числового значения (целое число).
- SpliceAI Delta position (donor loss) - фильтр по позиции (в п.н.) относительно варианта, которая не используется как донор сплайсинга. Фильтр с вводом числового значения (целое число).
- CADD raw - фильтр по степени, в которой профиль аннотации для данного варианта предполагает, что вариант, вероятно, будет «наблюдаемым» (отрицательные значения) по сравнению с «моделированным» (положительные значения), рассчитанной CADD. Фильтр с вводом числового значения (любое число).
- CADD phred - фильтр по Phred-масштабированной нормализованной оценке вредоносности вариантов, рассчитанной CADD. Фильтр с вводом числового значения (любое число).
- MaxEntScan Alt score - фильтр по оценке для альтернативного нуклеотида варианта, предсказывающей потерю естественного сайта сплайсинга, рассчитанной MaxEntScan. Фильтр с вводом числового значения (любое число).
- MaxEntScan Ref score - фильтр по оценке для референсного нуклеотида варианта, предсказывающей потерю естественного сайта сплайсинга, рассчитанной MaxEntScan. Фильтр с вводом числового значения (любое число).
- MaxEntScan diff - фильтр по разнице между оценками для альтернативного и референсного нуклеотидов варианта, предсказывающими потерю естественного сайта сплайсинга, рассчитанными MaxEntScan. Фильтр с вводом числового значения (любое число).
Protein function effect (additional)#
- [Алгоритм] [Оценка] - фильтр вариантов по оценкам предсказания эффекта аминокислотной замены на функцию белка, полученным с помощью различных алгоритмов и программ, где [Алгоритм] - алгоритм предсказания: BayesDel addAF, BayesDel noAF, Provean, Meta SVM, Meta LR, M-CAP, MutPred, MVP, MPC, Primate AI, DEOGEN2, DANN, FAtHMM-MKL, FAtHMM-XF, Eigen, и [Оценка] - оценка предсказания: rankscore, score, p-value. Фильтр с вводом числового значения (положительное рациональное число).
- [Алгоритм] prediction - фильтр вариантов по результатам предсказания эффекта аминокислотной замены на функцию белка, полученным с помощью различных алгоритмов и программ, где [Алгоритм] - алгоритм предсказания: BayesDel addAF, BayesDel noAF, Provean, Meta SVM, Meta LR, M-CAP, Primate AI, FAtHMM-MK, FAtHMM-XF. Фильтр с мультиселектом.
- Transcript ID - фильтр вариантов по идентификатору транскрипта в базе UniProtKB (например, P16422), для которого было сделано предсказание эффекта аминокислотной замены на функцию белка с помощью MutPred. Фильтр с вводом любого значения.
- Amino acid change - фильтр вариантов по аминокислотной замене, предсказание об эффекте которой на функцию белка было сделано с помощью MutPred. Фильтр с вводом любого значения. Пример: G35r.
- Top 5 features - фильтр вариантов по пяти основным эффектам замены на функцию белка, предсказанных с помощью MutPred. Фильтр с вводом любого значения.
Other frequencies#
- 1000G Allele frequency - фильтр вариантов по суммарной частоте аллеля из проекта 1000 Genomes. Фильтр с вводом числового значения (положительное рациональное число).
- 1000G [Популяция] AF - фильтр вариантов по суммарной частоте аллеля (AF) в различных популяциях из проекта 1000 Genomes, где [Популяция]: East Asian, European, African, American, South Asian. Фильтр с вводом числового значения (положительное рациональное число).
- UK10K allele frequency - фильтр вариантов по суммарной частоте аллеля в комбинированных генотипах в когорте UK10K из проекта UK10K. Фильтр с вводом числового значения (положительное рациональное число).
- UK10K allele count - фильтр вариантов по количеству альтернативного аллеля в комбинированных генотипах в когорте UK10K из проекта UK10K. Фильтр с вводом числового значения (натуральное число).
- UK10K [Когорта] AF - фильтр вариантов по суммарной частоте аллеля (AF) в комбинированных генотипах в различных когортах из проекта UK10K, где [Когорта]: TwinsUK, ALSPAC. Фильтр с вводом числового значения (положительное рациональное число).
- UK10K [Когорта] allele count - фильтр вариантов по количеству альтернативного аллеля в комбинированных генотипах в различных когортах из проекта UK10K, где [Когорта]: TwinsUK, ALSPAC. Фильтр с вводом числового значения (натуральное число).
- ESP [Популяция] AF - фильтр вариантов по суммарной частоте аллеля (AF) в различных популяциях из проекта NHLBI GO Exome Sequencing Project (ESP), где [Популяция]: African American, European American. Фильтр с вводом числового значения (положительное рациональное число).
- [Популяция] allele count - фильтр вариантов по количеству альтернативного аллеля в различных популяциях из проекта NHLBI GO Exome Sequencing Project (ESP), где [Популяция]: African American, European American. Фильтр с вводом числового значения (натуральное число).
Other#
- Coding sequence position - фильтр вариантов по позиции в кодирующей области с вводом числового значения (натуральное число).
- Protein position - фильтр вариантов по позиции в белке с вводом числового значения (натуральное число).
- Amino acids - фильтр вариантов по референсной аминокислоте с вводом любого значения.
- Codons - фильтр вариантов по аминокислотной замене, записанной в виде кодонов, с вводом любого значения. Пример: GAA/GAG.
- Amino acid position - фильтр вариантов по позиции в аминокислоте с вводом числового значения (натуральное число).
- APPRIS annotation - фильтр вариантов по изоформам сплайсинга человека (аннотация базы APPRIS): PRINCIPAL1-5; ALTERNATIVE1-2. Фильтр с мультиселектом.
- Geuvadis EQTL target gene - фильтр вариантов по гену из анализа eQTL проекта GEUVADIS. Фильтр с вводом любого значения.
- ENIGMA clinical significance - фильтр по клинической значимости варианта, расположенного в известном или предполагаемом гене предрасположенности к раку молочной железы и/или яичников (BRCA1, BRCA2 и др.), выявленной экспертами из консорциума ENIGMA. Фильтр с мультиселектом:
- Pathogenic - Патогенный;
- Likely pathogenic - Вероятно патогенный;
- Benign - Доброкачественный;
- Likely benign - Вероятно доброкачественный;
- Not provided - Не предоставлена.
- ENIGMA clinical significance comment - фильтр по комментарию о клинической значимости варианта, расположенного в известном или предполагаемом гене предрасположенности к раку молочной железы и/или яичников (BRCA1, BRCA2 и др.), выявленной экспертами из консорциума ENIGMA. Фильтр с вводом любого значения.
Custom annotation sources#
Группа колонок фильтрации представлена, если хотя бы одна пользовательская аннотация была добавлена в систему до того, как варианты в образце были проаннотированы. Названия колонок фильтрации выглядят следующим образом: [название пользовательской аннотации]: [название колонки аннотации]. Чтобы добавить условие фильтрации вариантов по колонке из пользовательской аннотации, сделайте следующее:
- Выберите оператор фильтрации. Если тип колонки аннотации текстовый, то операторы будут следующие:
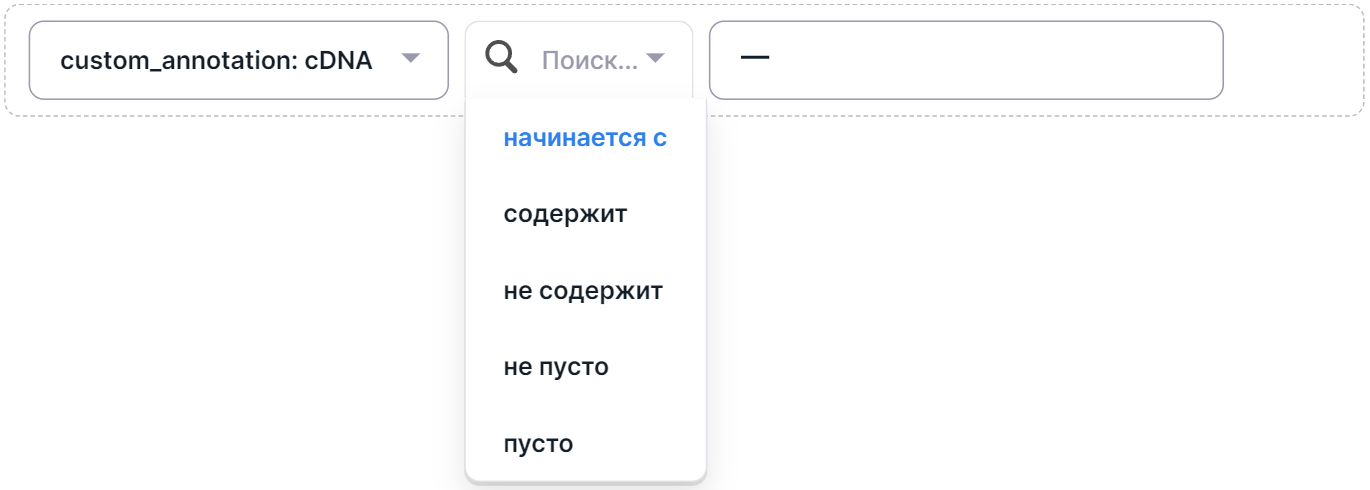
А если тип колонки аннотации числовой, то операторы будут такими:
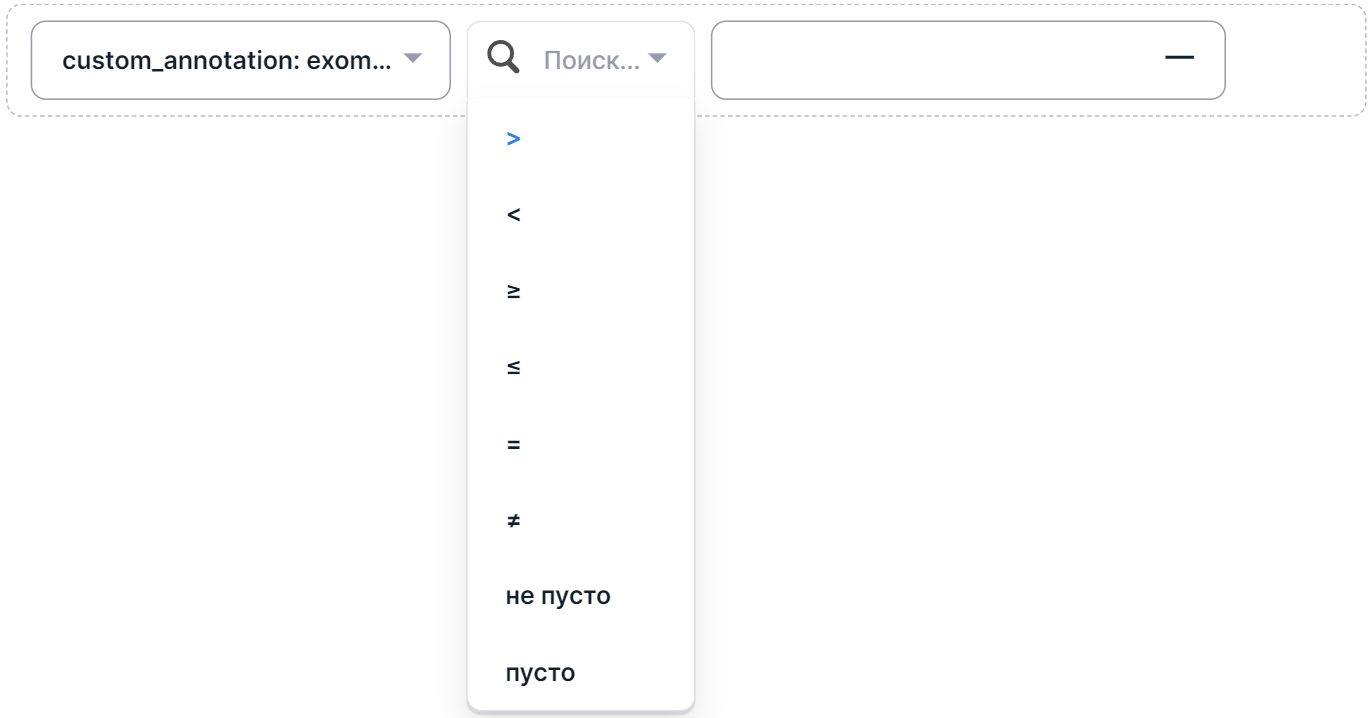
- Если были выбраны операторы, отличные от "не пусто" и "пусто", нажмите на
окно
вводаи введите нужное значение колонки фильтрации. Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
Режим фильтрации по компаунд-гетерозиготе#
Компаунд-гетерозигота - это такое генетическое состояние, когда у пациента два или более аллеля одного гена несут разные мутации. Такое состояние наблюдается, если пациент наследует по мутантному аллелю от каждого родителя или наследует одну мутацию от одного из родителей, а вторая возникла de novo. Компаунд-гетерозигота часто встречается при аутосомно-рецессивных заболеваниях.
Если есть данные об образцах секвенирования, в конструкторе запросов появляется кнопка для фильтрации по компаунд-гетерозиготе:
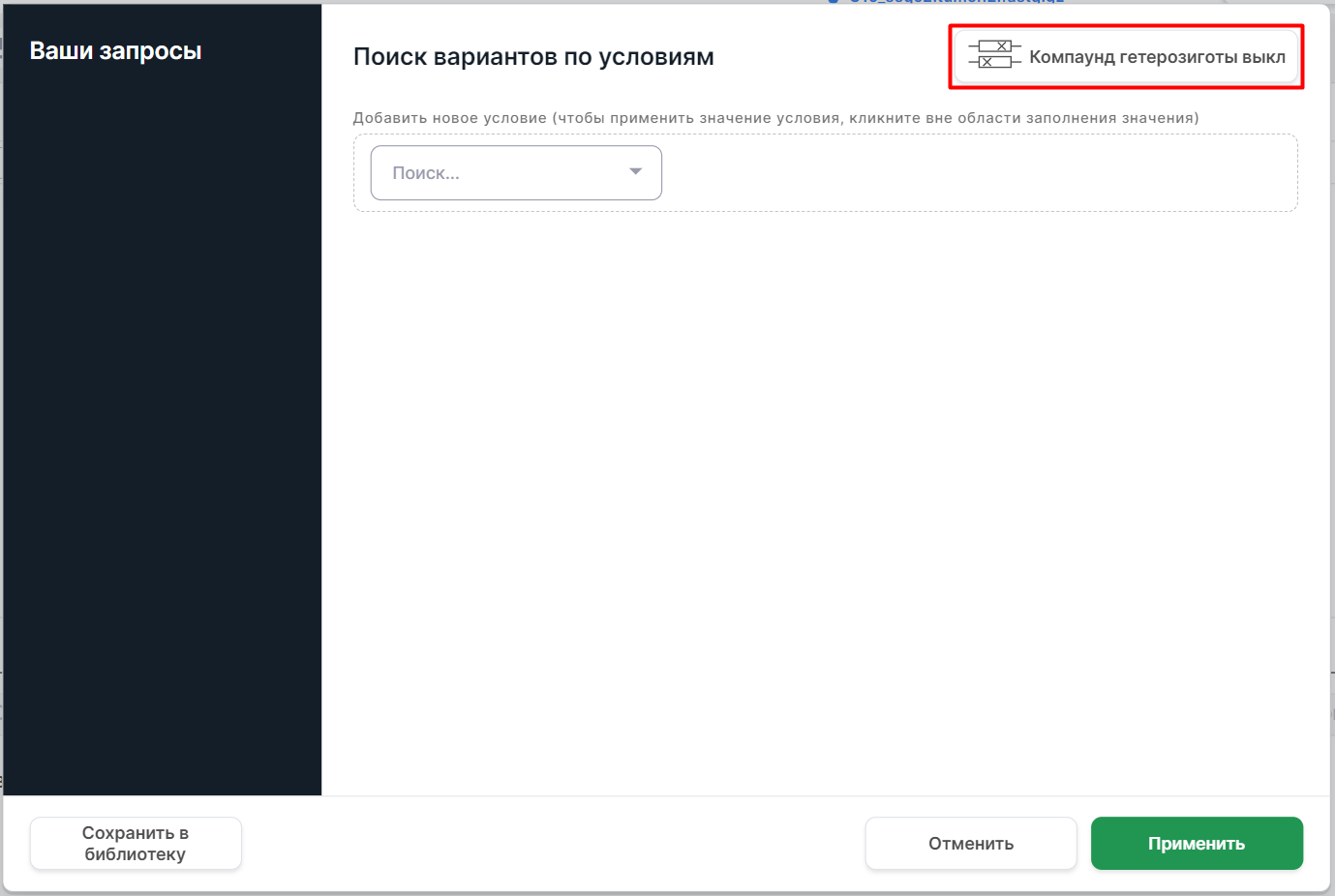
Фильтрация по компаунд-гетерозиготе зависит от типа SNV Viewer:
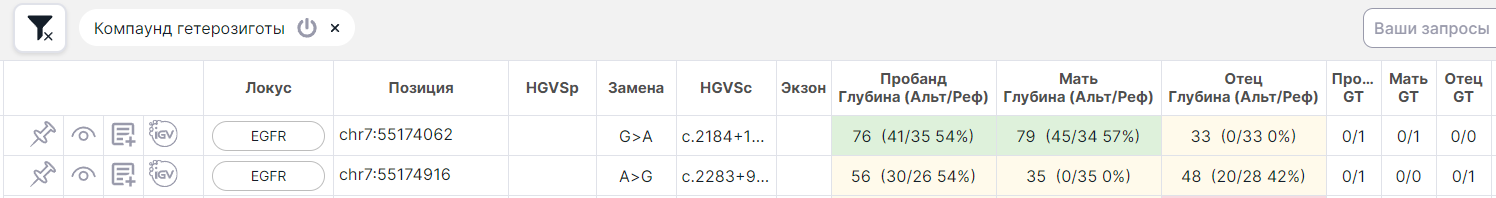
SNV Viewer семейного трио анализа (Пробанд, Отец, Мать): у пробанда в одном гене хотя бы две гетерозиготы (0/1, 1/0, 0|1 и т.п.) со следующими вариантами наследования:
- хотя бы одна из гетерозигот наследуется от отца, а хотя бы одна - от матери:
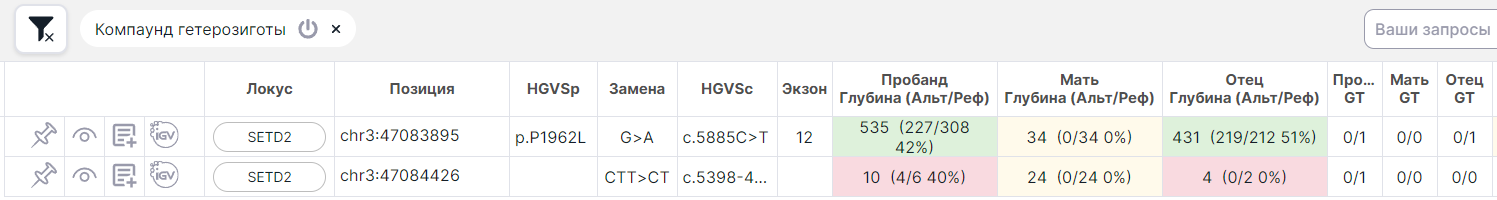
- хотя бы одна из гетерозигот возникла de novo (т.е. у отца и матери это гомозиготы по референсу 0/0), а вторая наследуется от отца или от матери:
Для применения фильтра по компаунд-гетерозиготе нажмите
на кнопку . В результате найдутся все случаи, когда хотя бы две гетерозиготы расположены в одном гене у пробанда, а у родителей это либо гетерозиготы, либо гомозиготы по референсу, либо нет информации о сайте (./.). Для выключения фильтра в конструкторе запросов нажмите на кнопку
 .
.
SNV Viewer семейного дуо анализа (Отец и Мать): случаи, когда в одном гене хотя бы одна гетерозигота (0/1, 1/0, 0|1 и т.п.) у матери и хотя бы одна - у отца:
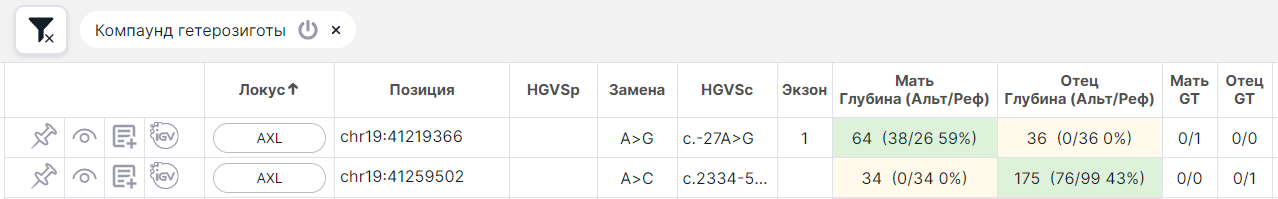
Для применения фильтра по компаунд-гетерозиготе нажмите
на кнопку , а для выключения фильтра в конструкторе запросов нажмите на кнопку
 .
.
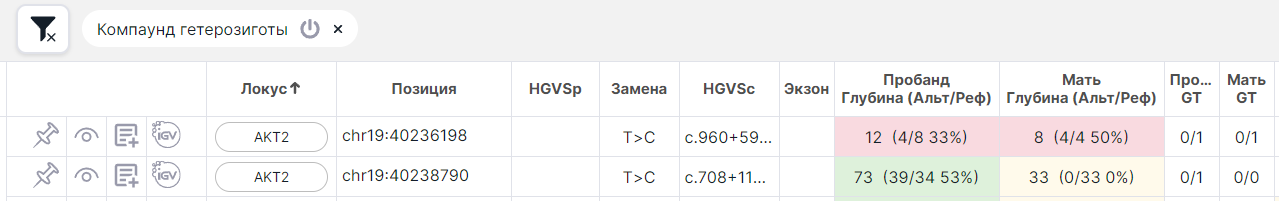
SNV Viewer семейного дуо анализа (Пробанд, Родитель (Отец или Мать)): у пробанда в одном гене хотя бы две гетерозиготы (0/1, 1/0, 0|1 и т.п.) со следующими вариантами наследования:
- хотя бы одна наследуется, а вторая не наследуется от родителя (т.е. у родителя это гомозигота по референсу 0/0):
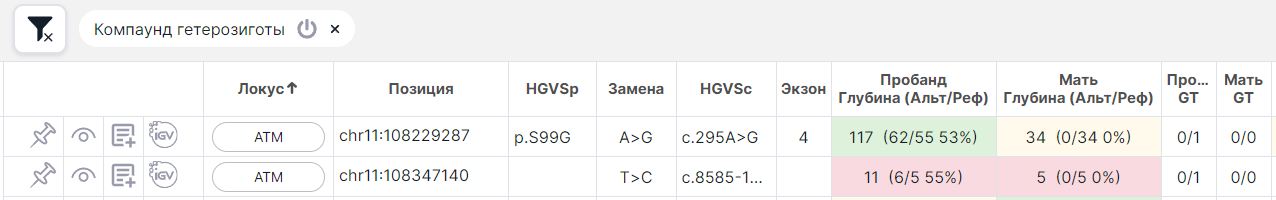
- обе не наследуются от родителя (т.е. у родителя это гомозиготы по референсу 0/0):
Для применения фильтра по компаунд-гетерозиготе нажмите
на кнопку , а для выключения фильтра в конструкторе запросов нажмите на кнопку
 .
.
SNV Viewer с одним образцом секвенирования (в частности, для одиночного образца опухолевой или здоровой ткани): найдутся
все случаи, когда хотя бы две гетерозиготы (0/1, 1/0, 0|1 и т.п.) расположены в одном гене.
Для применения фильтра по компаунд-гетерозиготе нажмите
на кнопку , а для выключения фильтра в конструкторе запросов нажмите на кнопку
 .
.
SNV Viewer группового анализа герминальной когорты или SNV Viewer с несколькими образцами секвенирования (в частности, для пары образцов опухоль/контроль), или SNV Viewer запуска (если есть данные об образцах секвенирования): нажав на кнопку фильтра по компаунд-гетерозиготе, вы увидите выпадающий список с образцами:
При выборе одного из образцов варианты будут отфильтрованы таким образом, что в этом образце в одном гене будут хотя бы две гетерозиготы (0/1, 1/0, 0|1 и т.п.) Чтобы выключить такой фильтр в конструкторе запросов, нужно нажать на кнопку фильтра по компаунд-гетерозиготе и выбрать опцию "Выключить".
Применение режима фильтрации по компаунд-гетерозиготе с другими условиями фильтрации:
При применении режима фильтрации по компаунд-гетерозиготе совместно с другими условиями фильтрации условие на компаунд-гетерозиготу применяется уже к тем вариантам, которые подходят под остальные условия:
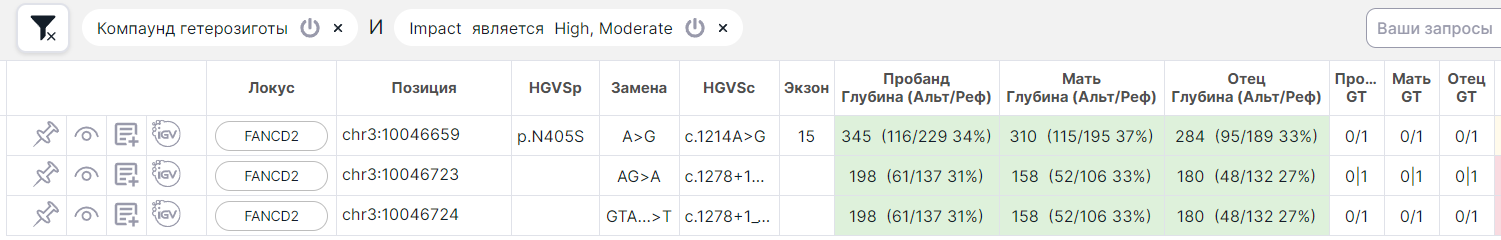
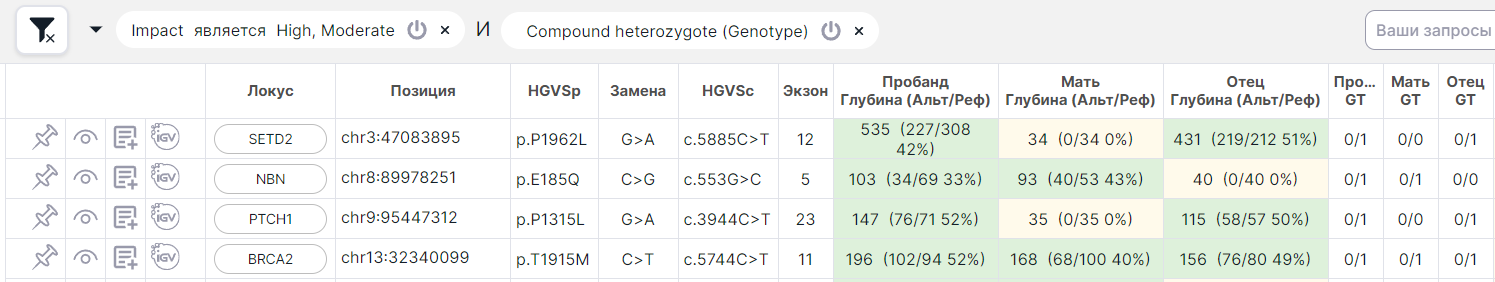
Этим режим фильтрации принципиально отличается от фильтра "Compound heterozygote (Genotype)", который применяется одновременно с остальными условиями фильтрации, т.е. в результате применения фильтра найдутся варианты, которые входят в компаунд-гетерозиготу, но остальные варианты из этой компаунд-гетерозиготы могут отфильтроваться по другим условиям:
Добавление первого условия фильтрации#
Чтобы добавить первое условие фильтрации, нажмите на выделенное окно:

Вы увидите список колонок аннотации, доступных для фильтрации вариантов:
Техника добавления условия фильтрации различается в зависимости от типа фильтра:
1. Фильтры с мультиселектом#
- Возможность выбора сразу нескольких значений колонки.
- Техника добавления условия фильтрации:
- Выберите колонку фильтрации из списка всех колонок;
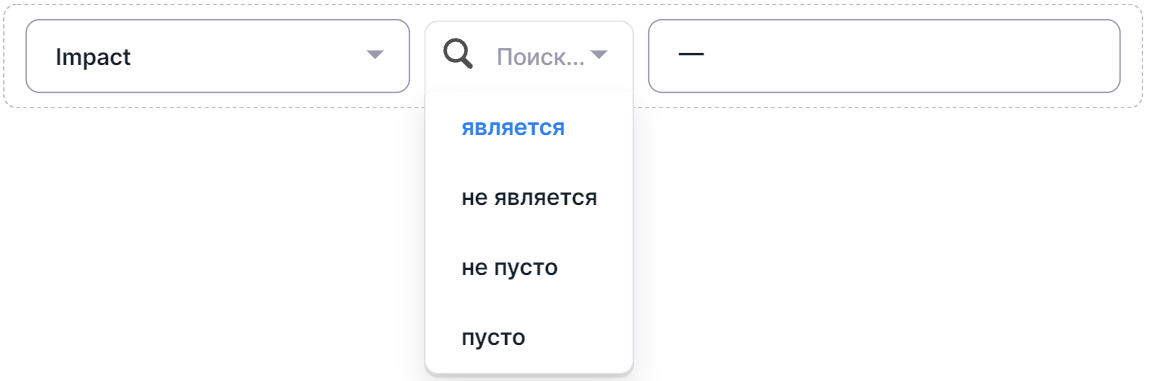
- Выберите оператор фильтрации:
- "является";
- "не является";
- "не пусто" - у искомых вариантов в аннотации выбранная колонка имеет не пустое значение (опции нет для колонок "Chromosome" и "Origin");
- "пусто" - у искомых вариантов в аннотации выбранная колонка имеет пустое значение (опции нет для колонок "Chromosome" и "Origin").
- Если были выбраны операторы "является" или "не является", выберите необходимые значения колонки фильтрации, нажав на строки значений:
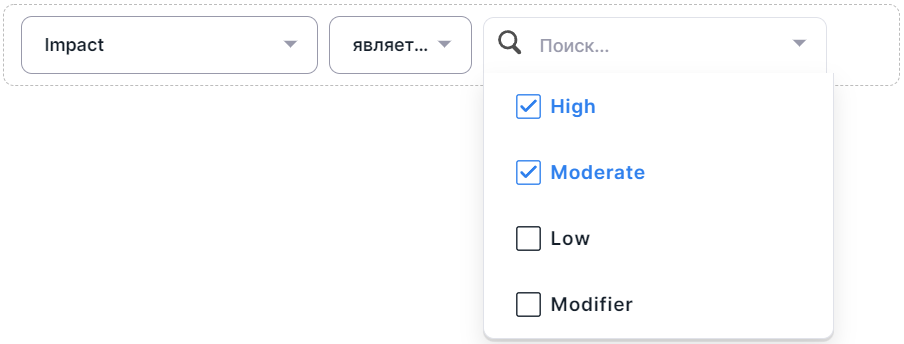
- Чтобы закончить добавление условия, кликните вне области заполнения значения.
2. Фильтры с выбором одного значения#
- Возможность выбора только одного значения фильтра.
- Техника добавления условия фильтрации:
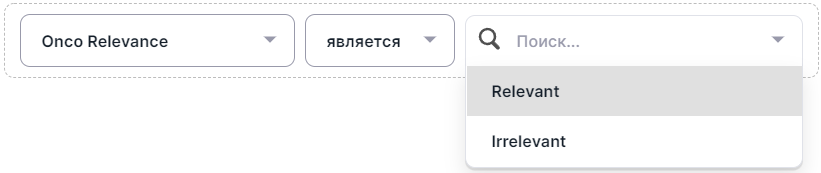
- Выберите фильтр из списка всех фильтров. Для фильтров этого типа автоматически выбран оператор фильтрации "является";
- Выберите необходимое значение фильтра, нажав на строку значения:
3. Фильтры с вводом идентификатора#
- Возможность ввода нужного значения колонки.
- Техника добавления условия фильтрации:
- Выберите колонку фильтрации из списка всех колонок;
- Выберите оператор фильтрации:
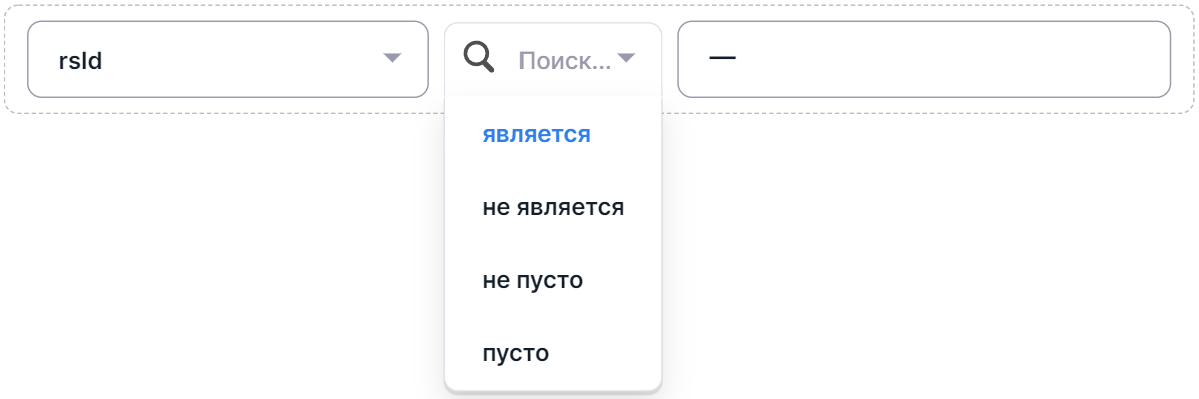
- Если были выбраны операторы "является" или "не является", нажмите на
окно
вводаи введите нужное значение колонки фильтрации:
Можно ввести сразу несколько идентификаторов списком через запятую
(например, rs1234, rs2799066, rs4648562), точку с запятой или пробел (rs1234 rs2799066 rs4648562).
- Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
4. Фильтры с вводом числового значения#
- Возможность ввода нужного значения фильтра.
- Техника добавления условия фильтрации:
- Выберите колонку фильтрации из списка всех колонок;
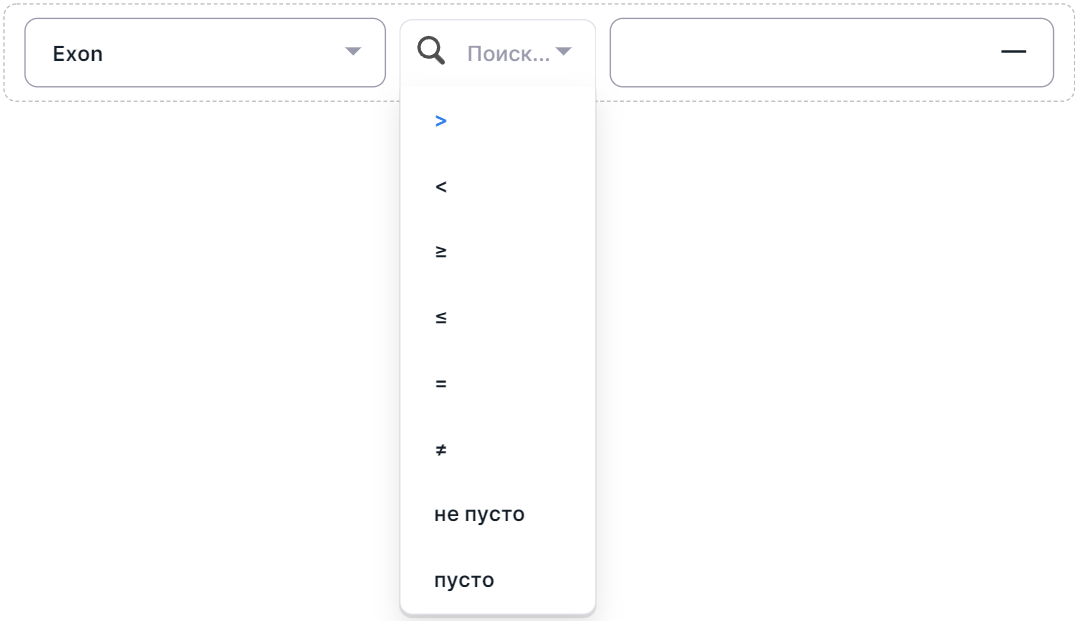
- Выберите оператор фильтрации:
- ">";
- "<";
- "≥";
- "≤";
- "=";
- "≠";
- "не пусто" - у искомых вариантов в аннотации выбранная колонка имеет не пустое значение (опции нет для колонок "Start" и "End");
- "пусто" - у искомых вариантов в аннотации выбранная колонка имеет пустое значение (опции нет для колонок "Start" и "End").
- Если в качестве оператора фильтрации был выбран математический оператор, нажмите на
окно
вводаи введите нужное значение колонки фильтрации:
- Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
5. Фильтры с вводом любого значения#
- Возможность ввода нужного значения фильтра.
- Техника добавления условия фильтрации:
- Выберите колонку фильтрации из списка всех колонок;
- Выберите оператор фильтрации:
- "начинается с" (опции нет для колонки "HGVSc");
- "содержит";
- "не содержит" (опции нет для колонки "HGVSc");
- "не пусто" - у искомых вариантов в аннотации выбранная колонка имеет не пустое значение (опции нет для колонки "Reference allele");
- "пусто" - у искомых вариантов в аннотации выбранная колонка имеет пустое значение (опции нет для колонки "Reference allele").
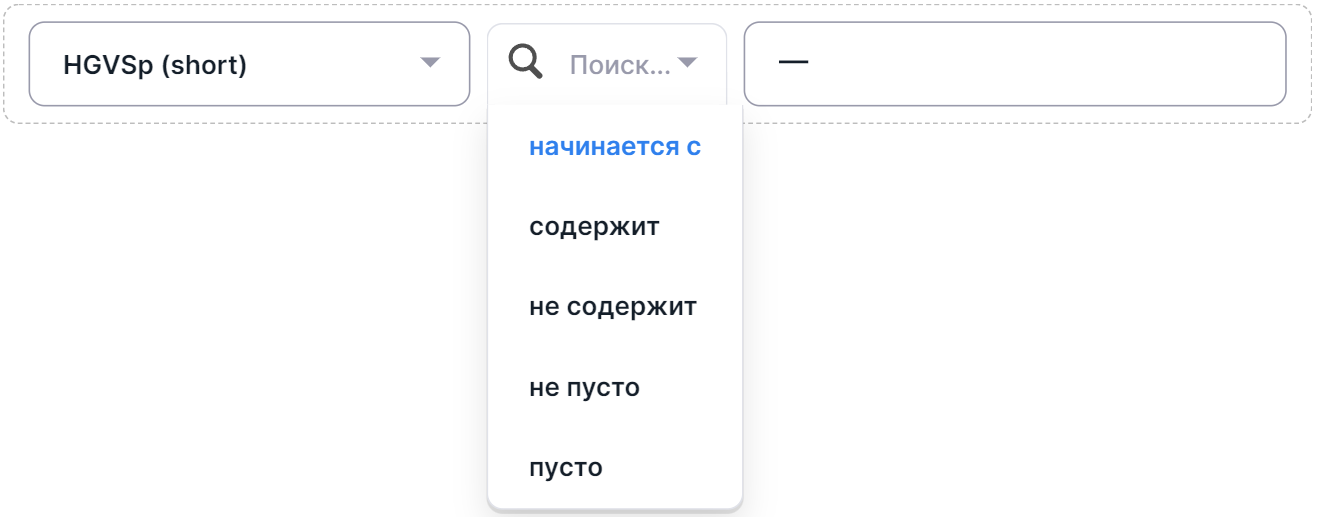
- Если были выбраны операторы "начинается с", "содержит" или "не содержит", нажмите на
окно
вводаи введите нужное значение колонки фильтрации:

- Чтобы закончить добавление условия, нажмите клавишу ввода или кликните вне области заполнения значения.
Редактирование условия#
В уже составленном условии можно поменять оператор и/или значение. Для этого нужно нажать на соответствующее поле:
и выбрать другую опцию:
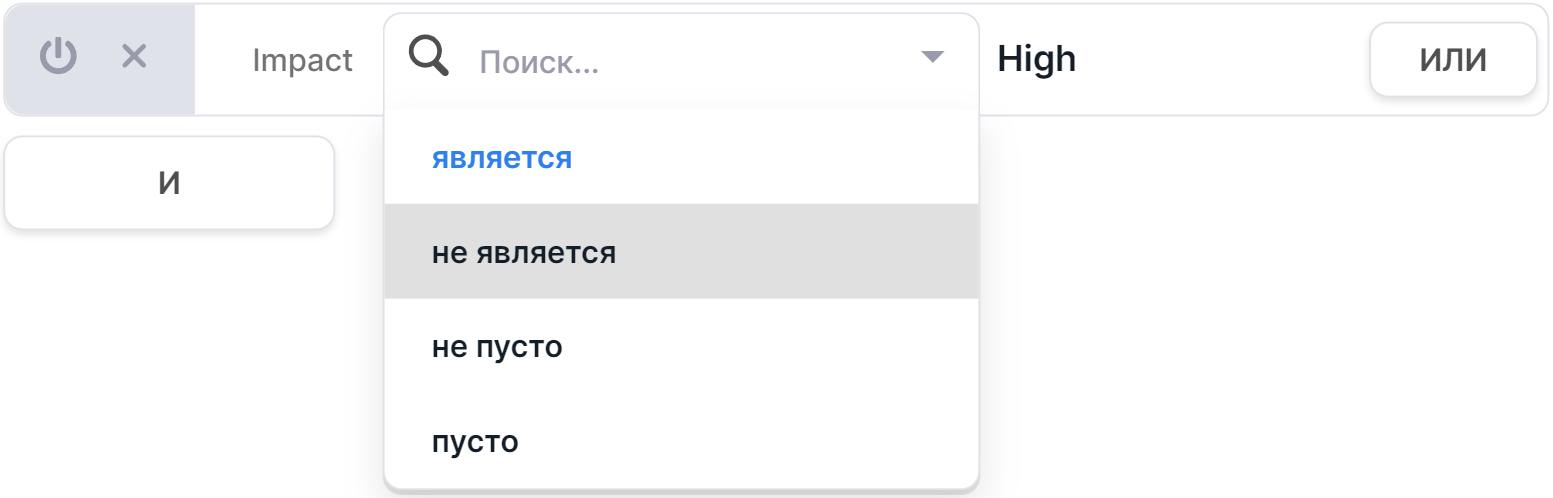
Выключение условия#
Эта опция полезна, если вы не хотите потерять составленное условие, но в данный момент вам не
нужна фильтрация по нему. Чтобы выключить условие, наведите на него курсор и
нажмите на :

Удаление условия#
Чтобы удалить условие, наведите на него курсор и
нажмите на :

Совет
Ещё не заполненное условие тоже можно удалить (если это не самое первое условие в конструкторе запросов):

Добавление следующего условия фильтрации#
Если вы хотите добавить ещё одно условие к запросу, сначала решите, хотите ли вы, чтобы искомый вариант строго подходил под оба условия (уже добавленное и новое) или же вариант может подходить под одно из условий:
Искомый вариант подходит под оба условия#
- После создания первого условия нажмите на
.
- Заполните условие, как описано в соответствующем разделе. Чтобы начать, нажмите на выделенное окно:

Искомый вариант подходит под одно из условий#
- После создания первого условия наведите курсор на него и нажмите на
появившуюся кнопку
.
- Заполните условие, как описано в соответствующем разделе. Чтобы начать, нажмите на выделенное окно:

Применение запроса#
После того, как вы заполнили все условия к искомым вариантам,
нажмите на .
Окно конструктора запросов закроется и запрос будет применен к вариантам. Сверху таблицы можно увидеть, какой
запрос применен:
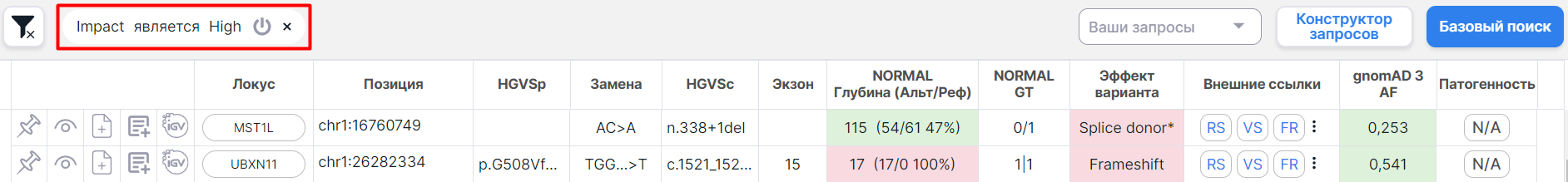
Условие можно выключить, нажав на , или удалить, нажав на
:

Для очистки всех условий фильтрации
нажмите на .
Чтобы изменить, сохранить или создать новый запрос, перейдите
в конструктор запросов,
нажав на  .
.
Чтобы вернуться к основному режиму SNV Viewer,
нажмите на .
Иногда условий к искомым вариантам много и запрос получается очень длинным:
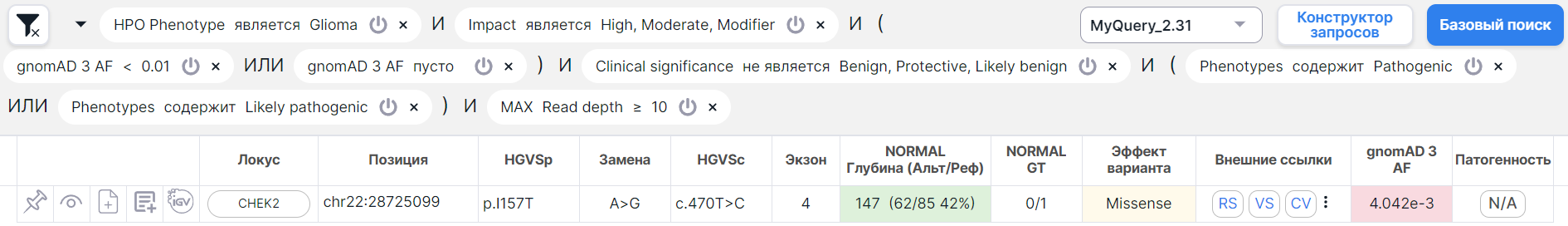
В таком случае можно свернуть условия запроса,
нажав на кнопку , расположенную справа от
кнопки Очистки всех условий фильтрации. Тогда будут показаны только начальные условия запроса:

Чтобы развернуть условия запроса обратно, нажмите на .
На заметку
На странице "Настройки пользователя" можно отрегулировать, будут ли условия запроса всегда в развёрнутом или свёрнутом состоянии сразу после применения запроса фильтрации.
Сохранение запроса#
Если вы хотите переиспользовать созданный запрос в дальнейшем, сохраните его. Для этого перейдите в конструктор
запросов, нажав на  , а затем нажмите на
, а затем нажмите на .
Введите имя запроса:
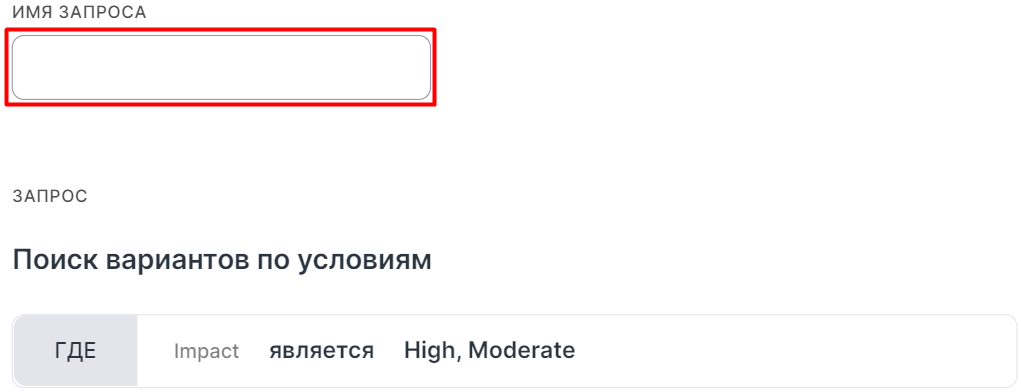
и нажмите .
Сохраненный запрос появится в конструкторе запросов слева, в списке Ваши запросы. Для применения сохраненного запроса нажмите на его строку:
а затем нажмите на .
Кроме того, сохраненный запрос появится в опциях базового фильтра "Ваши запросы".
Если вы хотите удалить сохраненный запрос, нажмите на строку запроса,
а затем на .