Аннотация
После успешной загрузки, идентификации и проверки образец переходит к основной стадии анализа - аннотации. Если в загруженной аннотации есть SNVs/Indels, то запустится стадия "Аннотация герминальных SNVs/Indels" (если образец был загружен как образец неопухолевой ткани) или "Аннотация соматических SNVs/Indels" (если образец был загружен как образец опухолевой ткани). Если же в загруженной аннотации не обнаружены SNVs/Indels, но есть структурные вариации, то запустится стадия "Аннотация герминальных структурных вариаций" (если образец был загружен как образец неопухолевой ткани) или "Аннотация соматических структурных вариаций" (если образец был загружен как образец опухолевой ткани). При ошибке выполнения любой из перечисленных ниже задач анализ образца останавливается.
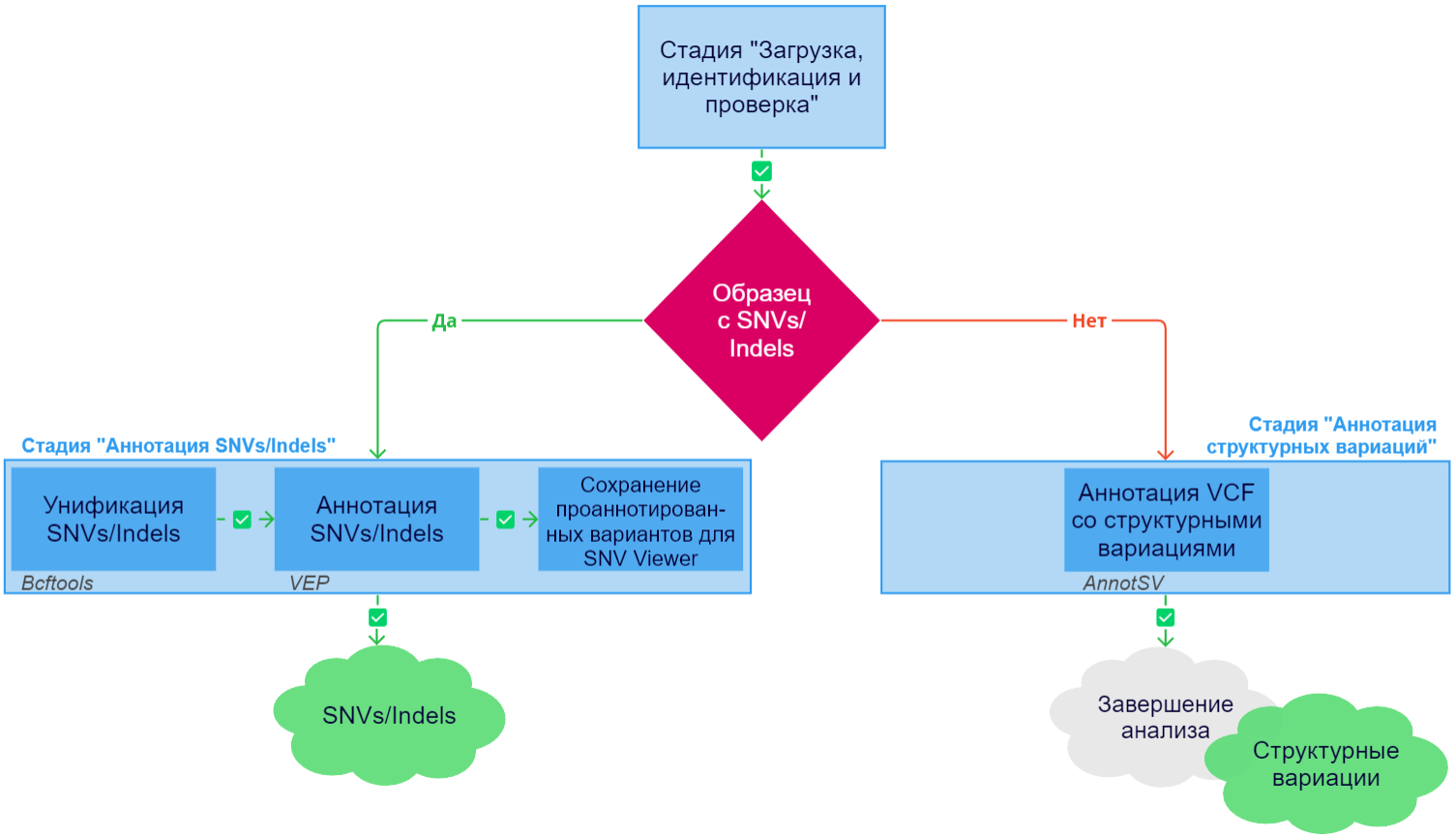
Задачи стадии "Аннотация SNVs/Indels":
- Унификация SNVs/Indels:
- Сортировка файла VCF с помощью GNU sort по словарю c названиями хромосом.
- Модификация заголовка файла и изменение названий образцов с помощью bcftools reheader.
- Замена или исправление заголовка файла с помощью GATK FixVCFHeader.
- Проверка того, соответствуют ли референсные аллели в файле референсной последовательности; разбивка мультиаллельных сайтов на биаллельные записи; вывод только первой записи для дублирующихся строк с помощью bcftools norm.
- Сжатие файла в GZIP архив с помощью bgzip. Получившийся файл можно скачать в разделе "Файлы с результатами" в деталях задачи "Унификация SNVs/Indels" ("Скачать VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере".
- Индексация файла с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать VCF_TBI").
Если в анализ образца были включены вычисление олигогенных рисков, вычисление полигенных рисков, расчет фармакогенетики и/или анализ происхождения, то после задачи "Унификация SNVs/Indels" начнется параллельное выполнение стадии "Геномные предсказания".
- Аннотация SNVs/Indels:
Аннотация SNVs/Indels в файле данными из баз RefSeq, 1000 Genomes, dbNSFP, dbSNP, gnomAD 3, gnomAD 4, ClinVar, CADD, SpliceAI, ENIGMA.
Определение влияния SNVs/Indels на гены, транскрипты, белковые последовательности и регуляторные области с помощью Ensembl Variant Effect Predictor (VEP):
PolyPhen предсказывает возможное влияние замены аминокислоты на структуру и функцию белка, используя простые физические и сравнительные соображения.
Отмечается, является ли транскрипт, в котором расположен вариант, каноническим транскриптом для гена.
Определяется номер аллеля из входных данных VCF.
Определяется номер затронутых экзонов и интронов.
Добавляется номенклатура HGVS на основе стабильных идентификаторов Ensembl.
Варианты определяются как upstream, если они расположены перед геном и расстояние между вариантом и транскриптом больше 2000 п.о., и downstream, если варианты расположены после гена и расстояние между вариантом и транскриптом больше 1000 п.о.
Получившийся файл с сырыми аннотированными вариантами в формате TSV можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация SNVs/Indels" ("Скачать Raw annotated TSV"). Его также можно открыть в таблицах Google.
Получившийся файл с аннотированными вариантами без дубликатов в формате TSV можно скачать в том же разделе ("Скачать All variants TSV"). Его также можно открыть в таблицах Google. Тот же файл, но в формате CSV можно скачать там же ("Скачать All variants CSV").
Конвертация результатов в формате TSV в формат VCF.
Замена или исправление заголовка файла VCF с помощью GATK FixVCFHeader.
Сжатие файла VCF в GZIP архив с помощью bgzip. Получившийся файл можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация SNVs/Indels" ("Скачать All variants VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере".
Индексация файла VCF с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать All variants VCF_TBI").
Вычисление статистики вариантов.
- Сохранение проаннотированных вариантов для SNV Viewer: сохранение результатов для показа во встроенном модуле для просмотра и анализа вариантов SNV Viewer и добавление информации о встречаемости варианта в других образцах пользователя.
После стадии "Аннотация SNVs/Indels" анализ может продолжиться генерацией отчётов.
Стадия "Аннотация структурных вариаций" включает одну задачу "Аннотация VCF со структурными вариациями":
- Фильтрация и аннотация структурных вариаций с помощью AnnotSV.
Получившийся файл со структурными вариациями в формате TSV можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация VCF со структурными вариациями" ("Скачать Filtered variants TSV"). Его также можно открыть в таблицах Google. Тот же файл, но в формате CSV можно скачать там же ("Скачать Filtered variants CSV"). - Сжатие получившегося файла VCF в GZIP архив с помощью bgzip. Получившийся файл можно скачать в том же разделе ("Скачать Filtered variants VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере".
- Индексация файла VCF с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать Filtered variants VCF_TBI").
После стадии "Аннотация структурных вариаций" анализ образца заканчивается.
На заметку
Если вы хотите добавить в вашу десктопную версию IGV трек с вариантами или вариациями, выявленными в результате анализа загруженного вами образца в Genomenal, вы можете сделать это через ссылку. Откройте детали нужной задачи ("Унификация SNVs/Indels", "Аннотация SNVs/Indels", "Аннотация VCF со структурными вариациями") и сделайте следующее:
- Нажмите правой кнопкой мыши на ссылку файла с вариантами (в зависимости от выбранной задачи это может быть ссылка "Скачать VCF_GZ", "Скачать All variants VCF_GZ" или "Скачать Filtered variants VCF_GZ") и выберите опцию "Копировать адрес ссылки".
- Загрузите трек через URL в вашу десктопную версию IGV, как это описано здесь.
- Нажмите правой кнопкой мыши на ссылку для скачивания индексного файла, соответствующего файлу аннотации, ("Скачать VCF_TBI", "Скачать All variants VCF_TBI" или "Скачать Filtered variants VCF_TBI") и выберите опцию "Копировать адрес ссылки".
- Добавьте URL индексного файла в соответствующее поле в IGV.
- Нажмите "OK". Готово! Трек с вариантами или вариациями, выявленными в образце, добавлен в IGV.