Отчёт со вторичными находками ACMG
Отчёт содержит выявленные в образце варианты, которые расположены в генах из списка вторичных находок (secondary findings; SF) Американского колледжа медицинской генетики и геномики (ACMG). Для этих генов известны специфические мутации, приводящие к расстройствам с определенным фенотипом, на которые можно клинически подействовать с помощью общепринятого вмешательства. ACMG рекомендует сообщать о патогенных или вероятно патогенных вариантах, обнаруженных в любом из этих генов, поскольку они имеют медицинское значение и могут быть использованы в будущем для информирования о клиническом лечении.
Внимание!
Условия построения отчёта#
Отчёт строится для образца, если выполняются следующие условия:
- Образец загружен в формате FASTQ или BAM или в виде файла VCF c единственным образцом.
- Анализ образца успешно завершился (то есть все стадии анализа имеют статус "Завершено").
- Для набора образцов успешно завершилась стадия аннотации вариантов: "Аннотация герминальных SNVs/Indels", если на этапе создания шаблона отчёта был выбран тип мутации Germline, или "Аннотация соматических SNVs/Indels", если Somatic.
- Шаблон отчёта, включающий блок "SNVs/Indels по генам списка ACMG SF", добавлен в систему до того, как была завершена обработка образца, и активен.
Содержание отчёта#
Отчёт включает два раздела со списками вариантов, выявленных в образце, которые наполняются соответственно следующей схеме:
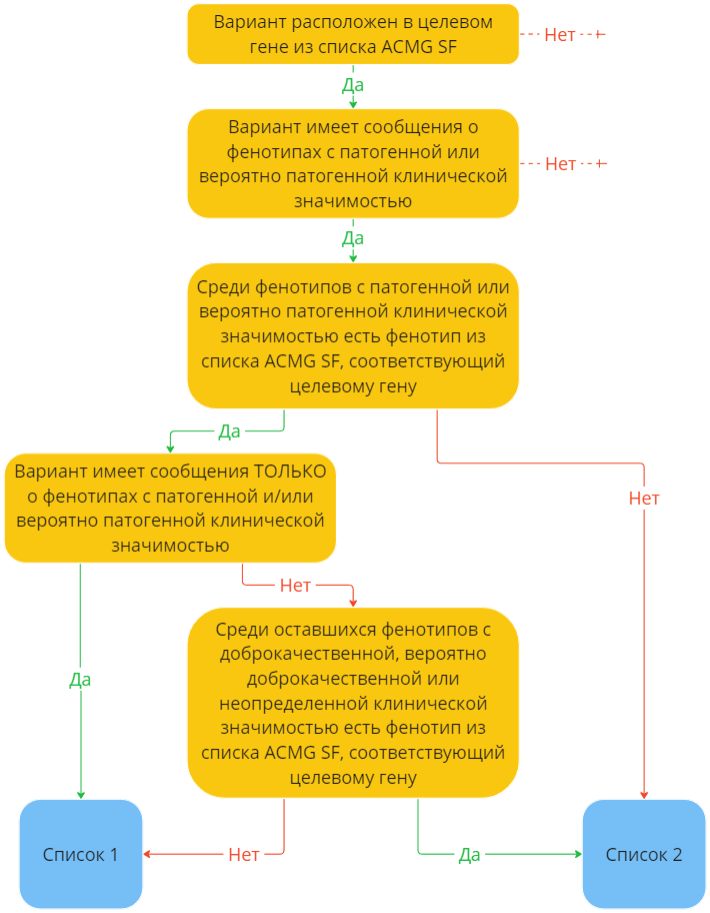
СПИСОК 1: Наиболее важные для интерпретации варианты, для которых выполняются следующие условия:
- Вариант расположен в гене из списка ACMG SF;
- Вариант ассоциирован по данным ClinVar с фенотипом только с патогенной или вероятно патогенной клинической значимостью, который указан в списке ACMG SF для гена, в котором расположен вариант;
- Частота аллеля по данным gnomAD 3 меньше порога частоты слишком частых патогенных вариантов, задаваемого в блоке шаблона отчёта "SNVs/Indels по генам списка ACMG SF" (рекомендуемое значение по умолчанию - 0,03), или имеет пустое значение;
- Сайт варианта содержит хотя бы один альтернативный аллель (т.е. имеет генотип 1/1, 1|1, 0/1, 1/0, 0|1 и т.п.) в случае файла VCF с единственным образцом;
- Для варианта выполняются все условия, указанные
в списке ACMG SF для гена, в котором расположен вариант.
Эти условия могут быть связаны:
- с клинической значимостью фенотипа (как уже было сказано выше, в отчёт в основном включаются варианты, ассоциированные с фенотипом только с патогенной или вероятно патогенной клинической значимостью по данным ClinVar; это условие не является обязательным только для вариантов в гене HFE);
- с типом наследования;
- с количеством выявленных в образце и подходящих под условия вариантов в одном гене из списка ACMG SF (например, для попадания в отчёт некоторых вариантов с аутосомно-рецессивным типом наследования в образце должны быть обнаружены минимум два таких варианта);
- с типом мутации (в список 1 попадают только те варианты, в суммарном происхождении аллеля которых не упоминается "соматическое");
- с генотипом (например, некоторые варианты сообщаются, только если являются гомозиготами по альтернативному аллелю);
- с конкретной нуклеотидной и аминокислотной заменой варианта.
СПИСОК 2: Варианты, которые требуют дальнейшего анализа и для которых выполняются следующие условия:
- Вариант расположен в гене из списка ACMG SF;
- Вариант ассоциирован хотя бы с одним фенотипом с патогенной и/или вероятно патогенной клинической значимостью по данным ClinVar;
- Частота аллеля по данным gnomAD 3 меньше порога частоты слишком частых патогенных вариантов, задаваемого в блоке шаблона отчёта "SNVs/Indels по генам списка ACMG SF" (рекомендуемое значение по умолчанию - 0,03), или имеет пустое значение;
- Сайт варианта содержит хотя бы один альтернативный аллель (т.е. имеет генотип 1/1, 1|1, 0/1, 1/0, 0|1 и т.п.) в случае файла VCF с единственным образцом;
- Вариант не подходит хотя бы под одно из условий Списка 1:
- вариант не ассоциирован по данным ClinVar с фенотипом, который указан в списке ACMG SF для гена, в котором расположен вариант;
- вариант ассоциирован по данным ClinVar хотя бы с одним фенотипом с доброкачественной, вероятно доброкачественной или неопределенной клинической значимостью, который указан в списке ACMG SF для гена, в котором расположен вариант;
- для варианта не выполняется хотя бы одно из условий, указанных в списке ACMG SF для гена, в котором расположен вариант.
Заметка
Для вариантов в гене TTN есть дополнительное условие на попадание в отчёт со вторичными находками: в отчёт попадают только варианты с высоким значением эффекта для генов: splice acceptor (вариант в акцепторном сайте сплайсинга), splice donor (вариант в донорном сайте сплайсинга), stop gained (вариант, приводящий к появлению стоп-кодона) или frameshift (инсерция или делеция, вызывающая сдвиг рамки считывания).
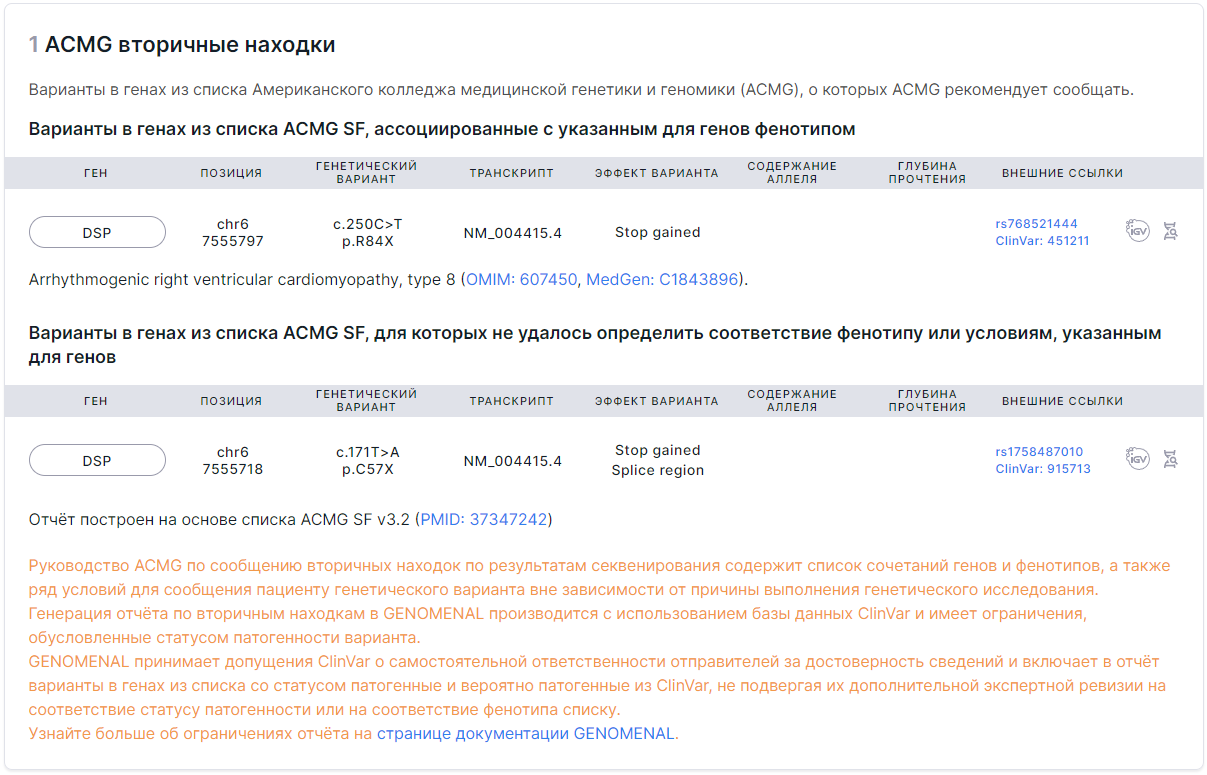
Каждый раздел содержит таблицу с вариантами со следующими колонками:
- Ген - название гена, в котором располагается вариант. При нажатии на ген появится окно со всеми транскриптами, известными для гена:
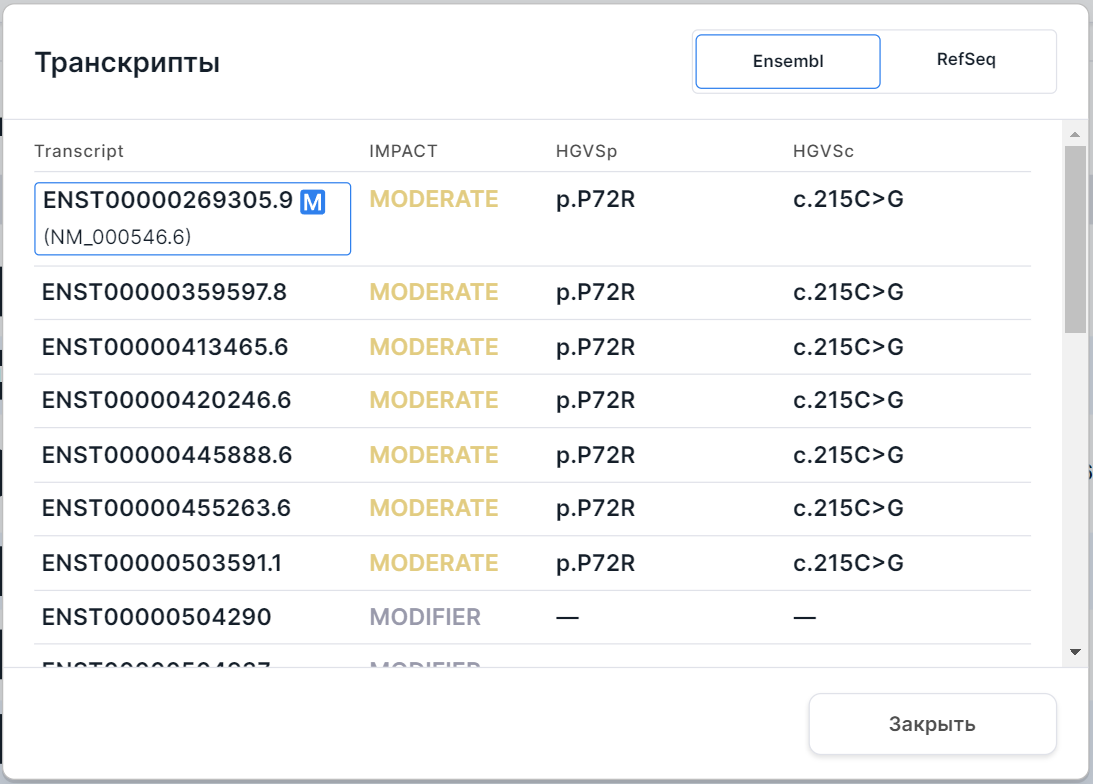
Описание колонок таблицы с транскриптами можно найти в описании раздела "Транскрипты" на панели детальной информации.
- Позиция - координаты варианта в геноме (хромосома + начальная позиция).
- Генетический вариант - нуклеотидная и аминокислотная замены в номенклатуре HGVS. Нуклеотидная: префикс “c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности) + геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель. Аминокислотная: префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены.
- Транскрипт - идентификатор основного транскрипта гена из базы RefSeq (NM_xxxxxx.x). При нажатии на транскрипт появится такое же окно со всеми транскриптами, известными для гена, как и при нажатии на поле "Ген".
- Эффект варианта - последствия варианта для генов. Подробное описание возможных значений можно посмотреть здесь.
- Содержание аллеля - частота альтернативного аллеля для данного образца (в процентах).
- Глубина прочтения - общее количество прочтений последовательности, перекрывающих позицию варианта, для данного образца.
- Внешние ссылки - ссылки на вариант в базах данных dbSNP, ClinVar и COSMIC (если база была загружена в виде пользовательской аннотации).
- Ссылки на вариант во встроенных модулях:
- модуль визуализации варианта в геноме,
- страница детальной информации о варианте в SNV Viewer (вкладка "Аннотация").
Под строкой варианта приведён фенотип, c которым вариант ассоциирован по данным ClinVar (с патогенной или вероятно патогенной клинической значимостью) и который указан в списке ACMG SF для гена, в котором расположен вариант. Для каждого фенотипа указаны ссылки на OMIM и/или MedGen.
Далее может быть приведён текст интерпретации варианта, если она была добавлена, как описано здесь.
В конце отчёта указана версия списка ACMG SF, использованного для построения отчёта.
Потенциальные находки#
Варианты, попавшие в отчёт со вторичными находками ACMG, можно использовать в качестве потенциальных находок. Для этого нужно включить соответствующий переключатель в шаблоне отчёта. В потенциальные находки можно включить варианты из Списка 1 (опция "Использовать варианты в генах из списка ACMG SF, ассоциированные с указанным для генов фенотипом, как потенциальные находки") и/или варианты из Списка 2 (опция "Использовать варианты в генах из списка ACMG SF, для которых не удалось определить соответствие фенотипу или условиям, указанным для генов, как потенциальные находки").
Ограничения отчёта, связанные с ClinVar как источником аннотации#
Построение отчёта со вторичными находками ACMG имеет ограничения по фенотипу и по патогенности, связанные с аннотацией вариантов с помощью базы данных ClinVar.
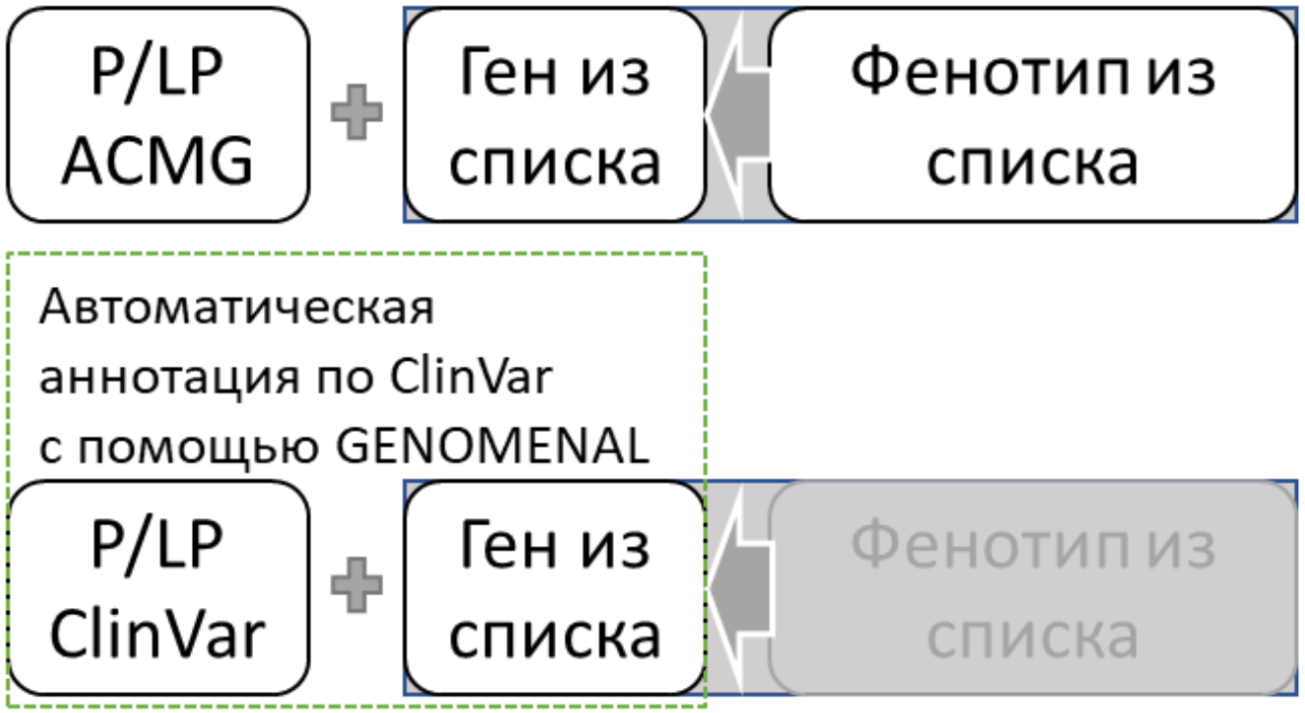
Руководство ACMG по сообщению вторичных находок по результатам секвенирования содержит список сочетаний ген-фенотип, а также ряд условий для сообщения пациенту генетического варианта вне зависимости от причины выполнения генетического исследования. Одновременно должны выполняться три условия:
- Вариант обнаружен в гене, включенном в список вторичных находок.
- Вариант ранее описан как ассоциируемый с фенотипом (состоянием, заболеванием), указанным в связке с этим геном в списке вторичных находок, или есть возможность установить связь варианта с таким фенотипом.
- Вариант должен соответствовать всем дополнительным условиям, указанным в списке вторичных находок, в число которых зачастую входит патогенная или вероятно патогенная клиническая значимость фенотипа.
Кратко эту формулу можно описать так:
Ограничения по фенотипу#
Для части вариантов, внесенных в ClinVar, невозможно извлечь данные о фенотипе (состоянии, заболевании). Отсутствие фенотипа связано с неопределенными формулировками, встречающимися в графе “Состояние” в сообщениях о статусе варианта лабораториями. Поле о фенотипе в ClinVar может содержать формулировки “не уточняется”, “не предоставлено” или быть заполнено всеми заболеваниями, которые ассоциированы с любыми патогенными вариантами в гене. В последнем случае правильный вывод о связи варианта с фенотипом может сделать только специалист. К тому же, мутации в разных участках некоторых генов могут вызывать различные наследственные заболевания, а в список вторичных находок для таких генов включены только отдельные фенотипы. Учитывая эти и другие факторы, в отчёте со вторичными находками Genomenal сообщаются два списка вариантов: те, для которых фенотип из списка вторичных находок совпадает с состоянием, указанным для вариантов в ClinVar, и те, которые расположены в гене из списка, но имеют несоответствующий фенотип.
Таким образом, к интерпретации вариантов из второго раздела отчёта нужно подходить с осторожностью.Ограничения по патогенности#
Как правило, классификация вариантов строится на критериях патогенности, рекомендованных ACMG. База данных ClinVar наполняется сообщениями лабораторий о клинической значимости вариантов с допущением, что отправители статуса вариантов описали их полно и в соответствии с рекомендациями ACMG. Genomenal принимает эти допущения и включает в отчёт варианты со статусом патогенные и вероятно патогенные из ClinVar, не подвергая их дополнительной экспертной ревизии на соответствие статусу патогенности. Дополнительно в литературе могут появляться новые доказательства влияния или отсутствия влияния вариантов на функцию гена, что может привести к пересмотру статуса варианта экспертом здесь и сейчас, но будет не учтено и не обновлено в ClinVar. Безусловно, системный пересмотр статуса вариантов является трудоемким процессом и запаздывает в сравнении с появлением новых знаний. Перечисленные факторы могут повлечь присутствие в отчёте избыточных или устаревших данных или повлечь отсутствие вариантов, для которых появились новые доказательства патогенности, ещё не отражённые в ClinVar. Некоторые варианты могут отсутствовать в отчёте, т.к. могут быть уникальными и ранее никем не задектектированными.
Таким образом в отчёт могут быть включены варианты:
- для которых неточно или ошибочно определена патогенность лабораторией, сообщающей статус варианта в ClinVar;
- патогенность которых может быть пересмотрена из-за появления новых данных (статус патогенности снизится).
В отчёт не попадут варианты:
- не зарегистрированные в ClinVar или обнаруженные у человека впервые;
- патогенность которых может быть пересмотрена из-за появления новых данных (статус патогенности повысится).
Внимание!
Используя отчёт со вторичными находками ACMG, вы соглашаетесь с тем, что понимаете указанные ограничения, и готовы обсудить результат аннотации со специалистом или с врачом, если вам потребуется разъяснение.
Экспорт отчёта#
Отчёт со вторичными находками ACMG можно скачать в формате PDF.
Для этого нажмите на кнопку в
правом верхем углу страницы отчёта.