Отчёт с мутациями, подходящими под заданные условия
Отчёт включает SNVs/Indels или вариации числа копий, выявленные в образце и соответствующие определённым условиям, которые пользователь сам может задать в шаблоне отчёта. Выбрав интересующие вас условия, вы можете настроить отчёт так, чтобы он включал мутации, наибольшим образом соответствующие типу вашего исследования. Например, вы можете наполнить отчёт клинически значимыми мутациями; мутациями, ассоциированными с конкретным фенотипом; мутации, локализованные в определённых генах, и т.п.
SNVs/Indels, подходящие под заданные условия#
Отчёт включает SNVs/Indels, выявленные в образце и соответствующие условиям, заданным в шаблоне отчёта "SNVs/Indels, подходящие под заданные условия". Составление условий для вариантов описано здесь.
SNVs/Indels, попавшие в отчёт, представлены в виде таблицы со следующими колонками:

Значок потенциальной находки
. Отображается, если в шаблоне отчёта была включена опция "Использовать SNVs/Indels, подходящие под заданные в блоке условия, как потенциальные находки". Работа с потенциальными находками описана в соответствующем разделе.
Ген - название гена, в котором располагается вариант. При нажатии на ген появится окно со всеми транскриптами, известными для гена:
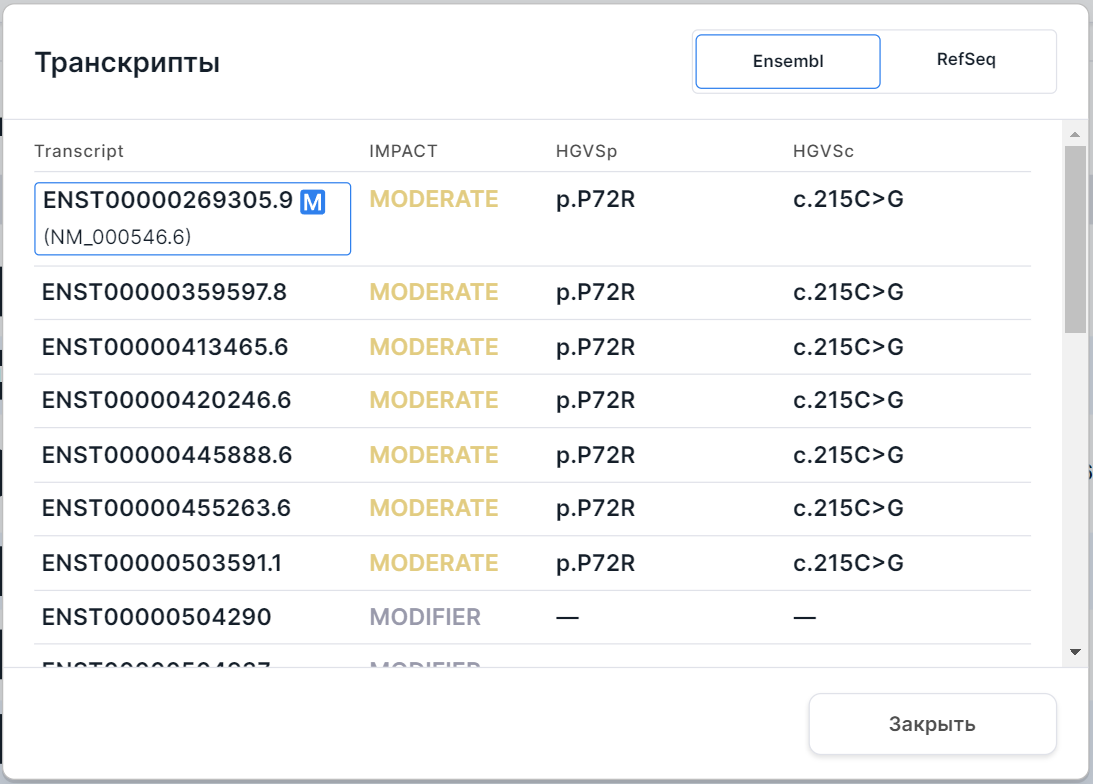
Описание колонок таблицы с транскриптами можно найти в описании раздела "Транскрипты" на панели детальной информации.
- Позиция - координаты варианта в геноме (хромосома + начальная позиция).
- Генетический вариант - нуклеотидная и аминокислотная замены в номенклатуре HGVS. Нуклеотидная: префикс “c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности) + геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель. Аминокислотная: префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены.
- Транскрипт - идентификатор основного транскрипта гена из базы RefSeq (NM_xxxxxx.x). При нажатии на транскрипт появится такое же окно со всеми транскриптами, известными для гена, как и при нажатии на поле "Ген".
- Эффект варианта - последствия варианта для генов. Подробное описание возможных значений можно посмотреть здесь.
- Содержание аллеля - частота альтернативного аллеля для данного образца (в процентах).
- Глубина прочтения - общее количество прочтений последовательности, перекрывающих позицию варианта, для данного образца.
- Внешние ссылки - ссылки на вариант в базах данных dbSNP, ClinVar и COSMIC (если база была загружена в виде пользовательской аннотации).
- Ссылки на вариант во встроенных модулях:
- модуль визуализации варианта в геноме,
- страница детальной информации о варианте в SNV Viewer (вкладка "Аннотация").
Под строкой варианта может быть приведён текст интерпретации варианта, если она была добавлена, как описано здесь.
CNVs, подходящие под заданные условия#
Отчёт включает вариации числа копий (copy number variations; CNVs), выявленные в образце и соответствующие условиям, заданным в блоке шаблона отчёта "CNVs, подходящие под заданные условия". Параметры фильтрации, доступные для составления условия на вариации, описаны здесь.
В шаблоне отчёта также нужно выбрать, вариациями какого типа региона должен наполняться отчёт: сегментами (крупными областями генома, которые получаются в результате объединения соседних бинов со схожей копийностью) или бинами (небольшими интервалами, которые создаются в результате разделения референсного генома на интервалы равного размера).
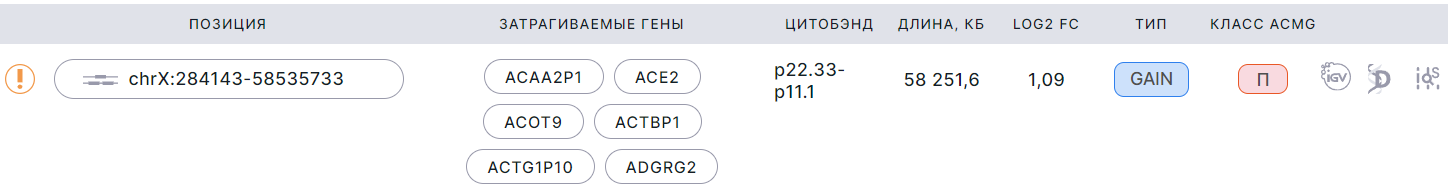
Пример сегмента в отчёте:
Пример бинов в отчёте:
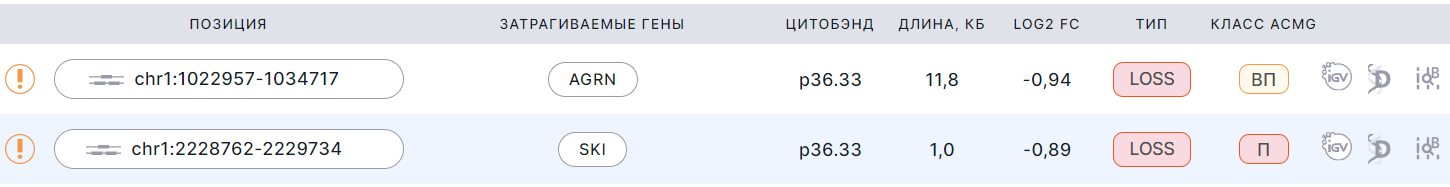
Таблица с вариациями в отчёте имеет следующие колонки:
- Значок потенциальной находки
. Отображается, если в шаблоне отчёта была включена опция "Использовать CNVs, подходящие под заданные в блоке условия, как потенциальные находки". Работа с потенциальными находками описана в соответствующем разделе.
- Позиция - координаты вариации числа копий в геноме (хромосома + начальная позиция + конечная позиция).
Позицию вариации можно скопировать в буфер обмена. Для этого наведите курсор на значение
колонки и нажмите на
.
- Затрагиваемые гены - названия генов, расположенных на затронутом вариацией участке. Нажав на ген, можно открыть его страницу в OMIM (если есть) или Ensembl.
- Цитобэнд - сокращённая запись положения вариации числа копий на хромосоме. Это положение указывается относительно полос (участков хромосом, чётко отличающихся от соседних), которые образуются на хромосомах в результате применения дифференциальных методов окрашивания. Эти полосы называются бэндами или сегментами. Район - это участок плеча хромосомы между двумя ближайшими маркерными бэндами (самыми заметными и чёткими светлыми или тёмными бэндами). Суббэнды, или субсегменты - это участки хромосом, получающиеся в результате деления бэндов при анализе хромосом с высоким разрешением. В дальнейшем суббэнды могут быть также разделены на части. Принцип обозначение бэндов (полос) на хромосоме следующий: плечо хромосомы (короткое плечо p или длинное плечо q), район, бэнд, суббэнд (может также дополнительно делиться на части). Например, запись "p.36.33" означает, что вариация находится в 3-й части 3-го суббэнда 6-го бэнда 3-го района короткого плеча хромосомы.
- Длина, кб - длина участка вариации числа копий в килобазах.
- Log2 FC - логарифмическое соотношение выявленного числа копий к нормальному числу копий (равно двум для аутосом и X-хромосомы в случае генотипа XX либо единице для половых хромосом в случае генотипа XY).
- Тип - тип изменения числа копий:
- делеция,
- дупликация.
- Класс ACMG - патогенность вариации, выставляемая соответственно категории, определённой
AnnotSV.
Значения колонки представлены
в виде значков:
- Патогенная;
- Вероятно патогенная;
- Неопределенная значимость;
- Вероятно доброкачественная;
- Доброкачественная.
При наведении курсора на значок можно посмотреть полное значение. - Переход
в модуль визуализации вариации на геноме -
.
Чтобы открыть одновременно два региона по границам достаточно крупной перестройки в IGV, нажмите на, а чтобы вернуться к визуализации всей вариации, нажмите на
. Если вы хотите перейти на страницу позиции, открытой в IGV, в геномном браузере Decipher, нажмите на
. Если вы хотите открыть CNV Viewer с фильтром по позиции, которая открыта в IGV, нажмите на кнопку
.
- Ссылка на позицию вариации
в геномном браузере Decipher -
.
- Переход к позиции вариации (с отображением детальных строк по генам
в древовидном режиме)
во встроенном модуле для просмотра и анализа вариаций числа копий CNV Viewer.
Для сегментов кнопка перехода будет выглядеть следующим
образом:
, а для бинов - следующим:
.
Экспорт отчёта#
Отчёт с вариантами или вариациями, подходящими под заданные условия, можно скачать в формате PDF.
Для этого нажмите на кнопку в
правом верхем углу страницы отчёта.