Страница детальной информации о варианте
Страница детальной информации о варианте может включать вкладки со следующей информацией:
- Подробную аннотацию варианта;
- Информацию об образце и пациенте;
- Информацию о встречаемости варианта в других образцах;
- ACMG классификацию;
- Научные публикации, ассоциированные с вариантом;
- Клинические рекомендации (если вариант является онкозначимым);
- Отчёт о варианте.
Чтобы перейти на страницу детальной информации о варианте из SNV Viewer,
нажмите на в строке варианта.
На открывшейся странице сверху приведена основная информация о варианте:
- Название гена, в котором располагается вариант;
- Нуклеотидная замена - либо в номенклатуре HGVS (HGVSc = префикс (“c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности)) + геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель), либо в виде замены (префикс "g." (genomic; для линейной геномной референсной последовательности) + начальная позиция варианта в геноме + референсный аллель > альтернативный аллель);
- Координаты варианта в геноме (хромосома + начальная позиция);
- Нуклеотидная замена (референсный аллель > альтернативный аллель);
- Идентификатор транскрипта, выбранного главным для данного варианта, из базы Ensembl (ENSTxxxxxxxxxxx);
- Номер экзона в транскрипте, который вариант затронул;
- Эффект варианта (последствия варианта для генов; см. подробно здесь);
- Ссылка на вариант в базе dbSNP.
Чтобы добавить вариант в отчёт со страницы детальной информации,
включите соответствующий переключатель  -
вариант будет включен в отчёт, и
откроется вкладка "Отчёты".
Включенные в отчёт варианты будут входить в блоки отчётов с типом
"Выбранные пользователем SNVs/Indels для добавления в отчёт".
Для исключения варианта из отчёта выключите переключатель обратно.
Также включенные в отчёт варианты можно удалить из отчёта на странице самого отчёта.
-
вариант будет включен в отчёт, и
откроется вкладка "Отчёты".
Включенные в отчёт варианты будут входить в блоки отчётов с типом
"Выбранные пользователем SNVs/Indels для добавления в отчёт".
Для исключения варианта из отчёта выключите переключатель обратно.
Также включенные в отчёт варианты можно удалить из отчёта на странице самого отчёта.
Если у варианта был определён класс патогенности, он автоматически
добавляется в результаты интерпретации образца.
Если вы хотите исключить вариант из результатов интерпретации со страницы детальной информации, отключите
соответствующий переключатель  .
.
Чтобы закрепить вариант в таблице SNV Viewer (подробно о закреплении можно
узнать здесь)
со страницы детальной информации, нажмите на . Для открепления нажмите ещё раз на ту же кнопку
.
Чтобы открыть встроенный модуль визуализации варианта в геноме (IGV),
нажмите на ![]() .
.
Чтобы закрыть страницу детальной информации о варианте,
нажмите на .
Шкала патогенности в правом верхнем углу страницы описана в соответствующем разделе:

Интерпретация варианта#
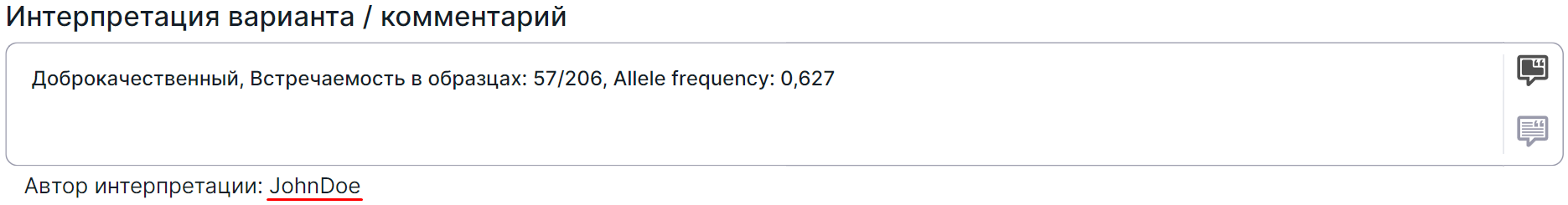
Интерпретация варианта - это комментарий с важной информацией о варианте, добавленный пользователем. То, где она будет отображаться после добавления, описано здесь. Чтобы добавить или отредактировать интерпретацию варианта, выполните следующее:
- Нажмите на окно ввода:

- Введите нужные данные или процитируйте текст со страницы детальной информации одним из следующих способов:
- Нажмите на
, а затем кликните на нужный текст - он появится в окне ввода интерпретации (удобно использовать, если вам нужен весь текст элемента, например полное название гена).
- Выделите нужный текст, а затем нажмите на
- выделенный текст появится в окне ввода интерпретации (удобно использовать, если вам нужна только определенная часть текста элемента, например одно предложение из целого резюме статьи).
После добавления интерпретации под полем ввода отобразится автор интерпретации - имя пользователя, который проинтерпретировал вариант:
Вкладка "Аннотация"#
На вкладке "Аннотация" приведены те разделы аннотации, которые включают хотя бы одно не пустое поле для варианта. Разделы аннотации соответствуют вкладкам панели детальной информации, которые подробно описаны в соответствующем разделе.
Вкладка "Образец"#
На вкладке "Образец" могут находиться два раздела:
- Информация об образце - соответствует информации об образце
со вкладки "Главное" страницы образца. Данные большинства полей
(за исключением данных о типе секвенирования и реагентах Capture Kit) можно добавить или отредактировать
как на странице образца, так и здесь, на вкладке "Образец".
Раздел содержит следующую информацию об образце:
- ТИП СЕКВЕНИРОВАНИЯ: Панель (секвенирование с помощью таргетной панели), WES (whole-exome sequencing; полноэкзомное секвенирование), WGS (whole-genome sequencing; полногеномное секвенирование), Low-pass WGS (полногеномное секвенирование с низким покрытием), UNKNOWN (тип секвенирования может быть не распознан для некоторых образцов, загруженных в формате VCF). Определяется автоматически, может быть основан на типе секвенирования и реагентах Capture Kit, выбранных на этапе создания набора образцов. Тип можно поменять на вкладке "Параметры" (параметр "Sequencing Type": опция "WGS" сооветствует типу "WGS" или "Low-pass WGS", опция "Направленный отбор" соответствует либо типу "Панель", либо "WES").
- ДАТА СБОРА ОБРАЗЦОВ - дата в формате д.М.гггг. Можно указать, нажав на поле значения.
- РЕАГЕНТЫ CAPTURE KIT - это таргетная панель, используемая при направленном отборе. Определяется автоматически Genomenal, либо самим пользователем на этапе загрузки образца. Если capture kit определен неправильно, его можно поменять на вкладке "Параметры". При анализе можно использовать как встроенные в систему реагенты capture kit (стандартные панели, часто применяемые при секвенировании), так и загруженные пользователем (на странице "Реагенты Capture Kit").
- ЛОКАЛИЗАЦИЯ ОБРАЗЦА (ОРГАН) - часть организма, из которой был взят образец для секвенирования. Можно указать, нажав на поле значения.
- МЕТОДИКА - методика выделения образца. Можно указать, нажав на поле значения.
- ОНКОЛОГИЧЕСКОЕ ЗАБОЛЕВАНИЕ, СТАДИЯ и УТОЧНЕНИЕ - онкологическое заболевание, его стадия и уточнение стадии, которые были определены у пациента на момент взятия материала образца секвенирования.
- ЛИНИЯ ТЕРАПИИ - линия терапии, используемая для лечения заболевания пациента на момент взятия материала образца секвенирования.
- РЕЗУЛЬТАТЫ ИНТЕРПРЕТАЦИИ - информация о количестве обнаруженных в образце
и включённых в результаты интерпретации мутаций
с определенным классом патогенности. Подробнее о завершении/возобновлении интерпретации образца
читайте ниже.
Раздел может включать информацию о SNVs/Indels, обозначенную "SNV",
и информацию о вариациях числа копий, обозначенную "CNV".
Информация представлена в следующем виде:
- значки патогенности с указанием количества мутаций с этой патогенностью:
- патогенные мутации:
;
- вероятно патогенные мутации:
;
- мутации неопределенной значимости:
;
- вероятно доброкачественные мутации:
;
- доброкачественные мутации:
.
- патогенные мутации:
- сообщение "Интерпретация не завершена" - если мутации с определённым классом патогенности были включены в результаты интерпретации, но интерпретация образца не была завершена.
- сообщение "Патогенных вариантов не обнаружено" - если интерпретация образца была завершена, но ни одна мутация с определённым классом патогенности не была включена в результаты интерпретации.
- пустое значение, если интерпретация образца не была завершена и ни одна мутация с определённым классом патогенности не была включена в результаты интерпретации.
- значки патогенности с указанием количества мутаций с этой патогенностью:
- Информация о пациенте - соответствует информации о пациенте
со страницы пациента. Данный раздел представлен на странице, если образец, в котором
был выявлен вариант, принадлежит определённому пациенту (а не только запуску). Данные можно добавить или
поменять как на странице пациента, так и здесь, на вкладке "Образец".
Раздел содержит следующую информацию о пациенте:
- КОД ПАЦИЕНТА - код, введенный при создании пациента (можно отредактировать, нажав на код). Может включать любые символы.
- ФАМИЛИЯ, ИМЯ пациента. Можно указать, нажав на поля значений.
- ДАТА РОЖДЕНИЯ пациента в формате д.М.гггг. Можно указать, нажав на поле значения.
- ПОЛ пациента: мужчина, женщина. Пол можно указать, нажав на поле значение и выбрав нужное значение из выпадающего списка.
- ПОСТАВЛЕННЫЙ ДИАГНОЗ - диагноз пациента, поставленный на момент взятия материала образца секвенирования. Можно указать, нажав на поля значений.
Интерпретация образца#
Здесь описан один из способов завершения интерпретации образца. Другие способы описаны здесь. Интерпретация образца - интерпретация патогенности мутаций, выявленных в образце. Чтобы добавить вариант в результаты интерпретации образца, необходимо следующее:
- Определить класс патогенности варианта (читайте подробнее в соответствующем разделе);
- Включить вариант в результаты интерпретации. Это происходит автоматически при определении класса патогенности варианта. Кроме того, включить в результаты интерпретации можно потенциальную находку, как описано здесь.
На заметку
При необходимости варианты можно добавить в результаты интерпретации образца уже после завершения интерпретации.
Чтобы завершить интерпретацию,
нажмите на  на вкладке "Образец"
страницы детальной информации о варианте. Вы увидите окно подтверждения. Для неопухолевых образцов оно выглядит так:
на вкладке "Образец"
страницы детальной информации о варианте. Вы увидите окно подтверждения. Для неопухолевых образцов оно выглядит так:
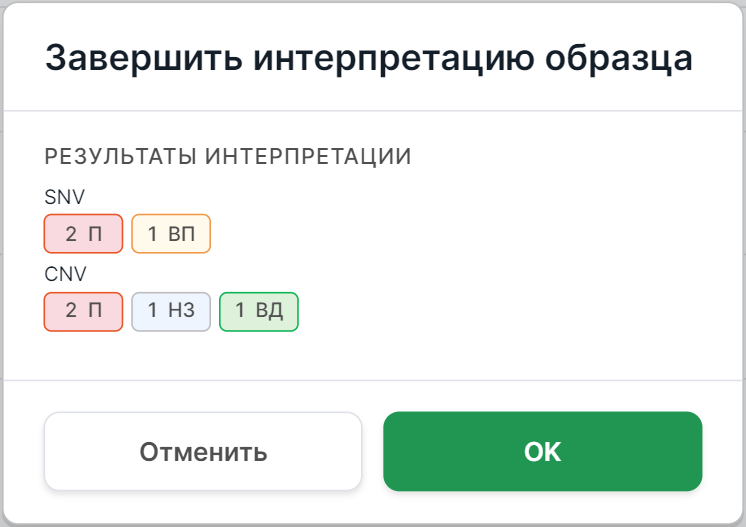
А для опухолевых образцов также включает поля об онкологическом заболевании:

В поле "Результаты интерпретации" приведена информация о количестве обнаруженных в образце и включённых в результаты интерпретации мутаций с определенным классом патогенности. Поле может включать информацию о SNVs/Indels, обозначенную "SNV", и информацию о вариациях числа копий, обозначенную "CNV". Информация представлена в виде значков патогенности с указанием количества мутаций с этой патогенностью:
- патогенные мутации:
;
- вероятно патогенные мутации:
;
- мутации неопределенной значимости:
;
- вероятно доброкачественные мутации:
;
- доброкачественные мутации:
.
Если никакие мутации с определённым классом патогенности не были включены в результаты интерпретации, то значением поля будет "Патогенных вариантов не обнаружено".
Для вариантов, обнаруженных в опухолевых образцах, также можно указать онкологическое заболевание, его стадию и уточнение стадии, которые были определены у пациента на момент взятия материала образца секвенирования, а также линию терапии, используемую для лечения заболевания пациента на момент взятия материала образца секвенирования.
Для подтверждения завершения интерпретации
нажмите .
Для возобновления завершённой интерпретации
нажмите на  .
.
Вкладка "Встречаемость"#
Среди других образцов, загруженных пользователем в систему, или групповых анализов, созданных пользователем, можно найти информацию о встречаемости следующих вариантов:
- таких же вариантов, как и вариант, страница с деталями которого открыта. Найденные варианты полностью совпадают с искомым вариантом по типу мутации (соматический или герминальный), хромосоме, стартовой позиции, референсному и альтернативному аллелю и гену. Сайты найденных вариантов содержат хотя бы один альтернативный аллель (т.е. имеют генотип 1/1, 1|1, 0/1, 1/0, 0|1, 1|0, 1/., 1|., ./1, .|1, 1, 0/0/1, 0/2, 1/2 и пр.)
- всех вариантов в той же позиции, что и вариант, страница с деталями которого открыта. Найденные варианты содержат хотя бы один альтернативный аллель и совпадают с искомым вариантом по типу мутации, хромосоме и стартовой позиции.
- вариантов в позиции ± 10 п.н. или ± 20 п.н. от стартовой позиции варианта, страница с деталями которого открыта. Найденные варианты совпадают с данным вариантом по типу мутации, а их стартовые позиции находятся в пределах интервала ± 10 п.н. или ± 20 п.н. от стартовой позиции данного варианта. Сайты найденных вариантов содержат хотя бы один альтернативный аллель.
Справа от кнопок переключения поиска описанных выше вариантов находится информация о количестве таких вариантов, найденных в других образцах:

При подсчете образцов учитываются все когда-либо загруженные в систему образцы (включая удаленные), для которых была успешно завершена стадия выявления вариантов (для образцов, загруженных в формате FASTQ или BAM) или стадия аннотации вариантов (для образцов, загруженных в формате VCF, TXT или TSV). Таким образом, для подсчета встречаемости соматического варианта учитываются те образцы, загруженные в формате FASTQ или BAM, у которых успешно завершена стадия "Выявление соматических SNVs/Indels", и те образцы, загруженные в формате VCF, TXT или TSV, у которых успешно завершена стадия "Аннотация соматических SNVs/Indels" а для подсчета встречаемости герминального варианта - образцы в формате FASTQ или BAM с успешно завершенной стадией "Выявление герминальных SNVs/Indels" и образцы в формате VCF, TXT или TSV с успешно завершенной стадией "Аннотация герминальных SNVs/Indels".
Далее приведены карточки с найденными вариантами. Пример карточки:
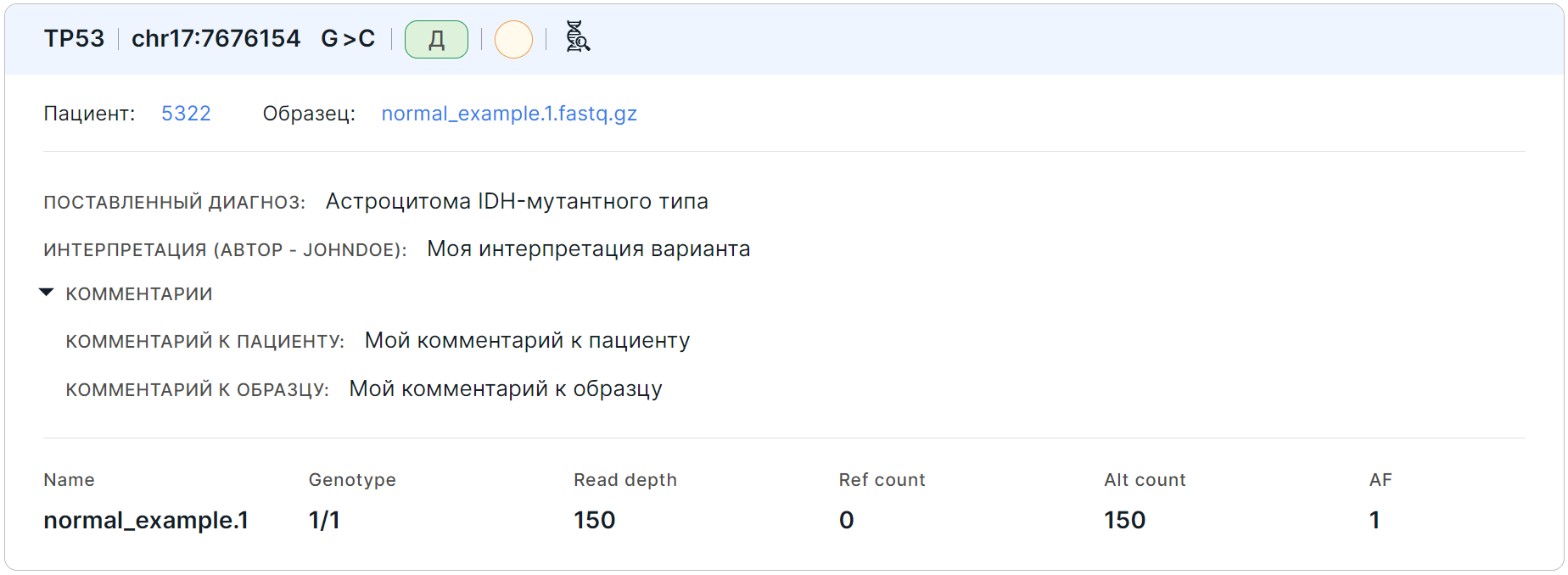
В карточке для варианта приведены:
- название гена, в котором располагается вариант;
- позиция варианта: хромосома + стартовая позиция;
- замена: референсный аллель > альтернативный аллель;
- патогенность (если была определена
для этого варианта; если значение патогенности
требует подтверждения, то рядом со значком
патогенности будет значок
);
- значимость (если была определена пользователем);
- ссылка на страницу детальной информации о
найденном варианте:
(если образец, в котором был выявлен вариант, удален, то ссылки на вариант не будет);
- ссылки на пациента и/или запуск, в которые загружен образец с вариантом (если пациент и/или запуск удалены, то вместо ссылок будут указаны код пациента или название запуска с пометкой об удалении);
- ссылка на образец, в котором был выявлен вариант (если образец удален, то вместо ссылки будет указано имя файла образца с пометкой об удалении);
- диагноз пациента, поставленный на момент взятия материала образца секвенирования (может быть определен на странице пациента или на вкладке "Образец" страницы детальной информации о варианте);
- интерпретация варианта (проинтерпретировать вариант можно на странице или на панели детальной информации о варианте; в скобках указано имя пользователя, который проинтерпретировал вариант);
- раздел с комментариями - отображается только при наличии хотя бы одного из комментариев: комментария к пациенту, описания запуска или комментария к образцу;
- информация об образцах секвенирования:
- Name - имя образца, для которого проводился анализ.
- Genotype - генотип, комбинация аллелей для данного образца (0 - референсный аллель, 1 - первый альтернативный аллель, 2 - второй альтернативный аллель и т.д.), разделенных ”/” (для нефазированного генотипа) или “|” (для фазированного генотипа). Количество аллелей говорит о плоидности образца.
- Read depth - глубина секвенирования, общее количество прочтений последовательности, перекрывающих позицию варианта, для данного образца.
- Ref count - количество раз, когда в последовательности считывался референсный аллель для данного образца.
- Alt count - количество раз, когда в последовательности считывался альтернативный аллель для данного образца.
- AF - частота альтернативного аллеля для данного образца.
В конце страницы приведена информация о встречаемости вариантов в групповых анализах, созданных пользователем:
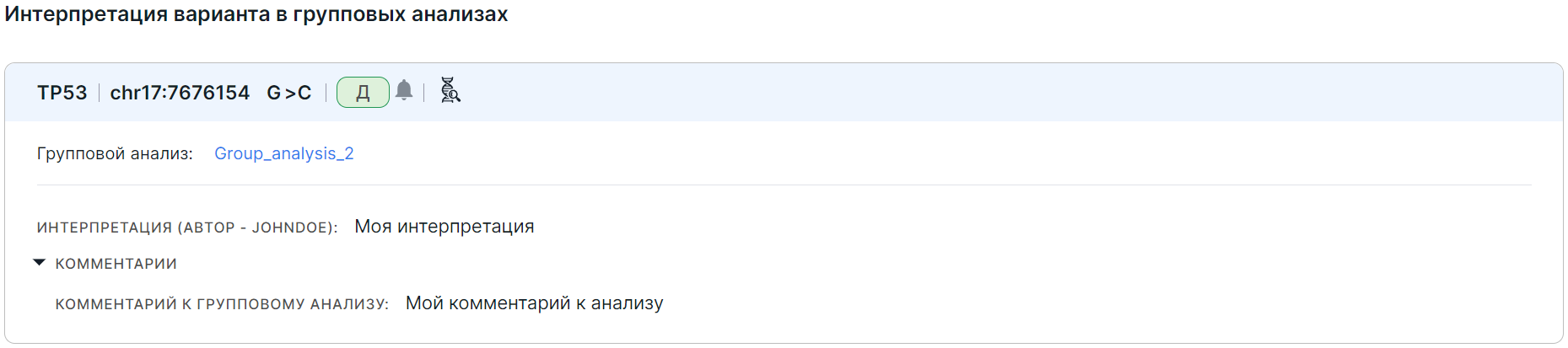
Здесь также приведены карточки вариантов со следующей информацией:
- название гена, в котором располагается вариант;
- позиция варианта: хромосома + стартовая позиция;
- замена: референсный аллель > альтернативный аллель;
- патогенность (если была определена
для этого варианта; если значение патогенности требует подтверждения, то рядом со значком
патогенности будет значок
);
- значимость (если была определена пользователем);
- ссылка на страницу детальной информации о
найденном варианте:
(если групповой анализ с вариантом был удален, то ссылки на вариант не будет);
- ссылка на групповой анализ, в котором был обнаружен вариант (если групповой анализ удален, то вместо ссылки будет указано имя группового анализа с пометкой об удалении);
- интерпретация варианта (проинтерпретировать вариант можно на странице или на панели детальной информации о варианте; в скобках указано имя пользователя, который проинтерпретировал вариант);
- комментарий к групповому анализу (если был добавлен на странице анализа).
Вкладка "ACMG Классификация"#
На вкладке представлены критерии для интерпретации вариантов, разработанные ACMG. Подробно вкладка описана в соответствующем разделе документации.
Вкладка "Литература"#
На вкладке приводятся найденные в NCBI LitVar научные публикации, ассоциированные с вариантом. Поиск вариантов в базе может занять какое-то время, поэтому впервые открыв вкладку "Литература" для варианта, вы можете увидеть сообщение "Ищем литературу в LitVar. Это может занять около минуты." В таком случае просто подождите, пока литература загрузится. Если литература для варианта не была найдена в LitVar, вы увидите сообщение "Литература не найдена в LitVar. Вы можете: Добавить PMID или Искать в Google Scholar." (о добавлении публикации по PMID и поиске в Google Scholar см. ниже).
Пример карточки статьи, найденной для варианта TP53:c.215C>G:

Для каждой статьи приведены журнал, дата публикации, ссылка на статью в PubMed
(PMID), название, авторы и резюме статьи. Статью можно добавить в курированные (избранные) статьи,
нажав на . Затем можно отфильтровать статьи и посмотреть только курированные, нажав на
. Чтобы удалить статью из курированного, нажмите на
.
Для поиска определенной публикации среди найденных статей воспользуйтесь окном поиска.
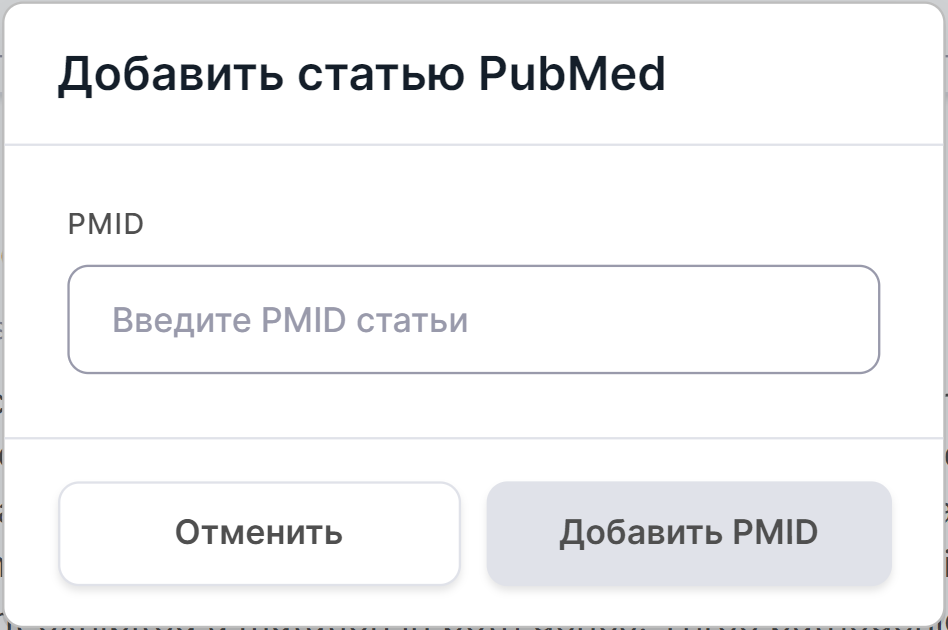
Вы можете добавить недостающую публикацию в список. Для этого:
- Нажмите на
;
- В появившемся окне введите PMID публикации и
нажмите
:
Для поиска дополнительных публикаций, ассоциированных с вариантом, можно воспользоваться
Google Scholar. Для этого
нажмите на  .
.
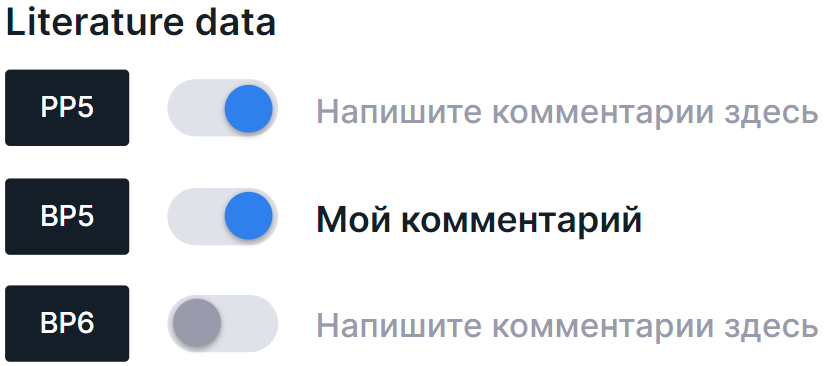
Связывание публикации с критерием ACMG#
Публикацию можно связать с критерием/критериями ACMG. Описание критериев можно посмотреть в соответствующем разделе. Для связывания нажмите на карточку статьи, а затем на переключатель нужного критерия. Здесь же можно оставить комментарий:
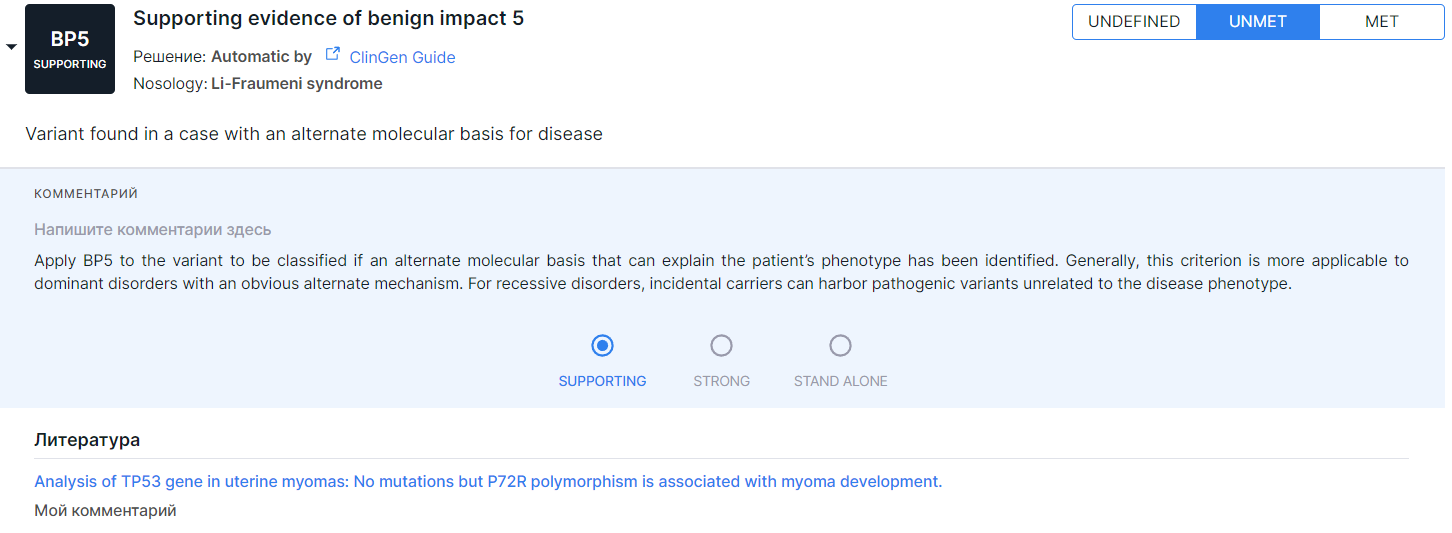
После связывания публикации с критерием ACMG она автоматически добавляется в курированные (избранные) . Если убрать все связи публикации с критериями (выключить все переключатели), то она автоматически удалится из курированных. Прикрепленные к критерию публикация (активная ссылка на статью в PubMed) и комментарий будут расположены в разделе "Литература" (у развернутого критерия на вкладке "ACMG Классификация"):
Вкладка "Клинические рекомендации"#
Вкладка есть на странице детальной информации о варианте, только если он является онкозначимым для немелкоклеточного рака легкого (НМРЛ), меланомы, рака поджелудочной железы, рака молочной железы (РМЖ), рака яичников, рака простаты, анапластического рака щитовидной железы или колоректального рака. На вкладке представлены клинические рекомендации, разработанные RUSSCO, АОР, КР МЗ РФ, NCCN, ESMO для пациентов с определенными онкологическими заболеваниями (с которыми ассоциирован вариант), их стадиями с уточнениями и линиями терапии. Подробно вкладка описана в соответствующем разделе документации.
Вкладка "Отчёты"#
На вкладке можно включить вариант в отчёт, посмотреть, как информация о нём будет выглядеть в отчёте, и экспортировать отчёт с этим вариантом.
Чтобы добавить вариант в отчёт со вкладки "Отчёты" страницы детальной информации,
включите соответствующий переключатель  .
Включенные в отчёт варианты будут входить в блоки отчётов с типом
"Выбранные пользователем SNVs/Indels для добавления в отчёт".
Для исключения варианта из отчёта выключите переключатель обратно.
Также включенные в отчёт варианты можно удалить из отчёта на странице самого отчёта.
.
Включенные в отчёт варианты будут входить в блоки отчётов с типом
"Выбранные пользователем SNVs/Indels для добавления в отчёт".
Для исключения варианта из отчёта выключите переключатель обратно.
Также включенные в отчёт варианты можно удалить из отчёта на странице самого отчёта.
После добавления варианта в отчёт станет доступен предварительный просмотр того, как вариант будет выглядеть в отчёте:
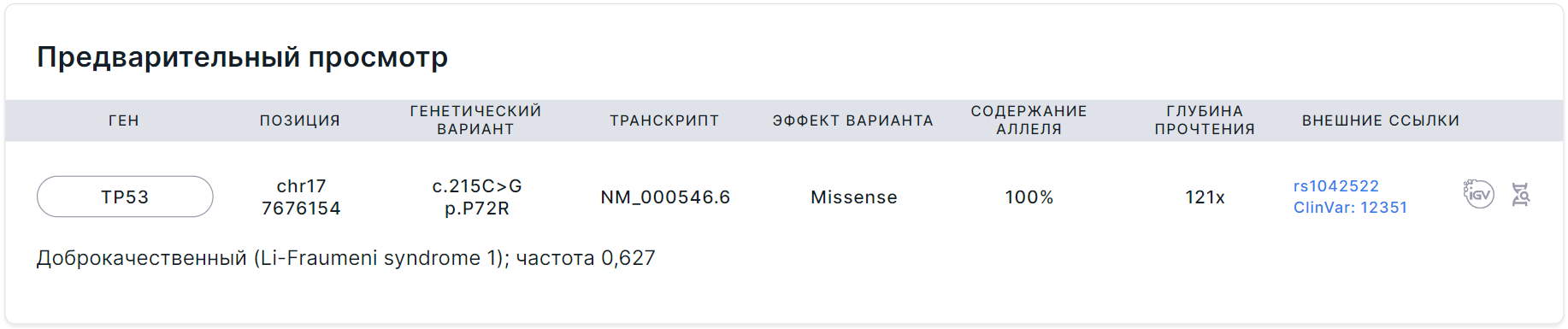
В отчёте вариант отображается в таблице со следующими колонками:
- Ген - название гена, в котором располагается вариант. При нажатии на ген появится окно со всеми транскриптами, известными для гена:
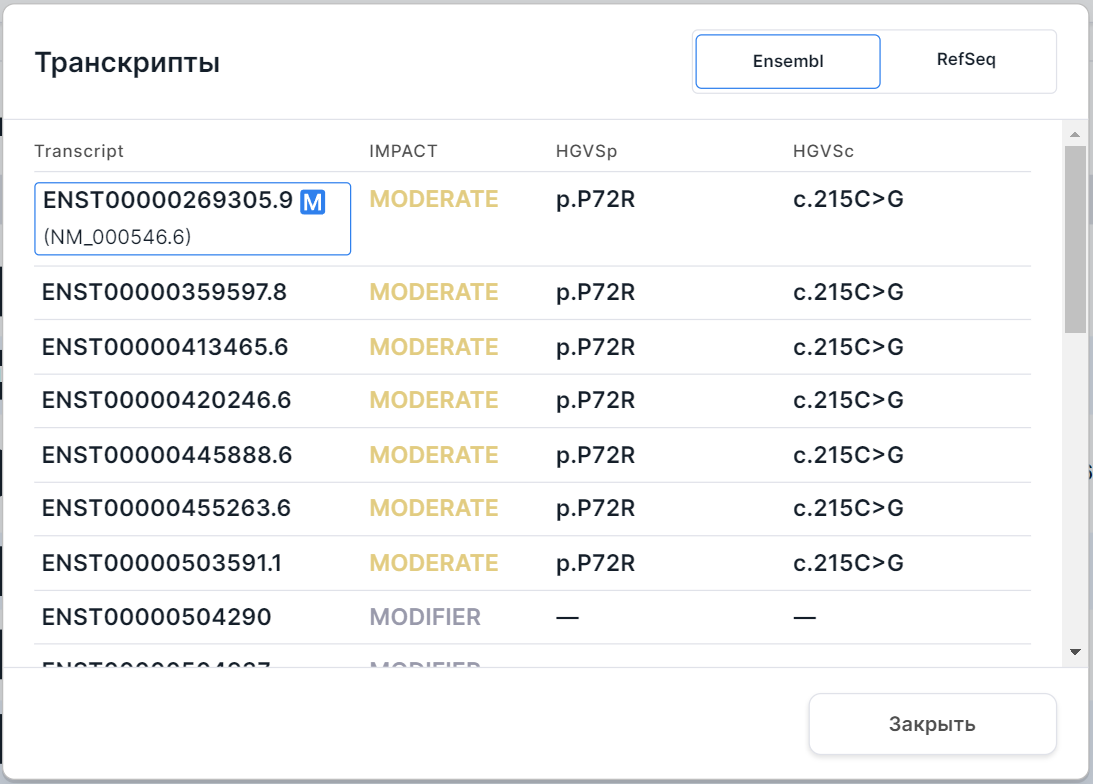
Описание колонок таблицы с транскриптами можно найти в описании раздела "Транскрипты" на панели детальной информации.
- Позиция - координаты варианта в геноме (хромосома + начальная позиция).
- Генетический вариант - нуклеотидная и аминокислотная замены в номенклатуре HGVS. Нуклеотидная: префикс “c.” (coding; для замены в кодирующей последовательности) или “n.” (non-coding; для замены в некодирующей последовательности) + геномная позиция замещенного нуклеотида + референсный аллель > альтернативный аллель. Аминокислотная: префикс “p.” (protein) + референсная аминокислота + позиция аминокислоты в белке + новая аминокислота, получившаяся в результате замены.
- Транскрипт - идентификатор основного транскрипта гена из базы RefSeq (NM_xxxxxx.x). При нажатии на транскрипт появится такое же окно со всеми транскриптами, известными для гена, как и при нажатии на поле "Ген".
- Эффект варианта - последствия варианта для генов. Подробное описание возможных значений можно посмотреть здесь.
- Содержание аллеля - частота альтернативного аллеля для данного образца (в процентах).
- Глубина прочтения - общее количество прочтений последовательности, перекрывающих позицию варианта, для данного образца.
- Внешние ссылки - ссылки на вариант в базах данных dbSNP, ClinVar и COSMIC (если база была загружена в виде пользовательской аннотации).
- Ссылки на вариант во встроенных модулях:
- модуль визуализации варианта в геноме,
- страница детальной информации о варианте в SNV Viewer (вкладка "Аннотация").
Под строкой варианта в таблице может быть интерпретация варианта, добавленная, как описано выше.
Чтобы экспортировать отчёт с вариантом в формате PDF,
нажмите .