Шаблоны отчётов
Если вы хотите, чтобы результаты анализа образца были представлены в виде структурированного отчёта с нужной вам информацией, вы можете создать, импортировать или отредактировать шаблон нужного отчёта на странице "Шаблоны отчётов".
На заметку
Количество и вид шаблонов на странице "Шаблоны отчётов" зависит от первоначальных настроек, сделанных администратором Системы.
Чтобы открыть раздел с шаблонами отчётов, перейдите на страницу "Шаблоны отчётов" из блока страниц "Настройки" через панель навигации слева:
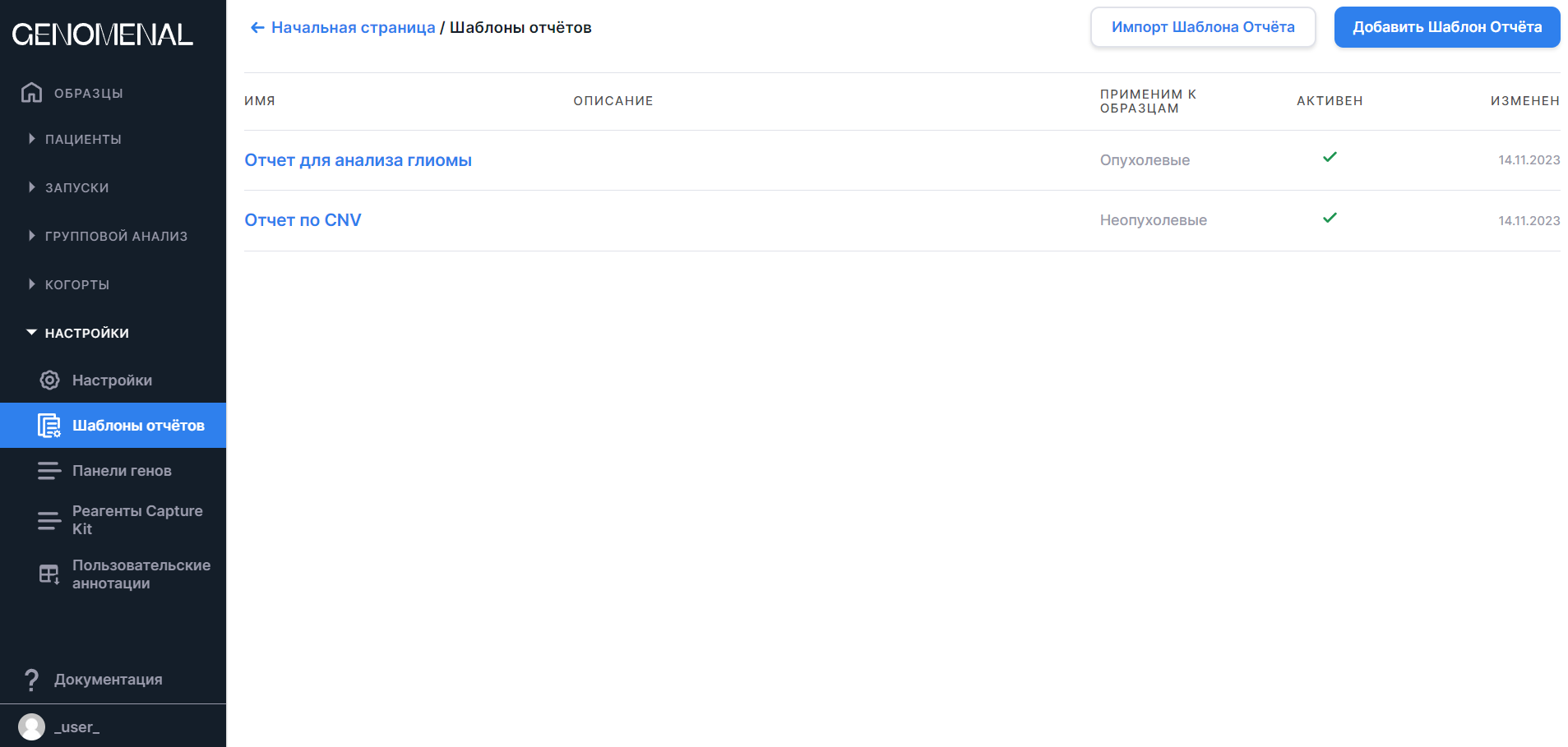
Добавление шаблона отчёта#
Нажмите на  , чтобы открыть страницу
добавления шаблона отчёта:
, чтобы открыть страницу
добавления шаблона отчёта:
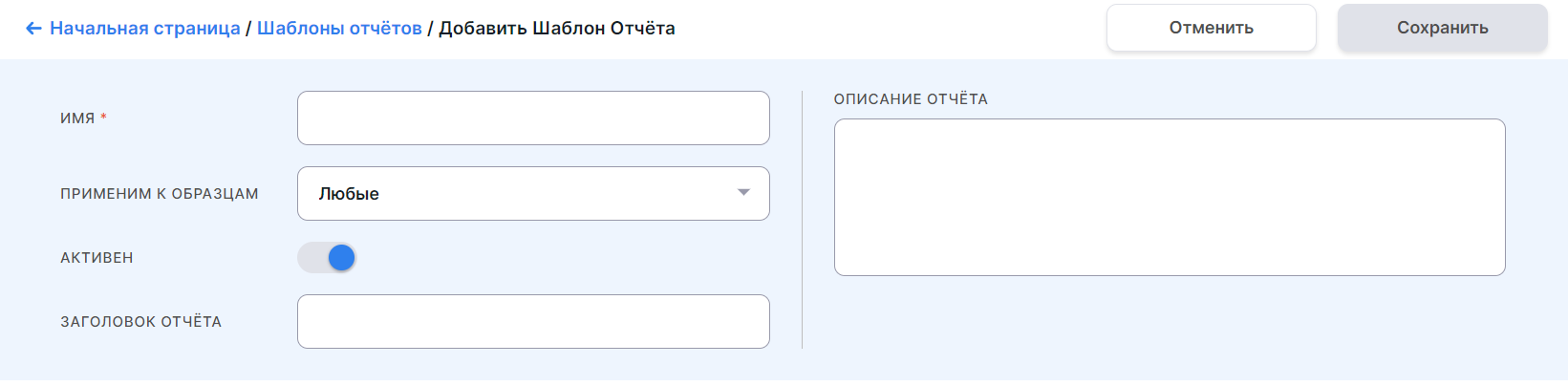
На странице можно заполнить следующие поля:
- Имя отчёта (дает название вкладке с отчётом на странице образца). Обязательно к заполнению.
- Применим к образцам - тип образцов (Любые, Опухолевые или Неопухолевые), к которым будет применим данный шаблон.
Внимание!
Значение поля "Применим к образцам" нельзя будет поменять после сохранения шаблона. Выбирайте нужный тип на этапе добавления. Значения всех остальных полей можно поменять в любой момент.
- Активен: если переключатель включен
, то шаблон будет активным, т.е. будет применен ко всем образцам с типом, соответствующим выбранному в поле "Применим к образцу"; если переключатель выключен
, то шаблон будет неактивным, т.е. не будет применен ни к одному загружаемом образцу.
- Заголовок отчёта.
- Описание отчёта (отображается под заголовком на странице отчёта).
Заполните все необходимые поля и нажмите
на , чтобы сохранить шаблон. Если вы покинете страницу, нажав на
, или
любым другим образом, шаблон не будет сохранен. Страница сохраненного шаблона выглядит так:
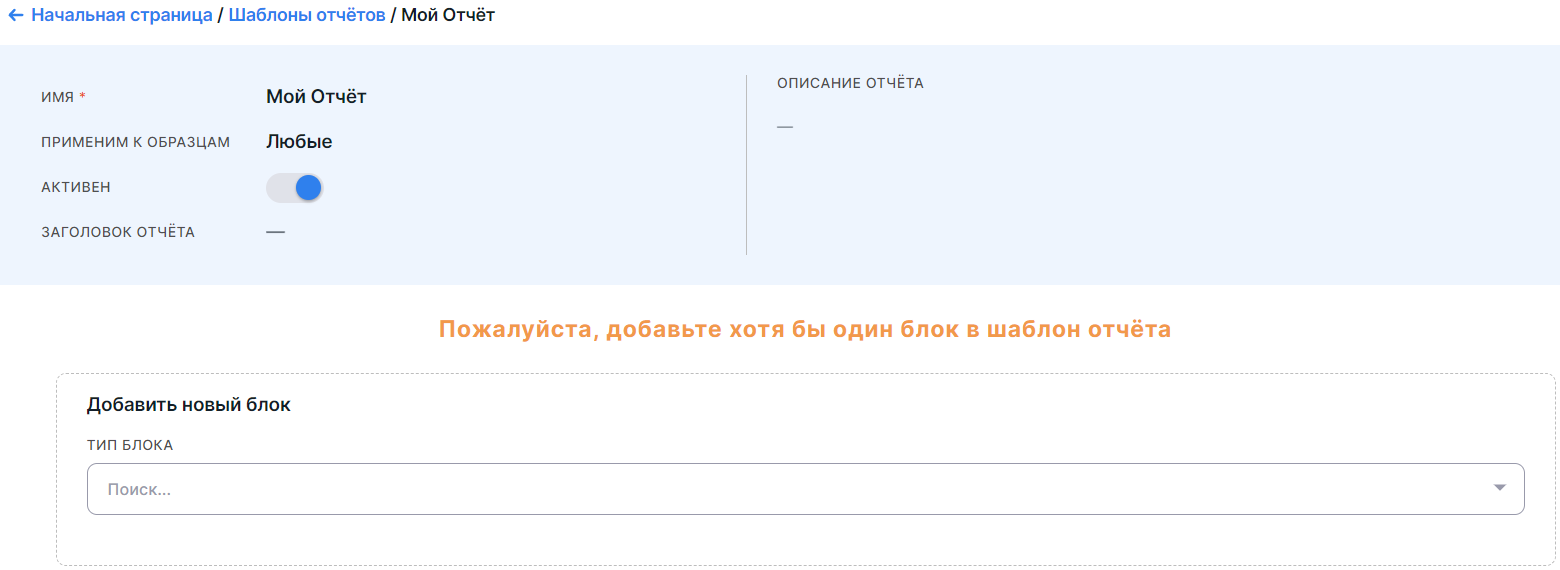
Для того чтобы шаблон был активен, в него необходимо добавить хотя бы один блок (помимо включенной опции "Активен").
Добавление блока в шаблон отчёта#
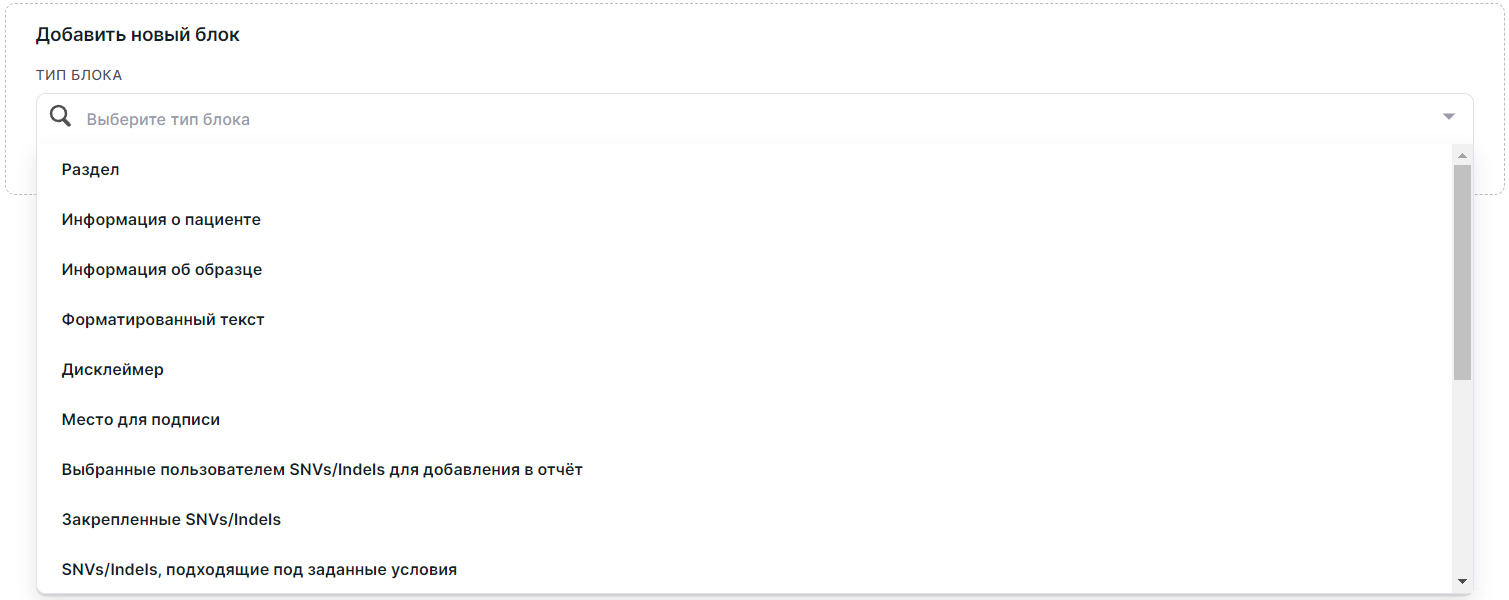
Для добавления блока в шаблон отчёта нажмите на окно поиска и выберите из появившегося списка нужный блок (типы блоков описаны ниже):
После выбора типа блока заполните все необходимые поля и
нажмите . Для отмены - нажмите
.
Типы блоков#
Каждый из нижеописанных блоков доступен для добавления в шаблон отчёта и сопровождается описанием его особенностей.
1. Раздел#
Блок с заголовком и описанием раздела отчёта.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно). При наличии нескольких разделов в отчёте они нумеруются автоматически.
- Описание – текст, отображаемый под заголовком.
2. Информация о пациенте#
Блок с информацией о пациенте, с которым связан образец.
Применим к образцам: любые, опухолевые, неопухолевые.
Отображаемые поля: отметьте галочкой поля, которые будут отображены в отчёте. После выбора всех нужных полей кликните вне области редактирования, чтобы сохранить изменения.
- Код пациента (Patient ID);
- Имя (First name);
- Фамилия (Last name);
- Отчество (Middle name);
- Дата рождения (Date of birth);
- Возраст (Age);
- Пол (Sex);
- Поставленный диагноз (Diagnosis);
- Дата постановки диагноза (Date of diagnosis);
- Комментарии (Comments).
Пример блока в отчёте: раздел с информацией о пациенте.
3. Информация об образце#
Блок с информацией об исследуемом образце, для которого построился отчёт.
Применим к образцам: любые, опухолевые, неопухолевые.
Отображаемые поля: отметьте галочкой поля, которые будут отображены в отчёте. После выбора всех нужных полей кликните вне области редактирования, чтобы сохранить изменения.
- Имя образца (Sample name) - название файла образца без расширения;
- Тип образца (Sample type);
- Тип секвенирования (Sequencing type);
- Дата сбора образцов (Sample collection date);
- Реагенты capture kit (Capture kit);
- Локализация образца, орган (Sample location);
- Методика (Methodology);
- Онкологическое заболевание (Oncological disease) - онкологическое заболевание, которое было определено у пациента на момент взятия материала образца секвенирования (НМРЛ - немелкоклеточный рак легкого; меланома; рак поджелудочной железы; РМЖ - рак молочной железы; рак яичников; рак простаты; анапластический рак щитовидной железы; колоректальный рак);
- Стадия онкологического заболевания (Onco stage);
- Уточнение стадии онкологического заболевания (Onco stage details);
- Линия терапии (Therapy line);
- Комментарии (Comments).
Пример блока в отчёте: раздел с информацией об образце.
4. Информация о контрольном (неопухолевом) образце#
Блок с информацией о неопухолевом образце, соответствующем опухолевому из пары опухоль/контроль. Строится только для опухолевого образца.
Применим к образцам: опухолевые.
Отображаемые поля: те же поля, что и в блоке "Информация об образце". Отметьте галочкой поля, которые будут отображены в отчёте. После выбора всех нужных полей кликните вне области редактирования, чтобы сохранить изменения.
Пример блока в отчёте: раздел с информацией о неопухолевом образце.
5. Форматированный текст#
Блок с любым текстом в формате Markdown.
Применим к образцам: любые, опухолевые, неопухолевые.
В текст можно вставить определенные автополя, их описание приведено ниже.
6. Дисклеймер#
Блок с сообщением об ограничении использования отчёта.
Применим к образцам: любые, опухолевые, неопухолевые.
Текст - стандратный текст заявления об отказе от клинической ответственности. Его можно отредактировать. В текст можно вставить определенные автополя, их описание приведено ниже.
Автополя в блоках "Форматированный текст" и "Дисклеймер"#
Автополе - это определенный маркер в фигурных скобках, который, если вставить его в текст блока шаблона,
будет автоматически заполняться определенным значением в готовом отчёте. Например, если вставить в текст блока
шаблона {url}, то в тексте отчёта на месте этого автополя появится URL-адрес текущей страницы (по сути это будет
ссылка на страницу образца). Значения в уже готовом отчёте будут меняться по мере изменения соответствующих данных о
пациенте или образце, для их обновления перезапускать генерацию отчёта не нужно.
Список возможных автополей и их значений:
{url}- адрес текущей страницы;{ReportName}- имя отчёта (задается на странице шаблона отчёта);{patient.code}- код пациента (задается при создании пациента, может быть изменен на странице пациента);{patient.firstName}- имя пациента (задается при создании пациента или на странице пациента);{patient.middleName}- отчество пациента (задается на странице пациента);{patient.lastName}- фамилия пациента (задается при создании пациента или на странице пациента);{patient.dateOfBirth}- дата рождения пациента (задается на странице пациента);{patient.age}- возраст пациента (определяется на странице пациента);{patient.sex}- пол пациента (задается на странице пациента);{patient.diagnosis}- поставленный пациенту диагноз (задается на странице пациента);{patient.dateOfDiagnosis}- дата постановки диагноза пациента (задается на странице пациента);{patient.comments}- комментарии к карточке пациента (добавляются на странице пациента);{sample.sampleName}- название образца (имя файла без расширения; может быть изменено на странице образца);{sample.sampleType}- тип образца: NORMAL или TUMOR (выбирается при загрузке образца, может быть изменен в окне редактирования набора образцов или при выборе другого типа в строке образца для запусков);{sample.sequencingType}- тип секвенирования образца (определяется автоматически, может быть основан на типе секвенирования и реагентах Capture Kit, выбранных на этапе создания набора образцов, может быть изменен на вкладке "Параметры" образца);{sample.sampleCollectionDate}- дата сбора образцов (задается на странице образца);{sample.captureKit}- реагенты Capture Kit (определяются автоматически или на этапе загрузки образца, могут быть изменены на вкладке "Параметры" образца);{sample.methodology}- методика выделения образца (задается на странице образца);{sample.oncologicalDisease}- онкологическое заболевание, которое было определено у пациента на момент взятия материала образца секвенирования (задается на странице образца);{sample.oncoStage}- стадия онкологического заболевания, которая была определена у пациента на момент взятия материала образца секвенирования (задается на странице образца);{sample.oncoStageDetails}- уточнение стадии онкологического заболевания, которое было определено у пациента на момент взятия материала образца секвенирования (задается на странице образца);{sample.therapyLine}- линия терапии, используемая для лечения заболевания пациента на момент взятия материала образца секвенирования (задается на странице образца);{sample.comments}- комментарии к образцу (добавляются на странице образца).
СОВЕТ
По правилам Markdown для перехода на новую строку в тексте используйте два пробела.
7. Место для подписи#
Блок с местом для подписи врача, которое будет выглядеть следующим образом в отчёте:
Применим к образцам: любые, опухолевые, неопухолевые.
8. Выбранные пользователем SNVs/Indels для добавления в отчёт#
Блок с вариантами, включенными в отчёт на странице SNV Viewer. Включение вариантов в отчёт описано в соответствующем разделе.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Тип мутации: соматическая или герминальная.
9. Закрепленные SNVs/Indels#
Блок с вариантами, закрепленными в таблице SNV Viewer. Закрепление вариантов описано в соответствующем разделе.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Тип мутации: соматическая или герминальная.
10. SNVs/Indels, подходящие под заданные условия#
Блок с вариантами, которые подходят под определенные условия, заданные в нём.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Тип мутации: соматическая или герминальная.
- Условия для включения вариантов в отчёт: если варианты, выявленные в образце, подойдут
под данные условия, то они отобразятся в этом блоке отчёта. Чтобы составить условие,
нажмите
. Откроется конструктор запросов, который полностью совпадает с формой для расширенного поиска вариантов на странице SNV Viewer. Составление условия в конструкторе запросов описано в соответствующем разделе.
- Использование SNVs/Indels в качестве потенциальных находок: чтобы включить SNVs/Indels, выявленные в образце и подходящие под заданные условия, в потенциальные находки, активируйте переключатель.
Пример блока в отчёте: отчёт с мутациями, подходящими под заданные условия.
11. CNVs, подходящие под заданные условия#
Блок с вариациями числа копий (CNVs), которые подходят под определенные условия, заданные в нём.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Тип регионов CNV: сегмент или бин. Подробнее про сегменты и бины читайте здесь.
- Условия для включения вариаций в отчёт: если вариации числа копий, выявленные в образце, удовлетворяют
заданным условиям, то они отображаются в этом блоке отчёта. Чтобы составить условие,
нажмите
 . Откроется
окно с параметрами фильтрации вариаций числа копий,
описанными в соответствующем разделе:
. Откроется
окно с параметрами фильтрации вариаций числа копий,
описанными в соответствующем разделе:
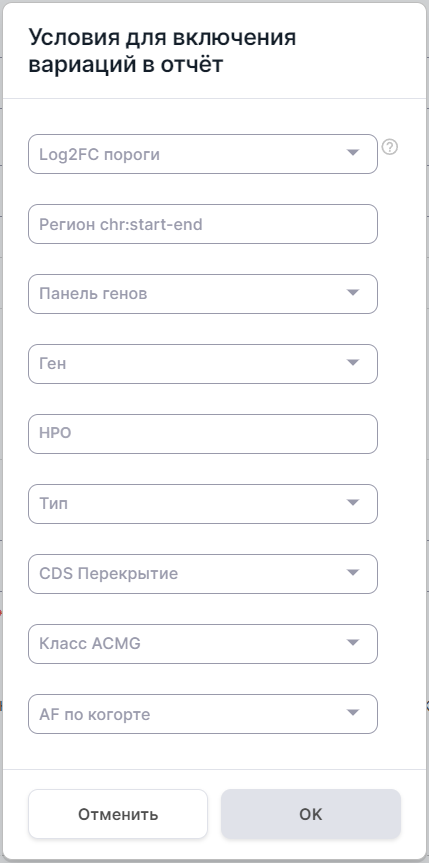
Подробнее о Log2FC порогах
Фильтр вариаций по строгости, а именно порогам log2FC - логарифмического соотношения выявленного числа копий к нормальному числу копий, имеет следующие возможные значения:
- Del: ≤-1, Dup: ≥0.58 (chrX: ≥1, chrY: ≥1) (XY) Del: ≤-1 (chrX: ≤0), Dup: ≥0.58 (chrX: ≥1.58, chrY: ≥0) (XX)
- Del: ≤-0.9 (chrX: ≤-3.8, chrY: ≤-3.8), Dup: ≥0.55 (chrX: ≥0.94, chrY: ≥0.94) (XY) Del: ≤-0.9 (chrX: ≤0.1), Dup: ≥0.55 (chrX: ≥1.55, chrY: ≥-0.1) (XX)
- Del: ≤-0.8 (chrX: ≤-2.74, chrY: ≤-2.74), Dup: ≥0.51 (chrX: ≥0.88, chrY: ≥0.88) (XY) Del: ≤-0.8 (chrX: ≤0.2), Dup: ≥0.51 (chrX: ≥1.51, chrY: ≥-0.23) (XX)
- Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82) (XY) Del: ≤-0.7 (chrX: ≤0.3), Dup: ≥0.46 (chrX: ≥1.46, chrY: ≥-0.37) (XX)
- Del: ≤-0.6 (chrX: ≤-1.64, chrY: ≤-1.64), Dup: ≥0.42 (chrX: ≥0.74, chrY: ≥0.74) (XY) Del: ≤-0.6 (chrX: ≤0.4), Dup: ≥0.42 (chrX: ≥1.42, chrY: ≥-0.55) (XX)
- Del: ≤-0.5 (chrX: ≤-1.27, chrY: ≤-1.27), Dup: ≥0.37 (chrX: ≥0.66, chrY: ≥0.66) (XY) Del: ≤-0.5 (chrX: ≤0.5), Dup: ≥0.37 (chrX: ≥1.37, chrY: ≥-0.77) (XX)
- Del: ≤-0.4 (chrX: ≤-0.95, chrY: ≤-0.95), Dup: ≥0.31 (chrX: ≥0.56, chrY: ≥0.56) (XY) Del: ≤-0.4 (chrX: ≤0.6), Dup: ≥0.31 (chrX: ≥1.31, chrY: ≥-1.04) (XX)
- Del: ≤-0.3 (chrX: ≤-0.67, chrY: ≤-0.67), Dup: ≥0.24 (chrX: ≥0.45, chrY: ≥0.45) (XY) Del: ≤-0.3 (chrX: ≤0.7), Dup: ≥0.24 (chrX: ≥1.24, chrY: ≥-1.41) (XX)
На примере условия Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82) (XY) Del: ≤-0.7 (chrX: ≤0.3), Dup: ≥0.46 (chrX: ≥1.46, chrY: ≥-0.37) (XX) разберём, что означают эти условия фильтрации:
- Если хромосомный пол образца определен* как мужской (XY), то в соответствующие блоки отчётов попадут вариации, удовлетворяющие условию Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82). Если хромосомный пол образца определен* как женский (XX), то в соответствующие блоки отчётов попадут вариации, удовлетворяющие условию Del: ≤-0.7 (chrX: ≤0.3), Dup: ≥0.46 (chrX: ≥1.46, chrY: ≥-0.37).
- Вариации числа копий на аутосомах с log2FC ≤ -0.7 принимаются за делеции. Ниже порога log2FC=-0.7 вызывается одна аутосома вместо двух в норме.
- Вариации числа копий на аутосомах с log2FC ≥ 0.46 принимаются за дупликации. Порог для дупликации на аутосоме рассчитывается по следующей формуле: log2FC = log2((2+D)/2), где D - "количество" копийности, которое "теряется" при log2FC=-0.7, а 2 - количество сестринских хроматид в аутосоме.
- Вариации числа копий на X-хромосоме с log2FC ≤ -2.11 для XY и log2FC ≤ 0.3 для XX принимаются за делеции, а с log2FC ≥ 0.82 для XY и log2FC ≥ 1.46 для XX - за дупликации.
- Вариации числа копий на Y-хромосоме с log2FC ≤ -2.11 для XY принимаются за делеции, а с log2FC ≥ 0.82 для XY и log2FC ≥ -0.37 для XX - за дупликации.
- Вариации, не удовлетворяющие порогам, т.е. вариации на аутосомах с -0.7 < log2FC < 0.46, вариации на X-хромосоме с -2.11 < log2FC < 0.82 для XY и 0.3 < log2FC < 1.46 для XX и вариации на Y-хромосоме с -2.11 < log2FC < 0.82 для XY и log2FC < -0.37 для XX, не удовлетворяют условию фильтрации и не включаются в отчёт.
*Хромосомный пол определяется CNVkit на основе относительного числа копий аутосом и половых хромосом.
- Использование CNVs в качестве потенциальных находок: чтобы включить вариации числа копий, выявленные в образце и подходящие под заданные условия, в потенциальные находки, активируйте переключатель.
Пример блока в отчёте: отчёт с мутациями, подходящими под заданные условия.
12. Маркеры иммунотерапии#
Блок, сообщающий значение мутационной нагрузки опухоли (tumor mutation burden; TMB).
Высокая TMB - более 19 мутаций на 1 МБ - свидетельствует в пользу положительного
ответа на иммунотерапию. Низкая TMB - менее 6 мутаций на 1 МБ - может указывать на резистентность к иммунотерапии.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: маркеры иммунотерапии.
13. Клинические испытания#
Блок с таблицей активных клинических испытаний различных лекарств, технологий или диагностических подходов с участием пациентов, имеющих определенный фенотип из базы ClinicalTrials.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Фенотипы - список фенотипов, по которым должны отображаться клинические испытания. Названия фенотипов должны быть разделены точкой с запятой (например, "Glioma;Astrocytoma").
- Фазы испытаний: Phase 1, Phase 2, Phase 3, Phase 4. После выбора всех нужных фаз кликните вне области редактирования, чтобы сохранить изменения.
Пример блока в отчёте: клинические испытания.
14. SNVs/Indels по генам списка ACMG SF#
Блок с выявленными в образце вариантами, которые локализованы в генах из списка вторичных находок (secondary findings; SF) Американского колледжа медицинской генетики и геномики (ACMG). Для этих генов известны специфические мутации, приводящие к расстройствам с определённым фенотипом, на которые можно клинически подействовать с помощью общепринятого вмешательства. ACMG рекомендует сообщать о вариантах, обнаруженных в любом из этих генов, поскольку они имеют медицинское значение и могут быть использованы в будущем для информирования о клиническом лечении.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Тип мутации: соматическая или герминальная.
- Порог частоты слишком частых патогенных вариантов: рекомендуемое значение по умолчанию - 0,03.
- Использование наиболее важных для интерпретации SNVs/Indels в качестве потенциальных находок: чтобы включить в потенциальные находки выявленные в образце SNVs/Indels, расположенные в генах из списка ACMG SF и ассоциированные с указанным для генов фенотипом, (варианты из Списка 1), активируйте переключатель.
- Использование SNVs/Indels, требующих дальнейшего анализа, в качестве потенциальных находок: чтобы включить в потенциальные находки выявленные в образце SNVs/Indels, расположенные в генах из списка ACMG SF, для которых не удалось определить соответствие фенотипу или условиям, указанным для генов, (варианты из Списка 2), активируйте переключатель.
Пример блока в отчёте: вторичные находки ACMG.
15. Глиома: варианты уровня I#
Блок, отображающий соматические однонуклеотидные варианты, вариации числа копий и структурные вариации, выявленные в образце и относящиеся к Уровню I (Tier I) классификации соматических мутаций. Уровень I включает мутации, имеющие высокую клиническую значимость для глиомы с высоким уровнем доказательств и важные для диагностики и лечения рака.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: варианты с высокой клинической значимостью.
16. Глиома: варианты уровня II#
Блок, отображающий соматические однонуклеотидные варианты, вариации числа копий и структурные вариации, выявленные в образце и относящиеся к Уровню II (Tier II) классификации соматических мутаций. Уровень II включает мутации, имеющие потенциальную клиническую значимость для глиомы, то есть мутации, которые явно играют роль в развитии рака, но эта роль ещё недостаточно доказана.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
17. Глиома: варианты уровня III#
Блок, отображающий соматические однонуклеотидные варианты, вариации числа копий и структурные вариации, выявленные в образце и относящиеся к Уровню III (Tier III) классификации соматических мутаций. Уровень III включает мутации, имеющие неизвестную клиническую значимость для глиомы, то есть мутации, которые имеют сильное влияние на продукт гена и выявлены в генах, ассоциированных с различными типами раков, отличными от глиомы.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
18. Глиома: предполагаемый диагноз#
Блок, отображающий предполагаемый тип глиомы, определенный для образца. Предполагаемый диагноз и прогнозируемое время выживания основываются на автоматической интерпретации мутаций уровней I и II на основе опубликованных исследований. Предположения опираются на то, что ДНК опухоли была получена из глиомы, поэтому используйте данный блок в отчёте, только если вы анализируете образцы секвенирования глиомы. Если однозначно предположить диагноз не удается, предлагается до трех наиболее вероятных диагнозов.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: предполагаемый диагноз.
19. Глиома: предлагаемые препараты#
Блок, отображающий потенциально эффективные и неэффективные противоопухолевые препараты и соответствующие биомаркеры, выявленные в образце.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: предлагаемые препараты.
20. Глиома: подробности о мутациях#
Блок с описанием мутаций, попавших в блоки "Глиома: варианты уровня I" и "Глиома: варианты уровня II".
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: подробнее о мутациях.
21. Глиома: подробности о панели генов#
Блок с информацией по покрытию генов, найденных в образце и специфичных для глиом. В блоке представлены значения фракции экзонной части самого длинного транскрипта, покрытой по крайней мере 50 секвенированными прочтениями (50X), для генов, ассоциированных с глиомами. Чем выше процент покрытия, тем более полно секвенирован транскрипт гена.
Применим к образцам: опухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: подробнее о панели генов для глиомы.
22. Глиома: герминальные патогенные варианты#
Блок отображает выявленные в образце герминальные однонуклеотидные варианты, которые являются патогенными или вероятно патогенными для глиом. В блок включаются следующие варианты:
- Патогенные по данным ClinVar;
- Вероятно патогенные по данным ClinVar;
- Оказывающие сильное влияние на продукт гена (Значение эффекта == HIGH);
- Оказывающие умеренное влияние на продукт гена (Значение эффекта == MODERATE) и имеющие частоту встречаемости в популяции менее 10%;
- Редкие (имеющие частоту встречаемости в популяции менее 5%).
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: патогенные или вероятно патогенные для глиом.
23. Глиома: герминальные патогенные варианты для заболеваний, отличных от глиом#
Блок включает выявленные в образце герминальные однонуклеотидные варианты, которые являются патогенными или вероятно патогенными и ассоциированы с заболеваниями, отличными от глиом.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
24. Глиома: герминальные варианты, ассоциированные с глиомой#
Блок отображает выявленные в образце герминальные однонуклеотидные варианты, ассоциированные с глиомами согласно опубликованным исследованиям, но не попавшие в блок "Глиома: герминальные патогенные варианты".
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: ассоциированные с глиомой.
25. Глиома: список литературы#
Блок содержит ссылки на статьи, публикации и книги, посвященные исследованию глиомы.
Применим к образцам: опухолевые.
26. Герминальные CNV: результаты#
Блок представляет результат выявления герминальных вариаций числа копий (copy number variations; CNV) в образце. Он включает сообщение о наличии или отсутствии отклонения от нормального числа копий какой-либо хромосомы или плеча хромосомы в исследованном образце.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Отображать предполагаемый пол: по результатам выявления вариации числа копий половых хромосом (X, Y) можно
сделать предположение о поле пациента. Чтобы добавить результат этого предположения в отчёт,
активируйте переключатель:
.
Пример блока в отчёте: отчёт по CNV.
27. Герминальные CNV: интерпретация#
Блок представляет интерпретацию результатов выявления герминальных вариаций числа копий в образце и выводы по результатам. Блок включает:
- Таблицу с хромосомами или плечами хромосом, в которых была обнаружена структурная вариация (уменьшение или увеличение числа копий);
- График по типу кариограммы с обозначенными CNV уровня хромосом;
- Информацию о глубине прочтения генома.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
Пример блока в отчёте: отчёт по CNV.
28. Онко: Онкозначимые SNVs/Indels, выбранные пользователем для добавления в отчёт#
Блок отображает таблицу с информацией об онкозначимых вариантах, включенных в отчёт на странице SNV Viewer.
Включение вариантов в отчёт описано
в соответствующем разделе.
Онкозначимые варианты - это варианты, для которых есть клинические рекомендации, собранные на основе данных
различных сообществ, изучающих раковые заболевания (RUSSCO, АОР, КР МЗ РФ, NCCN, ESMO).
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: онкозначимые варианты.
29. Онко: Клинические рекомендации для SNVs/Indels, выбранных пользователем для добавления в отчёт#
Блок отображает клинические рекомендации для онкозначимых вариантов, включенных в отчёт на странице SNV Viewer.
Включение вариантов в отчёт описано
в соответствующем разделе.
Блок содержит:
- Поля с выбором (a) онкологического заболения, (b) его стадии и (c) уточнения стадии, которые были определены у пациента на момент взятия материала образца секвенирования; (d) линии терапии, используемой для лечения заболевания пациента на момент взятия материала образца секвенирования, а также (e) класса мутации (4 класса в зависимости от механизма, нарушающего функцию белка);
- Клинические рекомендации, собранные на основе данных различных сообществ, изучающих раковые заболевания (RUSSCO, АОР, КР МЗ РФ, NCCN, ESMO), и соответствующие (a) онкозначимому варианту, включенному в отчёт, и (b) выбранным онкологическому заболеванию, стадии, уточнению и линии терапии.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
Пример блока в отчёте: клинические рекомендации.
30. Полигенные признаки#
Блок отображает результаты предсказания по полигенным признакам - характеристикам, которые являются результатом действия большого количества разных генов, каждый из которых вносит небольшой вклад в суммарный показатель.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Полигенные признаки - выбор признаков, результат предсказания которых будет отображён в отчёте:
- Рост;
- Вес;
- ИМТ (индекс массы тела);
- Избыточный вес;
- Рак простаты (отобразится, если пол пациента определен как мужской);
- Рак молочной железы (отобразится, если пол пациента определен как женский);
- Ишемическая болезнь сердца;
- Воспалительное заболевание кишечника;
- Диабет второго типа;
- Колоректальный рак.
Пример блока в отчёте: отчёт по полигенным признакам.
31. SNVs/Indels по фенотипам ClinVar#
Блок отображает фенотип из базы ClinVar и связанные с ним рекомендации.
Применим к образцам: любые, опухолевые, неопухолевые.
Редактируемые поля:
- Фенотипы ClinVar - таблица фенотипов из базы ClinVar, соответственно которым
в отчёте представляется информация о выявленных в образце патогенных вариантах,
которые повышают риск развития этих фенотипов. Чтобы добавить фенотип,
нажмите
и заполните открывшуюся форму:
- MedGen ID - идентификатор фенотипа из базы MedGen (например, "C0745103").
- Имя в ClinVar - название фенотипа из базы ClinVar. Название должно быть в точности как в базе, например: "Hypercholesterolemia, familial, 1". Использовать имя в ClinVar имеет смысл, если MedGen ID для фенотипа неизвестен, иначе предпочтительнее указывать ID.
Для добавления фенотипа обязательно нужно заполнить одно из полей "MedGen ID" или "Имя в ClinVar". - Имя в отчёте - название фенотипа, которое будет отображаться в отчёте.
- Рекомендации - рекомендации, связанные с фенотипом, которые могут помочь пациенту снизить риски возникновения этой патологии. Текст рекомендаций поддерживается в формате Markdown.
После заполнения всех обязательных полей нажмите. Таким же образом можно добавить сколько угодно фенотипов.
- Тип мутации: соматическая или герминальная.
- Порог частоты крайне редких патогенных вариантов: рекомендуемое значение по умолчанию - 0,01.
- Порог частоты слишком частых патогенных вариантов: рекомендуемое значение по умолчанию - 0,15.
- Порог достоверности: рекомендуемое значение по умолчанию - 0,01.
Достоверность выявления варианта рассчитывается как (1 - Bin(n; m; p; 1)),
где Bin(n; m; p; 1) - это рассчитанная на основе биномиального распределения вероятность того, что вариант встретится ровно n раз среди m представителей (образцов пациентов) популяции;
n - количество представителей популяции (образцов пациентов), у которых был выявлен данный вариант;
m - общее количество представителей (образцов пациентов) в популяции;
p - вероятность встретить вариант среди представителей человеческой популяции (суммарная частота аллеля в базе данных gnomAD 3). Если данных о частоте в популяции нет, то вероятность принимается за 0,001.
Значения n и m берутся из пользовательской базы частот (если она не была загружена в систему, порог достоверности для фильтрации вариантов применяться не будет). - Заголовок списка фенотипов - заголовок списка состояний, с риском развития которых ассоциированы выявленные в образце патогенные или вероятно патогенные генетические варианты.
- Заголовок, если патогенных вариантов не обнаружено - текст, отображаемый в разделе с результатом отчёта, если в образце не обнаружено патогенных вариантов, повышающих риск развития исследуемых патологий.
- Описание найденных генетических вариантов - заголовок таблицы с выявленными в образце патогенными или вероятно патогенными генетическими вариантами, ассоциированными с риском развития исследуемых патологий.
- Заголовок рекомендаций - заголовок, отображаемый в подробном отчёте для рекомендаций по уточнению диагноза или дальнейшего обследования (при наличии таких рекомендаций для выявленной патологии в шаблоне отчёта).
- Рекомендации для пациента - текст общей рекомендации для пациента.
- Информация для врача - текст справки о том, каким образом составляется отчёт.
Пример блока в отчёте: отчёт на основе фенотипа ClinVar.
32. Фармакогенетика#
Блок отображает результаты фармакогенетического анализа - интерпретацию генетических данных пациента для прогнозирования его индивидуальной реакции на лекарственные препараты.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
33. Олигогенные признаки#
Блок отображает результаты предсказания по олигогенным признакам - характеристикам, формирование которых зависит от небольшого числа генов, каждый из которых вносит существенный вклад в проявление признака.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Олигогенные признаки - выбор признаков, результат предсказания которых будет отображён в отчёте:
- Цвет волос;
- Цвет глаз;
- Цвет кожи;
- Веснушчатость;
- Непереносимость лактозы;
- Горький вкус;
- Группа крови;
- Метаболизм алкоголя;
- Тип ушной серы;
- Подмышечный осмидроз;
- Мизофония;
- Движение во время сна;
- Световой рефлекс чихания.
Пример блока в отчёте: отчёт по олигогенным признакам.
34. Происхождение#
Блок отображает результаты генетического анализа происхождения, который проводится инструментом ADMIXTURE с использованием референтной генетической панели проекта Human Genome Diversity Project.
Применим к образцам: неопухолевые.
Редактируемые поля:
- Имя – заголовок раздела (обязательно).
- Описание – текст, отображаемый под заголовком.
- Порог вероятности для популяций - минимальный порог значения вероятности принадлежности, полученной в результате анализа происхождения. В отчёт включаются только те популяции, для которых рассчитанная вероятность, отражающая вклад данной популяции в генетическую структуру образца, превышает указанный порог. Рекомендуемое значение по умолчанию - 0,01.
Пример блока в отчёте: отчёт по происхождению.
Редактирование шаблона отчёта#
В шаблоне отчёта можно отредактировать его поля ("Имя", "Активен", "Заголовок отчёта", "Описание отчёта"), а также добавленные в него блоки.
На заметку
Все изменения в шаблоне применятся к образцам, если те были загружены после внесения изменений или если для них была перезапущена любая стадия анализа после внесения изменений.
Блок шаблона#
Добавленный блок шаблона выглядит следующим образом (пример - блок "Информация об образце"):

Сверху написано название блока, а в основной части приведена информация о его содержании.
Перемещение блока в шаблоне#
Для того чтобы поднять добавленный блок выше в шаблоне (выше предыдущего блока)
нажмите на , а чтобы опустить ниже - нажмите на
.
Редактирование блока шаблона#
Для того чтобы отредактировать добавленный блок
нажмите на , отредактируйте, а затем нажмите
для сохранения изменений. Для отмены внесенных изменений нажмите
.
Удаление блока шаблона#
Для того чтобы удалить добавленный блок
нажмите на , а затем на
в появившемся окне подтверждения. Для отмены удаления нажмите
или вне окна подтверждения.
Экспорт шаблона отчёта#
Опция эспорта доступна на странице шаблона, если в сохранённый шаблон был добавлен хотя бы один блок.
Чтобы экспортировать шаблон отчёта в формате JSON,
нажмите на  .
.
Импорт шаблона отчёта#
Чтобы импортировать шаблон отчёта в систему,
нажмите на  на странице "Шаблоны отчётов". Перетащите файл шаблона в формате JSON в открывшееся окно или нажмите на окно, чтобы открыть диспетчер файлов. После загрузки нажмите на
на странице "Шаблоны отчётов". Перетащите файл шаблона в формате JSON в открывшееся окно или нажмите на окно, чтобы открыть диспетчер файлов. После загрузки нажмите на  .
.
Удаление шаблона отчёта#
Чтобы удалить шаблон отчёта, наведите курсор на строку этого шаблона на странице "Шаблоны отчётов"
и нажмите на появившуюся корзину , а затем на
в появившемся окне подтверждения. Для отмены удаления нажмите
или вне окна подтверждения.
О построении отчёта#
Отчёт строится для образца, если выполняются следующие условия:
- Тип образца соответствует типу, к которому применим шаблон отчёта (Любые, Опухолевые или Неопухолевые).
- Анализ образца успешно завершился (то есть все стадии анализа имеют статус "Завершено").
- Для образца были получены необходимые для построения отчёта результаты. Если шаблон включает блоки, зависящие от определенных стадий анализа, (блоки с SNVs/Indels, вариациями числа копий, структурными вариациями или полигенными признаками), то построение отчёта будет зависеть от выполнения хотя бы одной из соответствующих стадий анализа образца. Блоки отчёта, зависящие от результатов стадий, не включенных в ход анализа, в отчёт не войдут. Так, если шаблон отчёта включает блок "Герминальные CNV: результаты", то построение этого блока будет зависеть от успешного завершения стадии "Выявление вариации числа копий".
- Шаблон отчёта активен.
- Шаблон отчёта имеет хотя бы один добавленный блок.
- Шаблон отчёта был добавлен раньше, чем был обработан образец.
Отчёт делится на разделы и блоки. Если вы не добавили ни одного блока "Раздел" в шаблон, то все блоки будут автоматически пронумерованы соответственно расположению в шаблоне (1, 2, 3, ...). Добавленные в шаблон разделы также нумеруются соответственно своему расположению в шаблоне (1, 2, 3, ...). Входящие в раздел блоки нумеруются соответственно расположению в шаблоне и в зависимости от номера раздела (1.1, 1.2, 1.3, ...). Если блок не был включен в итоговый отчёт, то он не учитывается в нумерации.