Поиск вариаций
Нужные вариации числа копий можно найти путём фильтрации. Параметры фильтрации находятся над таблицей:

На заметку
Фильтрация таблицы сохраняется при выходе со страницы CNV Viewer.
1. Log2FC пороги#
Фильтр вариаций по строгости, а именно порогам log2FC - логарифмического соотношения выявленного числа копий к нормальному числу копий. Возможные значения этого фильтра зависят от хромосомного пола образца, определяемого CNVkit на основе относительного числа копий аутосом и половых хромосом.
Если хромосомный пол образца определён как мужской (XY) или если пол определить не удалось, то возможными значениями фильтрации будут:
- Del: ≤-1, Dup: ≥0.58 (chrX: ≥1, chrY: ≥1) (XY)
- Del: ≤-0.9 (chrX: ≤-3.8, chrY: ≤-3.8), Dup: ≥0.55 (chrX: ≥0.94, chrY: ≥0.94) (XY)
- Del: ≤-0.8 (chrX: ≤-2.74, chrY: ≤-2.74), Dup: ≥0.51 (chrX: ≥0.88, chrY: ≥0.88) (XY)
- Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82) (XY) - значение по умолчанию
- Del: ≤-0.6 (chrX: ≤-1.64, chrY: ≤-1.64), Dup: ≥0.42 (chrX: ≥0.74, chrY: ≥0.74) (XY)
- Del: ≤-0.5 (chrX: ≤-1.27, chrY: ≤-1.27), Dup: ≥0.37 (chrX: ≥0.66, chrY: ≥0.66) (XY)
- Del: ≤-0.4 (chrX: ≤-0.95, chrY: ≤-0.95), Dup: ≥0.31 (chrX: ≥0.56, chrY: ≥0.56) (XY)
- Del: ≤-0.3 (chrX: ≤-0.67, chrY: ≤-0.67), Dup: ≥0.24 (chrX: ≥0.45, chrY: ≥0.45) (XY)
Если хромосомный пол образца определён как женский (XX), то возможными значениями фильтрации будут:
- Del: ≤-1 (chrX: ≤0), Dup: ≥0.58 (chrX: ≥1.58, chrY: ≥0) (XX)
- Del: ≤-0.9 (chrX: ≤0.1), Dup: ≥0.55 (chrX: ≥1.55, chrY: ≥-0.1) (XX)
- Del: ≤-0.8 (chrX: ≤0.2), Dup: ≥0.51 (chrX: ≥1.51, chrY: ≥-0.23) (XX)
- Del: ≤-0.7 (chrX: ≤0.3), Dup: ≥0.46 (chrX: ≥1.46, chrY: ≥-0.37) (XX) - значение по умолчанию
- Del: ≤-0.6 (chrX: ≤0.4), Dup: ≥0.42 (chrX: ≥1.42, chrY: ≥-0.55) (XX)
- Del: ≤-0.5 (chrX: ≤0.5), Dup: ≥0.37 (chrX: ≥1.37, chrY: ≥-0.77) (XX)
- Del: ≤-0.4 (chrX: ≤0.6), Dup: ≥0.31 (chrX: ≥1.31, chrY: ≥-1.04) (XX)
- Del: ≤-0.3 (chrX: ≤0.7), Dup: ≥0.24 (chrX: ≥1.24, chrY: ≥-1.41) (XX)
Алгоритм фильтрации:
- Нажмите на окно фильтрации;
- Из появившегося списка выберите нужную опцию фильтрации либо введите интересующую вас опцию в окно поиска и выберите из найденных.
Готово! Фильтр применен. То, какой выбран фильтр, можно увидеть в окошке:
При сбросе всех фильтров для фильтра по порогам log2FC применяется значение по умолчанию. Его можно изменить или очистить только в самом фильтре (см. далее).
На примере условия по умолчанию для XY: Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82), разберём, что означают эти условия фильтрации:
- Вариации числа копий на аутосомах с log2FC ≤ -0.7 принимаются за делеции. -0.7 - это порог log2FC по умолчанию, ниже которого вызывается одна аутосома вместо двух в норме.
- Вариации числа копий на X- и Y-хромосомах с log2FC ≤ -2.11 принимаются за делеции.
- Вариации числа копий на аутосомах с log2FC ≥ 0.46 принимаются за дупликации. Порог для дупликации на аутосоме рассчитывается по следующей формуле: log2FC = log2((2+D)/2), где D - "количество" копийности, которое "теряется" при log2FC=-0.7, а 2 - количество сестринских хроматид в аутосоме.
- Вариации числа копий на X- и Y-хромосомах с log2FC ≥ 0.82 принимаются за дупликации.
- Вариации, не удовлетворяющие порогам, т.е. вариации на аутосомах с -0.7 < log2FC < 0.46 и вариации на половых хромосомах с -2.11 < log2FC < 0.82, не проходят фильтрацию и исключаются из таблицы CNV Viewer.
Расчёт порогов log2FC для делеций и дупликаций на аутосомах и половых хромосомах#
Порог вызова одной копии вместо двух на аутосомах вычисляется следующим
образом: T21 = log2(C/2),
где C - количество вызванных копий.
Дельта числа копий: D = 2 – C = 2 – 2 × 2T21.
Тогда соответствующий порог вызова трёх копий вместо двух на аутосомах вычисляется
так: T23 = log2((2+D)/2) = log2(2-2T21).
Порог для вызова дупликаций на аутосомах: hi = T23.
Для вычисления порога для вызова трёх копий вместо двух на Х-хромосоме нормализуем генотип XY, поделив его на число копий, равное 1: T23X= log2((2+D)/1) = log2(2+2-2×2T21) = 1 + log2(2-2T21) = 1 + T23.
Вычисление порога для вызова дупликаций на X-хромосоме:
- если нет Y-хромосомы: Xhi = 1 + T23;
- если есть Y-хромосома: Xhi = log2(3-2×2T21).
Порог для вызова одной копии вместо двух на Х-хромосоме: T21X = log2((2-D)/1) = log2(2-2+2×2T21) = 1 + T21.
Порог для вызова ни одной копии вместо одной на X-хромосоме: T10X = log2((1-D)/1) = log2(1-2+2×2T21) = log2(-1+2×2T21).
Вычисление порога для вызова делеций на X-хромосоме:
- если нет Y-хромосомы: Xlo = 1 + T21;
- если есть Y-хромосома: Xlo = log2(-1+2×2T21).
Порог для вызова одной копии вместо ни одной на Y-хромосоме в случае генотипа XX: T01 = log2((0+D)/1) = log2(2-2×2T21) = log2(2×(1-2T21)) = 1 + log2(1-2T21).
Вычисление порога для вызова дупликаций Yhi и делеций Ylo на Y-хромосоме:
- если нет Y-хромосомы: Yhi = 1 + log2(1-2T21)); Ylo = -∞;
- если есть Y-хромосома: Yhi = Xhi; Ylo = Xlo.
2. Регион chr:start-end#
Фильтр вариаций по позиции. Для фильтрации введите позицию искомого геномного интервала: соответствующие хромосому и стартовую и конечную позиции. Возможные варианты записи позиции:
chr1:161,190,824-161,193,921chr1:161190824-161193921chr1:161190824_1611939211:161190824-161193921
Также можно фильтровать сразу по более, чем одному интервалу. Например, по chr1:161190824-161193921, chrX:151110769-151113866.
Результатом фильтрации будут строки с вариациями, чьи позиции пересекаются с введённым интервалом.
3. Панель генов#
Фильтр вариаций по расположенным на их участках генам из панели. Вы можете добавить панель генов на странице "Панели генов" или импортировать панель из библиотеки самых распространенных клинических панелей. Для импорта нажмите на "Импорт из библиотеки":
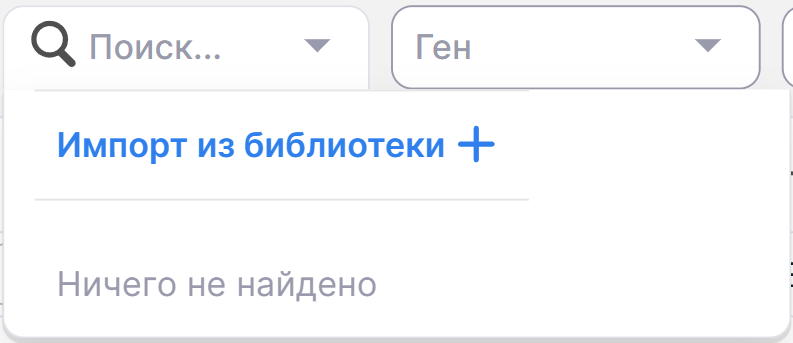
В открывшейся форме выберите нужные панели, отметив их галочками:

и нажмите на , чтобы добавить панель.
4. Ген#
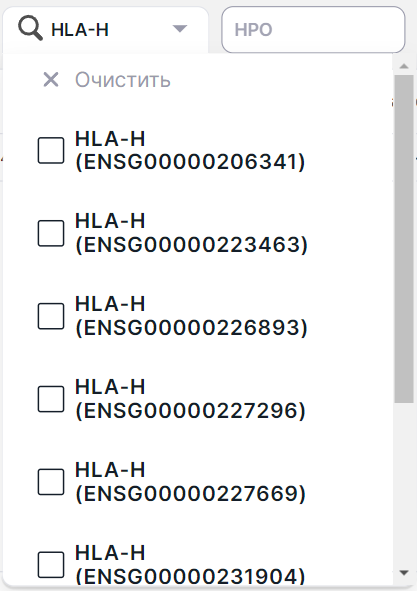
Фильтр вариаций по расположенным на их участках транскриптам генов. Транскрипт приведен в скобках рядом с официальным названием гена. Можно отфильтровать вариации сразу по нескольким генам. Для этого отметьте галочками нужные гены и кликните вне области заполнения значений:
Если для гена известно несколько транскриптов, в списке представлены все:
5. HPO#
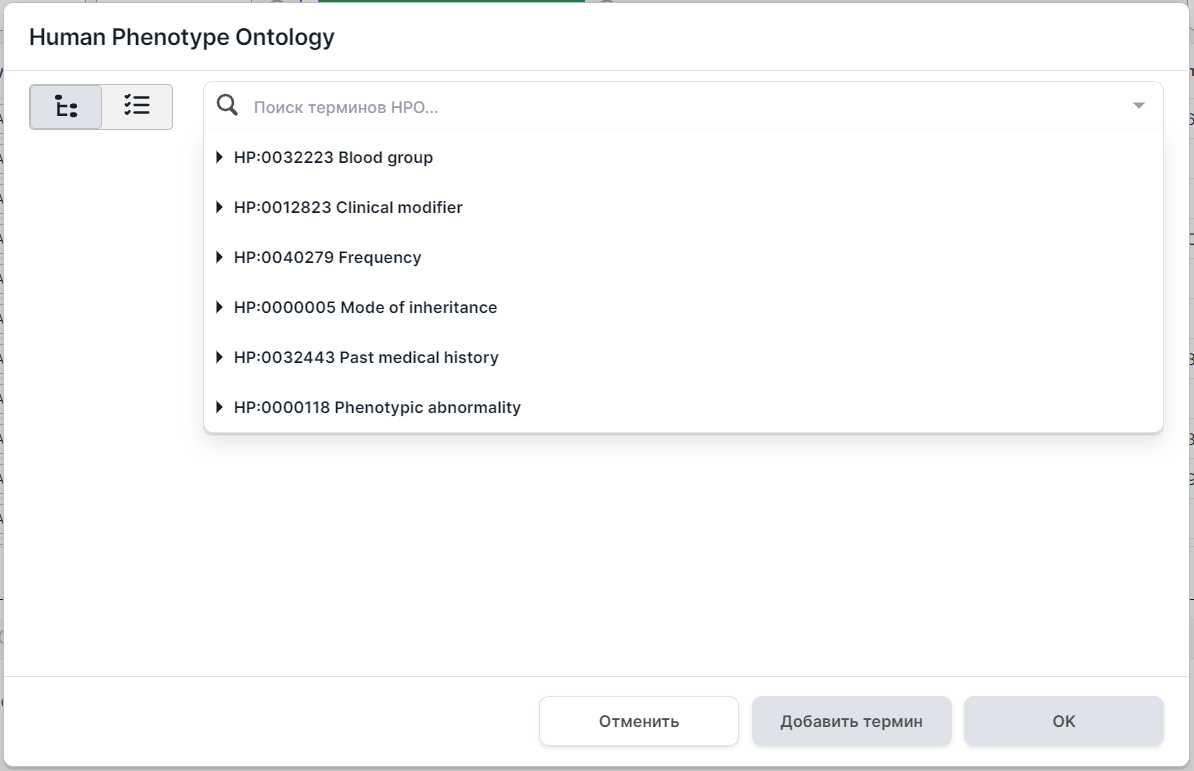
Фильтр вариаций по расположенным на их участках генам, ассоциированным с терминами из словаря фенотипических аномалий человека Human Phenotype Ontology (HPO). Нажмите на окно фильтрации по HPO, чтобы открыть форму поиска фенотипа. В форме в выпадающем списке представлены основные фенотипические группы:
Способы поиска и выбора фенотипа для фильтрации:
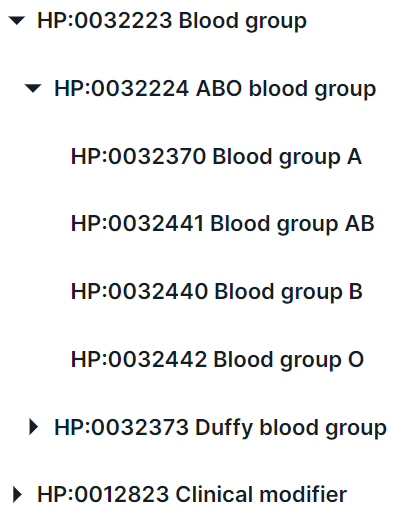
- Поиск фенотипа непосредственно в выпадающем списке. С фенотипическими группами можно
взаимодействовать с помощью кнопок
и
для раскрытия и сворачивания списка терминов группы, соответственно:
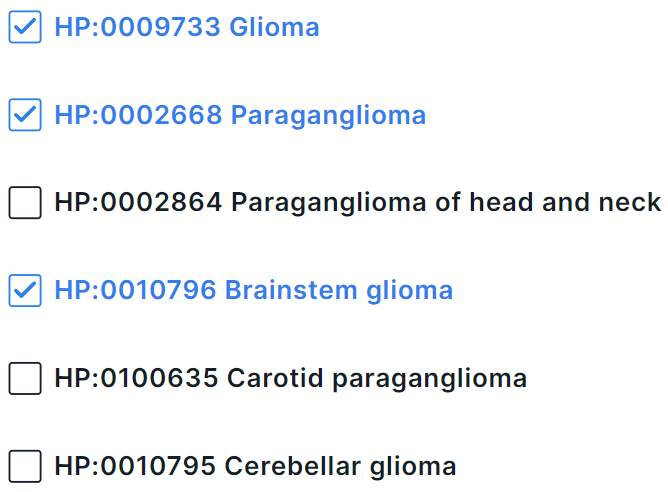
Кроме того, фенотип можно найти через поиск по названию или идентификатору из HPO. Например, при поиске заболевания “glioma” находятся несколько терминов:
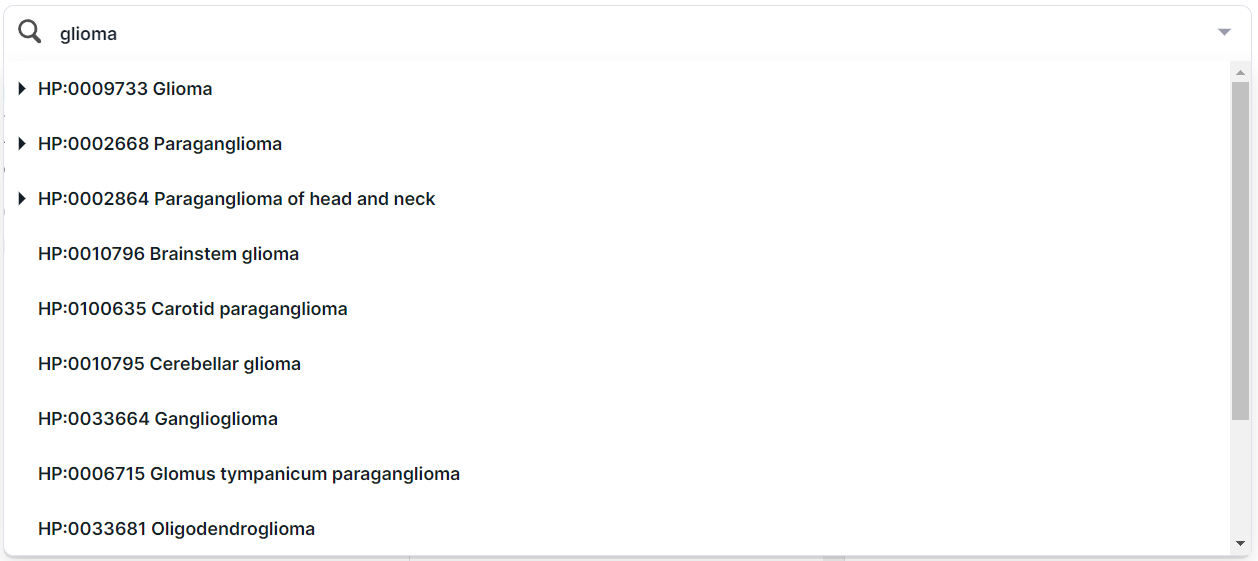
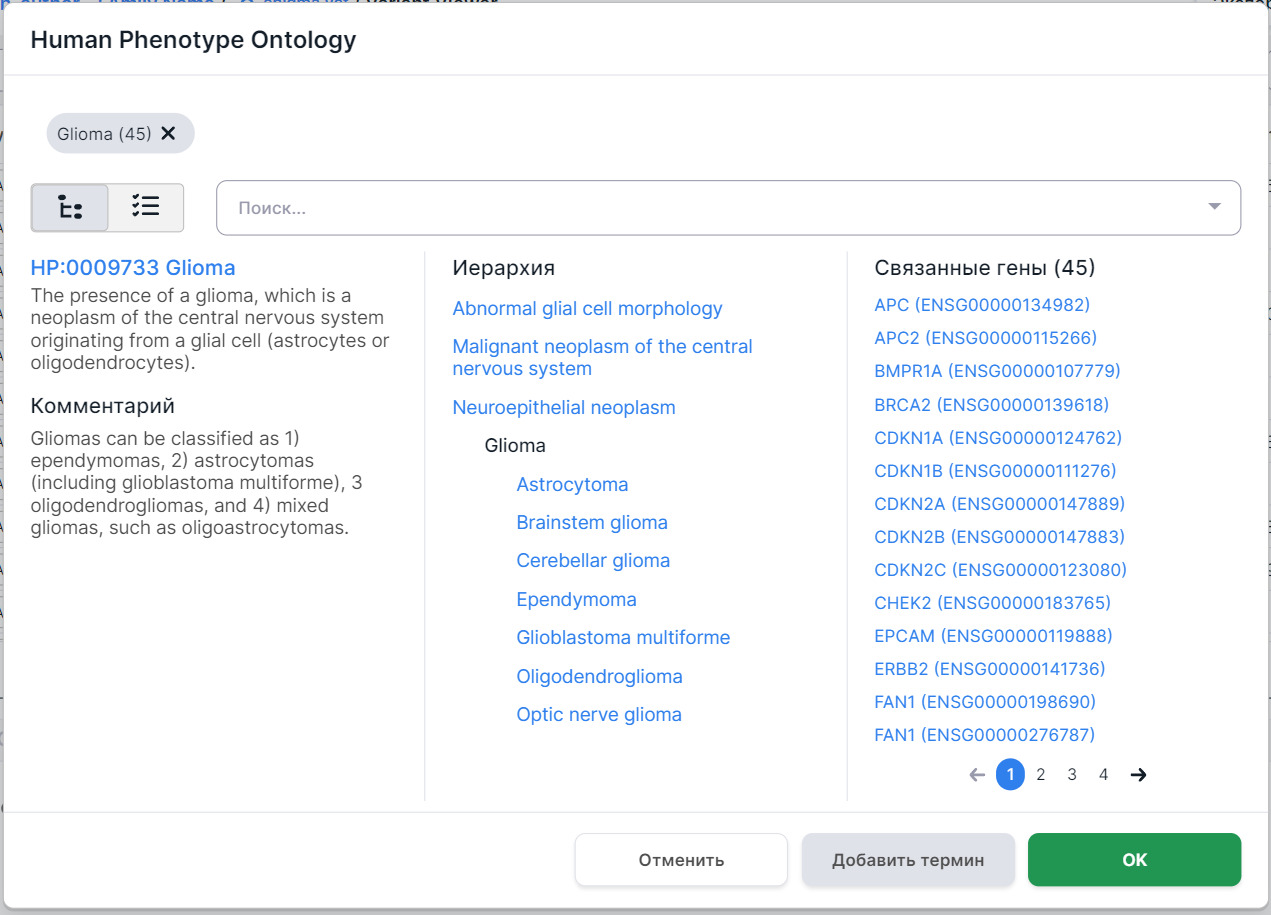
Для выбора фенотипа для фильтрации нажмите на его строку. Можно выбрать как целую группу, так и конкретный фенотип. Выбрав фенотип (например, "Glioma"), вы увидите его карточку:
Над карточкой фенотипа отмечены термины, выбранные для фильтрации. В скобочках указано количество
связанных с фенотипом генов. Нажав на фенотип, вы увидите его карточку. Фенотип можно удалить,
нажав на крестик :
Слева в карточке фенотипа даны его определение и комментарий. В середине представлена иерархия фенотипа: выше выбранного заболевания Glioma находятся его родительские термины (те фенотипические группы, которые включают Glioma), а ниже - его дочерние термины (заболевания, которые входят в группу Glioma). Справа представлены гены, ассоциированные с данным фенотипом (в данном случае было найдено 45 генов).
Поиск фенотипа по иерархии в карточке фенотипа: нажав на название родительского или дочернего фенотипа, вы попадете на его страницу. Если фенотип ещё не выбран для фильтрации и найдены ассоциированные с ним гены, то его можно выбрать для фильтрации вариаций, нажав на кнопку
.
Поиск фенотипа в режиме мультиселекта: для этого переключите режим поиска, нажав на
в левом верхнем углу окна. Вы увидите список основных фенотипические группы с флажками для выбора. Фенотип можно найти через поиск по названию или идентификатору из HPO. Например, при поиске заболевания “glioma” находятся несколько терминов:
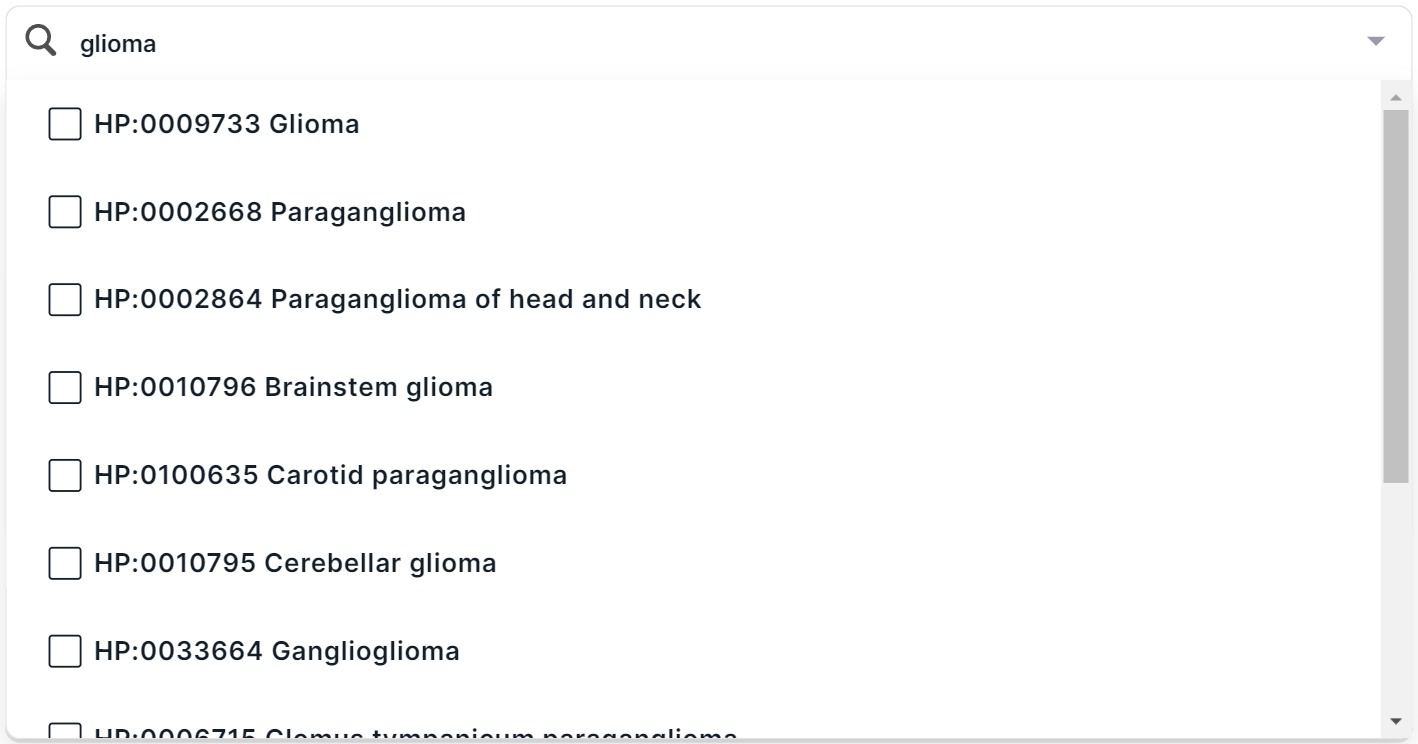
В режиме мультиселекта можно выбрать сразу несколько терминов в выпадающем списке. Для выбора фенотипа для фильтрации нажмите на его строку:
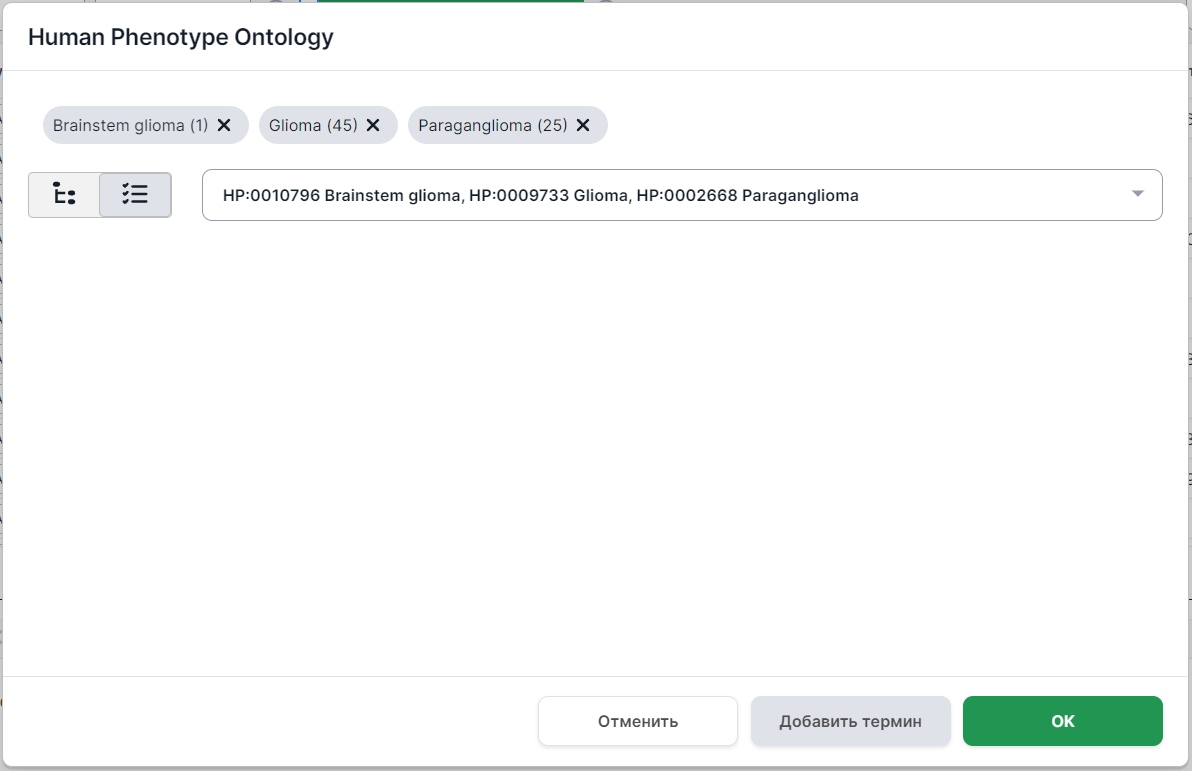
Чтобы закончить добавление фенотипа для фильтрации, кликните вне области выпадающего списка в
пределах формы поиска фенотипа. Наверху формы вы увидите термины, выбранные для фильтрации. В скобочках
указано количество связанных с фенотипом генов. Нажав на фенотип, вы увидите его карточку.
Фенотип можно удалить, нажав на крестик .
Чтобы отфильтровать вариации по генам, связанным с выбранным фенотипом,
нажмите .
Выбранный фенотип можно увидеть во всплывающей подсказке, наведя курсор на поле фильтрации по HPO:

Для того чтобы отменить фильтрацию по всем терминам, наведите курсор на окно фильтрации по HPO и нажмите на появившийся крестик:
или нажмите на поле и отредактируйте свой выбор в форме поиска фенотипа.
6. Тип#
Фильтр вариаций по типу изменения числа копий: LOSS - делеция, GAIN - дупликация.
7. CDS Перекрытие#
Фильтр вариаций по перекрытию с кодирующей последовательностью (CoDing Sequence; CDS):
- Overlapped CDS - вариация затрагивает кодирующую последовательность (процент перекрытия с CDS больше 0);
- Not overlapped CDS - вариация не затрагивает кодирующую последовательность (процент перекрытия с CDS равен 0).
8. Класс ACMG#
Фильтр вариаций по патогенности, выставляемой соответственно категории, определённой AnnotSV:
- Pathogenic - Патогенная;
- Likely pathogenic - Вероятно патогенная;
- Uncertain significance conflict - Противоречивые интерпретации патогенности;
- Uncertain significance - Неопределенная значимость;
- Likely benign - Вероятно доброкачественная;
- Benign - Доброкачественная;
- Undefined - Нет интерпретации.
Можно отфильтровать вариации сразу по нескольким значениям патогенности. Для этого отметьте галочками нужные значения и кликните вне области заполнения значений.
9. AF по когорте#
Фильтр вариаций по частоте в когорте, состоящей из 188 нормальных образцов. Значения частот есть только для герминальных вариаций числа копий, выявленных в образцах полногеномного секвенирования (WGS или low-pass WGS), для нормализации которых использовались панели референсных образцов "BGI WGS" с размером бина 3000 или 5000 или "BGI WGS v2" с размером бина 3000. Фильтр отображается только в CNV Viewer с варициями числа копий, выявленными в бинах.
Возможные значения:
- <0.05 или пусто;
- <0.02 или пусто;
- <0.01 или пусто;
- <0.005 или пусто;
- <0.0001 или пусто.
Сброс фильтрации#
Для сброса всех примененных фильтров
нажмите на .
Сохранится только фильтрация по порогам log2FC по
умолчанию: - Del: ≤-0.7 (chrX: ≤-2.11, chrY: ≤-2.11), Dup: ≥0.46 (chrX: ≥0.82, chrY: ≥0.82).
Для сброса отдельного фильтра:
- Нажмите на окно фильтрации и затем на
.
- Для сброса фильтрации по региону наведите курсор на окно фильтрации и нажмите на появившийся крестик:

- У фильтра по гену можно убрать одну из выбранных опций фильтрации. Для этого нажмите на окно фильтрации и снимите галочку у опции.
- Для изменения выбора фенотипа HPO, по которому идёт фильтрация вариаций, нажмите на поле фильтрации по HPO и отредактируйте свой выбор в форме поиска фенотипа. Для сброса фильтрации по всем выбранным терминам наведите курсор на окно фильтрации по HPO и нажмите на появившийся крестик: