Вариации числа копий
Секции "Соматические вариации числа копий" и "Герминальные вариации числа копий" содержат результаты выявления, соответственно, соматических и герминальных участков изменения числа копий (copy number variations; CNVs) и расчёта значений копийности для данного образца на фоне других образцов.
Соматические вариации являются приобретенными и характерны для опухолевого процесса. Соответственно, выявление таких вариаций проводится только для образцов опухолевых тканей и результаты представлены только на странице образца опухолевой ткани.

Герминальные вариации являются наследуемыми и могут быть обнаружены в любых клетках организма. Соответственно, выявляние таких вариаций проводится как для образцов опухолевых (в случае одиночных образцов опухолевой ткани без контроля), так и для образцов здоровых тканей. В случае набора опухолевого и контрольного образцов результаты выявления герминальных вариаций у контрольного образца представлены на страницах обоих образцов набора.

Для одиночного образца опухолевой ткани без контроля результаты выявления соматических и герминальных вариаций объединены в одну секцию:

Обнаруженные и аннотированные вариации числа копий (copy number variations; CNVs) во всех секциях представлены в следующем виде:
- CNV Viewer - встроенный модуль для просмотра и анализа вариаций. Подробно описан
в соответствующем разделе.
Нажав на кнопку
 ,
вы увидите две опции: "Сегменты" и "Бины", которые открывают CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно:
,
вы увидите две опции: "Сегменты" и "Бины", которые открывают CNV Viewer с
вариациями числа копий, выявленными в сегментах или бинах, соответственно:

- Файл в формате AnnotSV CSV - таблица аннотированных участков изменения копий.
Участки представлены как в виде полной делеции или инсерции,
так и в виде разбитых на гены участков внутри делеции/инсерции.
Нажав на кнопку
, вы увидите опции для скачивания. Их количество будет зависеть от того, остались ли какие-то вариации после фильтрации. Возможные опции: "Сегменты - все", "Сегменты после фильтрации", "Бины - все" и "Бины после фильтрации". Чтобы скачать файл с аннотированными вариациями, выявленными в сегментах или бинах, до или после фильтрации, выберите соответствующую опцию:

Нажмите, чтобы посмотреть основные поля AnnotSV CSV
Основные поля участков изменения копий#
| Поле | Описание |
| AnnotSV_ID | Идентификатор вариации, присваиваемый аннотатором |
| SV_chrom | Хромосома |
| SV_start | Начало участка |
| SV_end | Конец участка |
| SV_length | Длина участка |
| SV_type | Тип изменения числа копий (DEL - делеция, DUP - дупликация) |
| Filter | Значение фильтрации: PASS (вариация удовлетворяет всем фильтрам) или пусто (вариация не прошла фильтрацию). Значение будет всегда "PASS" в файлах после фильтрации. |
| log2FC | Логарифмическое соотношение выявленного числа копий к нормальному числу копий (равно двум для аутосом и X-хромосомы в случае генотипа XX либо единице для половых хромосом в случае генотипа XY). |
| deltaCP | Относительная копийность - количество копий некоторого участка на гаплоидный геном. |
| Annotation_mode | Тип записи: full - участок целиком, split - участок, разбитый по генам. Каждому full-участку могут соответствовать несколько split-участков. |
Описание остальных 126 полей аннотации можно найти по ссылке: AnnotSV Manual.
- Файл AnnotSV в таблицах Google (может быть показана только часть вариаций).
Нажав на кнопку
, вы увидите опции файла. Их количество будет зависеть от того, остались ли какие-то вариации после фильтрации. Возможные опции: "Сегменты - все", "Сегменты после фильтрации", "Бины - все" и "Бины после фильтрации". Чтобы открыть файл с аннотированными вариациями, выявленными в сегментах или бинах, до или после фильтрации, выберите соответствующую опцию:

- Файл покрытия CNV в формате TSV - неаннотированный файл,
удобный для рассмотрения изменения копийности на крупных участках (плечи хромосом, хромосомы).
Чтобы скачать файл, нажмите на
.
Нажмите, чтобы посмотреть основные поля файла покрытия CNV
Основные поля описания варианта#
| Поле | Описание |
| ID | Название локуса (плеча, хромосомы) |
| Call | Вердикт по количеству копий. Принимает значения "+" (увеличенное количество копий по сравнению с нормой), "-" (уменьшенное число копий по сравнению с нормой), "." (не было замечено отклонений в числе копий) и "Not considered" (локус вообще не рассматривался при анализе). |
| Ampl_cov | Покрытие (в нуклеотидах) эффективной длины локуса сегментами с количеством копий выше порога амплификации |
| Ampl_cov_frac | Доля (в %) эффективной длины локуса, покрытая сегментами с количеством копий выше порога амплификации |
| Arm | Плечо хромосомы |
| Chrom | Хромосома |
| Depl_cov | Покрытие (в нуклеотидах) эффективной длины локуса сегментами с количеством копий ниже порога деплеции |
| Depl_cov_frac | Доля (в %) эффективной длины локуса, покрытая сегментами с количеством копий ниже порога деплеции |
| Effective_end | Максимальное значение координаты эффективной длины |
| Effective_length | Суммарная эффективная длина |
| Effective_start | Минимальной значение координаты эффективной длины |
| End | Координата конца локуса |
| Length | Длина локуса |
| Region type | Тип локуса: плечо или вся хромосома |
| Start | Координата начала локуса |
| is_masked | Была ли проведена маскировка повторов в последовательности: True/False |
| seg_val | Среднее значение сегмента |
- Полногеномный график сегментов CNV.
Нажав на кнопку
, вы увидите следующие опции:
- Опция "Сводный график" открывает полногеномный график сегментов CNV в формате JPG. Пример графика:
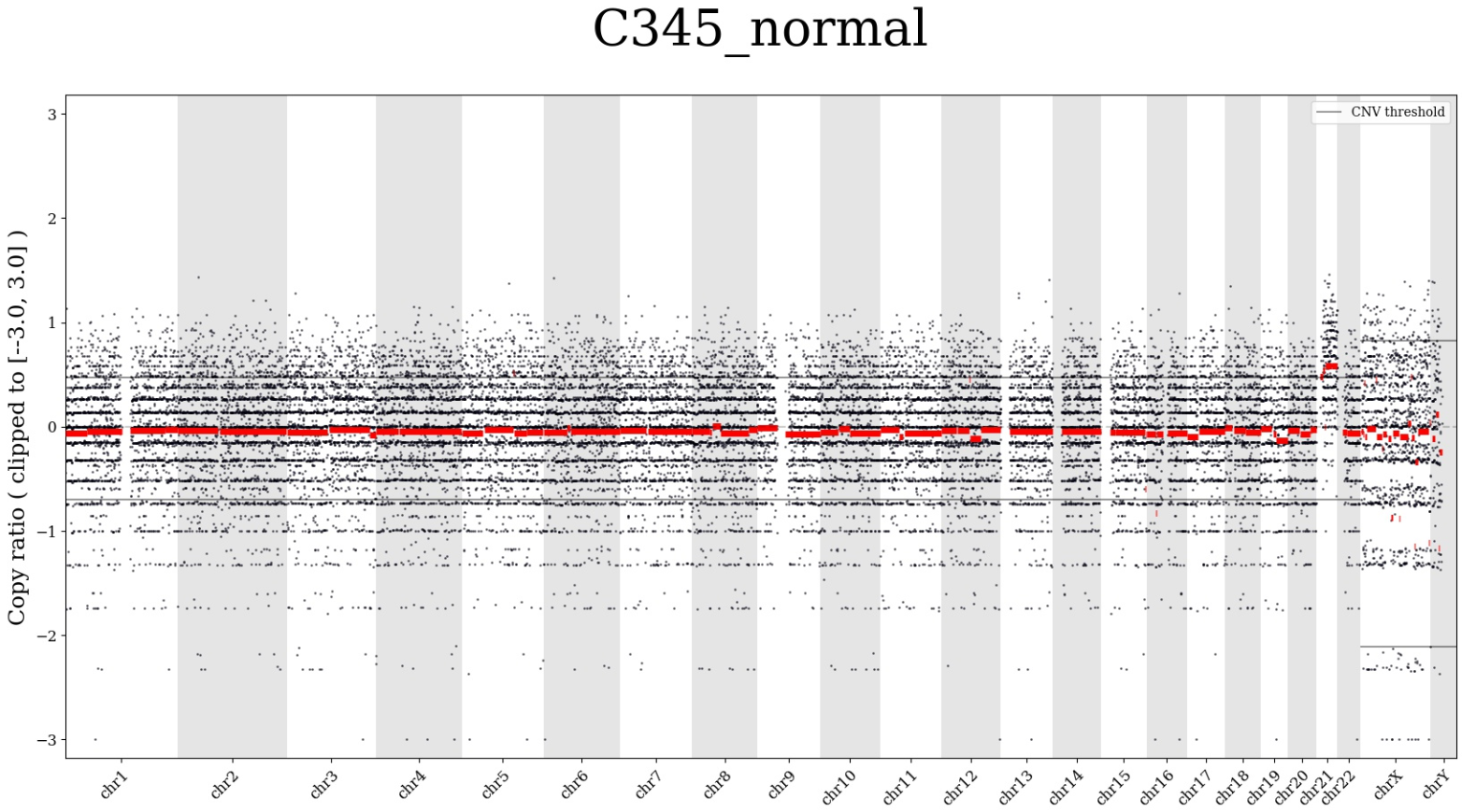
На горизонтальной шкале мы видим хромосомы, по вертикали - копийность хромосомы. Так, для этого образца копийность хромосомы 21 увеличена в несколько раз.
- Опция "Графики по хромосомам" открывает файл в формате PDF с графиками сегментов CNV для каждой хромосомы. Пример графика для chr21:
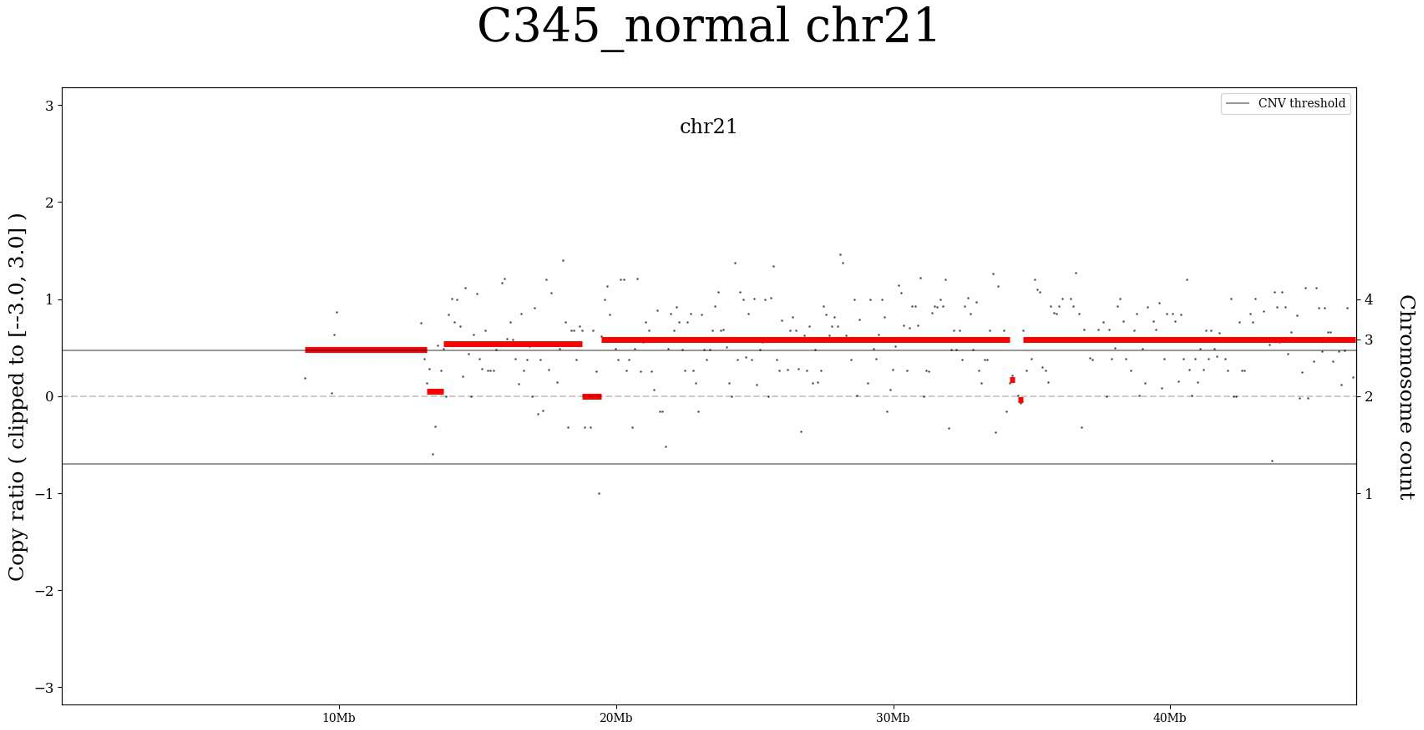
На горизонтальной шкале - геномные интервалы хромосомы, по вертикали - копийность хромосомы. На графике мы видим, что для этого образца копийность хромосомы 21 увеличена в несколько раз.
- График по типу кариограммы с обозначенными CNV уровня хромосом в формате JPG. Чтобы открыть график,
нажмите на
. Пример графика:
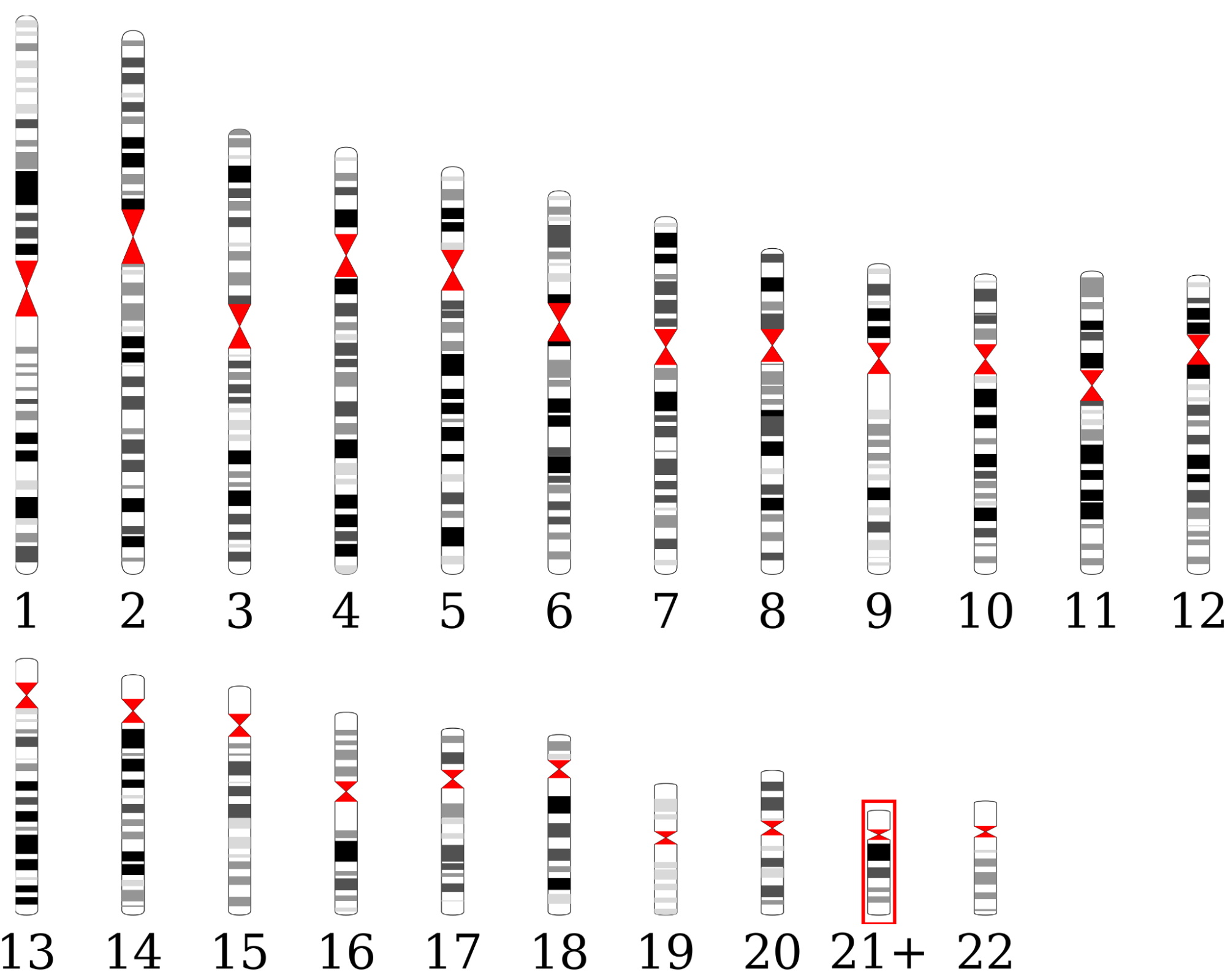
На кариограмме обозначено, что в образце наблюдается увеличение числа копий хромосомы 21.
- Integrative Genomics Viewer (IGV) - встроенный
модуль визуализации вариаций на геноме. Чтобы открыть модуль,
нажмите на
. Чтобы открыть одновременно два региона по границам некоторого участка в IGV, нажмите на
, а чтобы вернуться к визуализации всего участка, нажмите на
. Если вы хотите перейти на страницу позиции, открытой в IGV, в геномном браузере Decipher, нажмите на
.