Панель детальной информации о вариации
Нажмите на строку вариации, чтобы увидите панель с детальной информацией о ней под таблицей. Вид панели отличается для общих и детальных строк по генам. Панель с детальной информацией об участке вариации целиком:
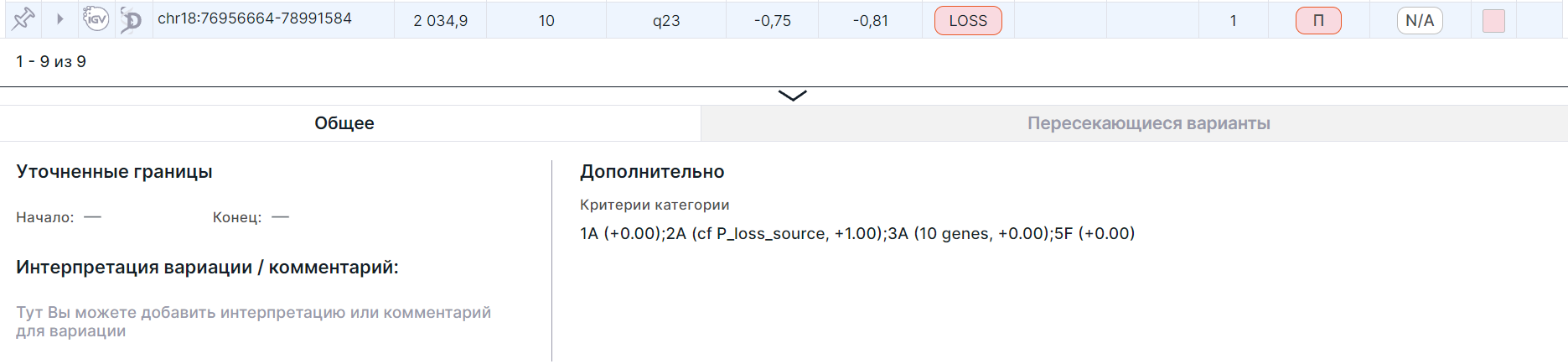
Панель с детальной информацией об участке, разбитом по генам:
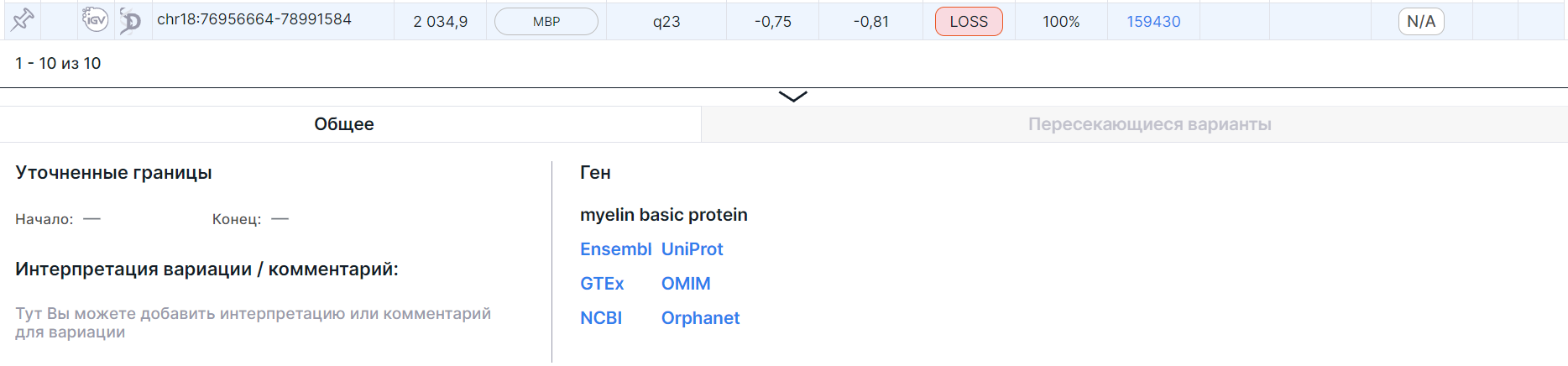
Размер панели можно менять, наведя курсор на верхнюю границу панели и потянув вниз или вверх.
Также панель можно скрыть и развернуть, нажав на соответствующие
стрелки или
.
На заметку
На странице "Настройки пользователя" можно отрегулировать, будет ли панель детальной информации о вариации развёрнута или скрыта в CNV Viewer по умолчанию.
Панель разделена на вкладки "Общее" и "Пересекающиеся варианты".
На панели визуально выделяются только те вкладки, которые включают хотя бы одно не пустое поле аннотации
вариации. Например, вкладка "Пересекающиеся варианты", наполненная
данными, выглядит так: , а пустая - так:
.
При перемещении между вариациями в таблице выбранная вкладка панели сохраняется.
Общее#
Вид вкладки "Общее" отличается для общих и детальных строк. Вкладка с общей информацией об участке вариации целиком выглядит так:

Вкладка с общей информацией об участке, разбитом по генам, выглядит следующим образом:

Вкладка содержит основную информацию о вариации и может включать следующие разделы:
- Уточненные границы;
- Интерпретация вариации;
- Дополнительно (только для общих строк);
- Ген (только для детальных строк);
- Связанные фенотипы OMIM (только для детальных строк, если предоставлен доступ к базе данных OMIM).
Уточненные границы#
В разделе можно уточнить стартовую и конечную позиции вариации, введя их в соответствующие поля "Начало" и
"Конец". Вводить можно только цифры без каких-либо дополнительных знаков.
После уточнения границ вариации появятся кнопки перехода в модуль визуализации вариации в уточненных границах
на геноме (IGV) - и в учтоненные границы вариации в геномном браузере Decipher -
:

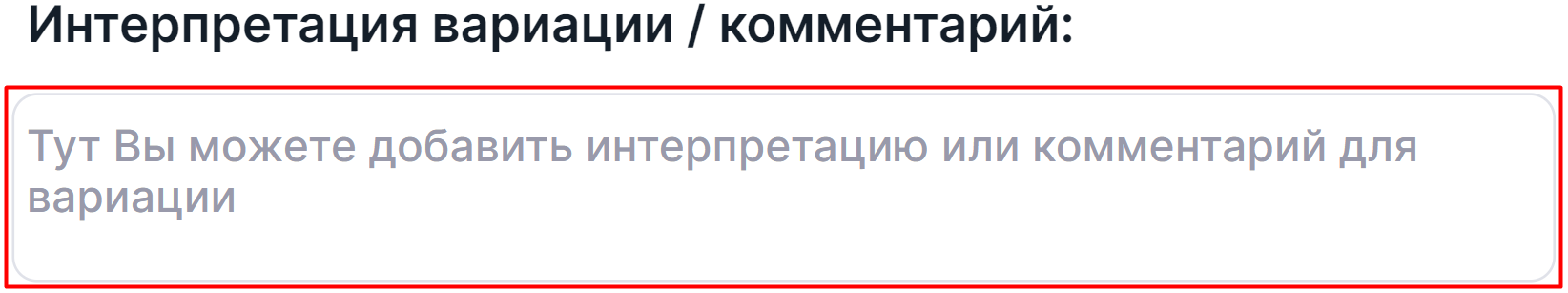
Интерпретация вариации / комментарий#
Интерпретация вариации - это комментарий с важной информацией о вариации, добавленный пользователем. Чтобы добавить или отредактировать интерпретацию вариации, нажмите на поле ввода, введите нужный текст и кликните вне текстового поля для сохранения интерпретации:
Дополнительно#
Для общих строк, представляющих участок вариации целиком, приведен критерий категории - критерий принятия решения, объясняющий категорию, предоставляемую AnnotSV. Полные и подробные рекомендации по оценке и расшифровке критерия категории доступны в файле AnnotSV_Scoring_Criteria.xlsx (см. Таблицу 1 для дупликаций и Таблицу 2 для делеций). Категория - рейтинговая оценка патогенности вариации, основанная на рекомендациях ACMG и ClinGen1. Согласно рейтинговой оценке выставляется класс ACMG:
- Вариациям с рейтинговой оценкой ≥ 0.99 выставляется класс 5 - патогенные;
- Вариациям с рейтинговой оценкой от 0.90 до 0.98 выставляется класс 4 - вероятно патогенные;
- Вариациям с рейтинговой оценкой от 0.89 до −0.89 выставляется класс 3 - неопределенная значимость;
- Вариациям с рейтинговой оценкой от −0.90 до −0.98 выставляется класс 2 - вероятно доброкачественные;
- Вариациям с рейтинговой оценкой ≤ −0.99 выставляется класс 1 - доброкачественные.
Ген#
Для детальных строк, представляющих участки вариации, разбитые по генам, приведена информация о гене, расположенном на этом участке:
- Полное название гена. При наведении курсора на название гена можно увидеть, из каких баз данных было взято полное название:

- Ссылки на страницы с информацией о гене в различных базах данных (Ensembl, UniProt, GTEx, OMIM, NCBI, Orphanet).
Связанные фенотипы OMIM#
Доступ к данным из базы OMIM внутри Genomenal предоставляется при наличии у пользователя лицензии на доступ к базе. Если вам предоставлен доступ к OMIM, то для детальных строк, представляющих участки вариации, разбитые по генам, справа на вкладке "Общее" отобразится информация по фенотипу, ассоциированному с геном, расположенным на участке вариации:
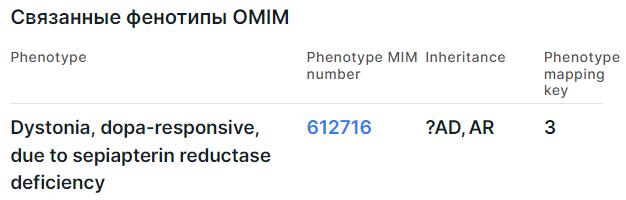
Информация приводится в виде таблицы со следующими колонками:
- Phenotype - полное название фенотипа, связанного с геном. Если перед названием фенотипа стоит знак вопроса (например, "?Bleeding disorder, platelet-type, 22"), это означает, что взаимосвязь между фенотипом и геном является предварительной. Если название фенотипа заключено в квадратные скобки (например, "[Urate oxidase deficiency]"), это указывает в основном на генетические вариации, которые приводят к явно аномальным значениям лабораторных тестов. Если название фенотипа заключено в фигурные скобки (например, "{Prostate cancer/brain cancer susceptibility, somatic}"), это обозначает мутации, которые способствуют предрасположенности к многофакторным заболеваниям или восприимчивости к инфекциям.
- Phenotype MIM number - идентификатор фенотипа в базе OMIM. Перейдя по ссылке, вы попадете на страницу фенотипа в OMIM.
- Inheritance - тип наследования фенотипа:
AD - autosomal dominant, аутосомно-доминантный;
AR - autosomal recessive, аутосомно-рецессивный;
DD - digenic dominant, дигенный доминантный;
DR - digenic recessive, дигенный рецессивный
IC - isolated cases, единичные случаи;
Mi - mitochondrial, митохондриальный;
Mu - multifactorial, мультифакторный;
PADom - pseudoautosomal dominant, псевдоаутосомно-доминантный;
PARec - pseudoautosomal recessive, псевдоаутосомно-рецессивный;
SomMos - somatic mosaicism, соматический мозаицизм;
SMu - somatic mutation, соматическая мутация;
XL - X-linked, X-сцепленный;
XLD - X-linked dominant, X-сцепленный доминантный;
XLR - X-linked recessive, X-сцепленный рецессивный;
YL - Y-linked, Y-сцепленный. - Phenotype mapping key - ключ карты фенотипа в OMIM (карта отображает связи данного фенотипа с
ассоциированными фенотипами, группами родственных фенотипов (фенотипическими рядами) и генами):
1 - фенотип помещен на карту из-за его связи с геном, но лежащий в его основе дефект неизвестен;
2 - фенотип помещен на карту в результате использования статистических методов, мутация не обнаружена;
3 - молекулярная основа фенотипа известна, в гене была найдена мутация;
4 - синдром смежной дупликации или делеции генов, в котором участвуют несколько генов.
Пересекающиеся варианты#
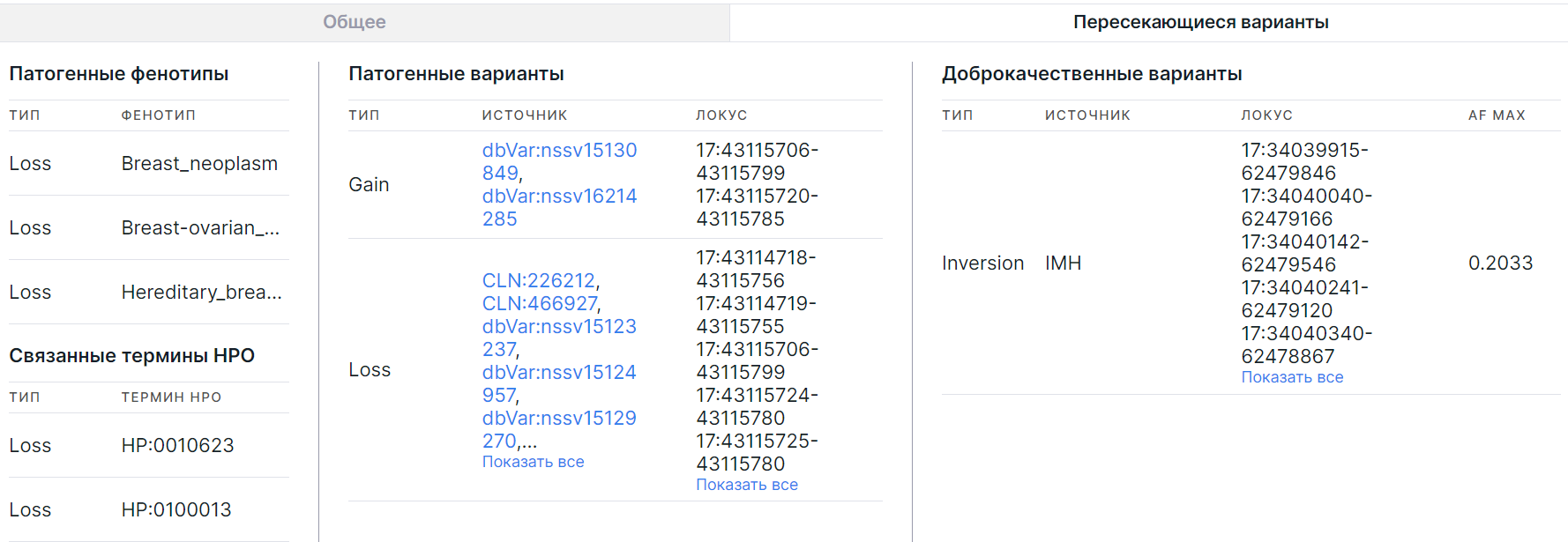
Вкладка содержит информацию о фенотипе и терминах HPO, ассоциированных с геномной областью патогенной делеции, дупликации или инсерции, которая полностью перекрывается вариацией числа копий, а также информацию о патогенных и доброкачественных вариантах, пересекающихся с вариацией.
Патогенные фенотипы#
Фенотипы представлены в виде таблицы со следующими колонками:
- Тип - тип изменения числа копий: Loss - делеция, Gain - дупликация, Insertion - инсерция.
- Фенотип - название фенотипа геномной области патогенной делеции, дупликации или инсерции, полностью перекрывающейся вариацией. При наведении курсора на значение колонки можно увидеть полное название фенотипа.
Связанные термины HPO#
Термина HPO представлены в виде таблицы со следующими колонками:
- Тип - тип изменения числа копий: Loss - делеция, Gain - дупликация, Insertion - инсерция.
- Термин HPO - термин из словаря фенотипических аномалий человека Human Phenotype Ontology (HPO), ассоциированный с геномной областью патогенной делеции, дупликации или инсерции, полностью перекрывающейся вариацией.
Патогеннные варианты#
Патогенные пересекающиеся варианты представлены в виде таблицы со следующими колонками:
- Тип - тип изменения числа копий: Loss - делеция, Gain - дупликация, Insertion - инсерция.
- Источник - источники сведений об установленных патогенных генах или геномных областях, полностью перекрывающихся вариацией. Возможные источники: dbVar, ClinVar (CLN), ClinGen (TS3 для дупликаций или HI3 для делеций), название гена, ассоциированного с заболеваниями, из OMIM (morbid).
- Локус - позиции патогенных геномных областей, полностью перекрывающихся вариацией (хромосома:стартовая позиция-конечная позиция).
Доброкачественные варианты#
Доброкачественные пересекающиеся варианты представлены в виде таблицы со следующими колонками:
- Тип - тип изменения числа копий: Loss - делеция, Gain - дупликация, Insertion - инсерция, Inversion - инверсия.
- Источник - источники сведений об установленных доброкачественных геномных областях, полностью перекрывающихся вариацией. Возможные источники: gnomAD, ClinVar (CLN), ClinGen (TS40 для дупликаций или HI40 для делеций), DGV (dgv, nsv или esv), DDD, 1000 genomes (1000g), лаборатория Ira M. Hall (IMH), Children’s Mercy Research Institute (CMRI).
- Локус - позиции доброкачественных геномных областей, полностью перекрывающихся вариацией (хромосома:стартовая позиция-конечная позиция).
- AF MAX - максимальная частота аллелей известных доброкачественных геномных областей, перекрывающихся вариацией.
- Riggs E.R., Andersen E.F., Cherry A.M. et al. Technical standards for the interpretation and reporting of constitutional copy number variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics (ACMG) and the Clinical Genome Resource (ClinGen). Genet Med 22, 245-257 (2020)↩