Выявление и аннотация герминальных SNVs/Indels
Выявление герминальных SNVs/Indels#
После успешного выполнения стадии "Предварительная обработка для выявления вариантов" для одиночного образца опухолевой ткани или образца неопухолевой ткани может запуститься выявление герминальных SNVs/Indels, если стадия включена в анализ.
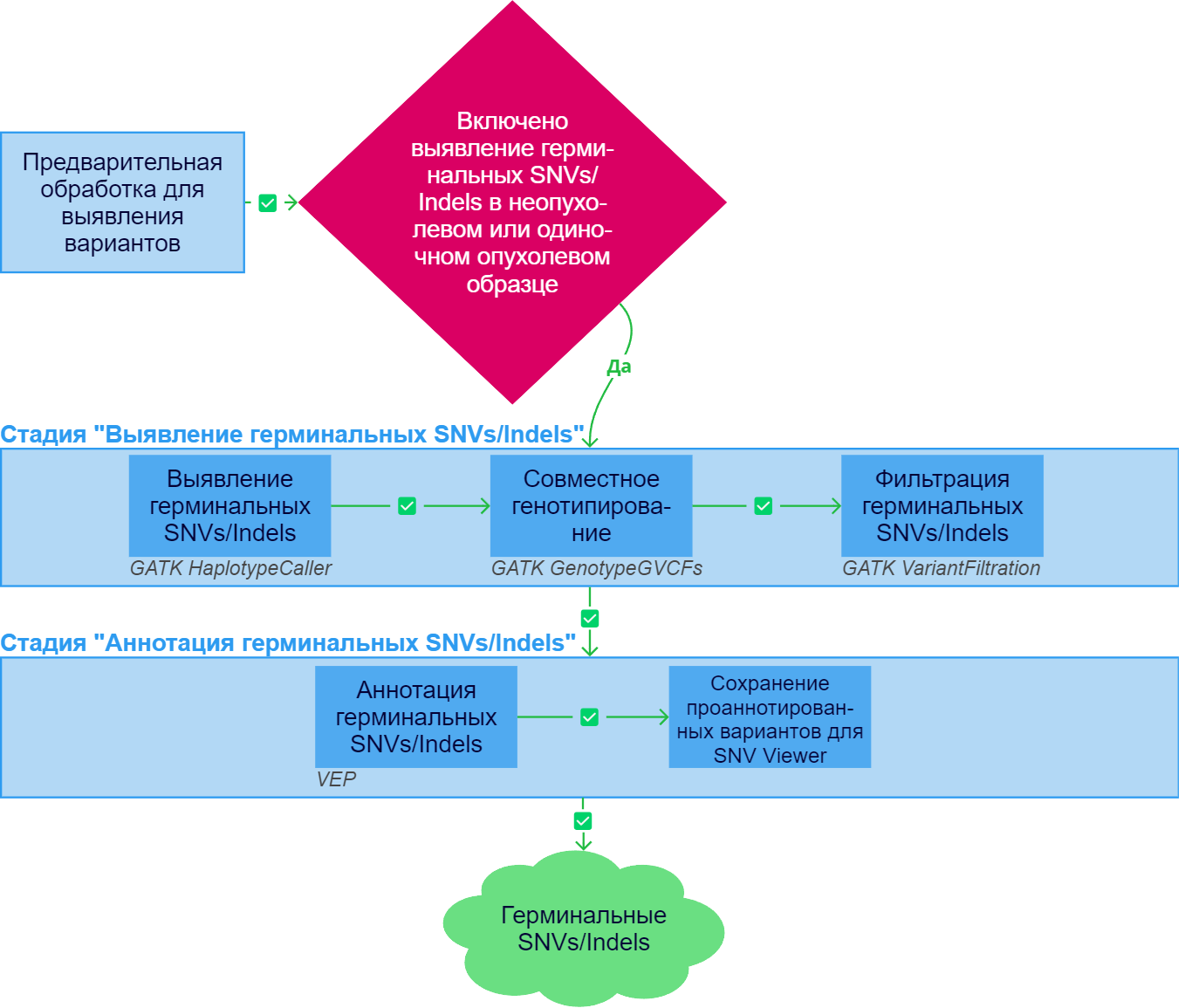
1. Выявление герминальных SNVs/Indels#
Выявление герминальных однонуклеотидных вариантов (single-nucleotide variants; SNVs) и коротких инсерций/делеций (indels) происходит с помощью инструмента GATK HaplotypeCaller.
I. Разделение данных на интервалы#
Для ускорения процесса и сохранения ресурсов используется операция Scatter-Gather, которая делит входные данные на 64 геномных интервала. При этом, если известен capture kit образца, то данные разделяются с помощью bedtools intersect, который выявляет области, пересекающиеся между отдельными геномными интервалами и интервалами набора capture kit.
II. Поиск вариантов#
В каждом из получившихся интервалов происходит поиск вариантов с помощью GATK HaplotypeCaller. HaplotypeCaller способен одновременно распознавать однонуклеотидные варианты и инделы посредством локальной сборки гаплотипов de novo в активном регионе. Другими словами, всякий раз, когда программа встречает регион с признаками вариации, она отбрасывает существующую информацию о картировании и полностью пересобирает прочтения в этом регионе. Это повышает точность при распознавании регионов, которые традиционно сложно распознать, например, когда они содержат разные типы вариантов близко друг к другу.
Встроенная референсная модель HaplotypeCaller создается с помощью сжатия гомозиготных референсных сайтов в полосы сходного качества генотипа в формате GVCF (Genomic VCF). Ключевое различие между обычным VCF и GVCF заключается в том, что GVCF содержит записи для всех сайтов, независимо от того, есть ли там распознавание варианта или нет. Цель состоит в том, чтобы каждый сайт был представлен в файле, чтобы на последующих этапах можно было провести совместный анализ когорты. Записи в GVCF включают точную оценку того, насколько мы уверены в определении того, являются ли сайты гомозиготными по референсу или нет.
По умолчанию при поиске вариантов учитываются мягко отсекаемые основания (soft clipped bases), т.е. основания, находящиеся на концах секвенирования последовательности и не совпадающие с референсной последовательностью. Это регулируется соответствующей настройкой.
III. Объединение полученных файлов GVCF#
С помощью GATK CombineGVCFs проводится объединение GVCF файлов, полученных HaplotypeCaller, в один GVCF файл с соответствующими аннотациями. Получившийся файл сжимается в GZIP архив с помощью bgzip. Его можно скачать в разделе "Файлы с результатами" в деталях задачи "Выявление герминальных SNVs/Indels" ("Скачать VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, файл индексируется с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать VCF_TBI").
2. Совместное генотипирование#
На следующем этапе GATK GenotypeGVCFs выполняет совместное генотипирование образцов, в которых предварительно был проведен поиск вариантов с помощью HaplotypeCaller. Получившийся файл сжимается в GZIP архив с помощью bgzip. Его можно скачать в разделе "Файлы с результатами" в деталях задачи "Совместное генотипирование" ("Скачать VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файл индексируется с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать VCF_TBI").
3. Фильтрация герминальных SNVs/Indels#
I. Выбор SNVs/Indels#
GATK SelectVariants выбирает подмножество вариантов (отдельно однонуклеотидные варианты и отдельно инделы) из VCF файла. Получившиеся файлы с однонуклеотидными вариантами и с инделами сжимаются в GZIP архивы с помощью bgzip. Их можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация герминальных SNVs/Indels" ("Скачать Unfiltered SNPs VCF_GZ" и "Скачать Unfiltered INDELs VCF_GZ", соответственно). Также эти файлы можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файлы индексируются с помощью tabix. Получившиеся индексные файлы можно скачать в том же разделе ("Скачать Unfiltered SNPs VCF_TBI" и "Скачать Unfiltered INDELs VCF_TBI").
II. Фильтрация SNVs/Indels#
Далее осуществляется хардфильтрация (hard filtering) вызовов вариантов (отдельно однонуклеотидных вариантов и инделов) с помощью GATK VariantFiltration на основе аннотаций INFO и/или FORMAT. Записи отфильтровываются путём изменения значения в поле "FILTER" на значение, отличное от "PASS". Отфильтрованные записи сохраняются в выходных данных. Фильтрация производится на основе параметров хардфильтрации, заданных для образца в настройках.
Получившиеся файлы с отфильтрованными однонуклеотидными вариантами и инделами сжимаются в GZIP архивы с помощью bgzip. Их можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация герминальных SNVs/Indels" ("Скачать Filtered SNPs VCF_GZ" и "Скачать Filtered INDELs VCF_GZ", соответственно). Также эти файлы можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файлы индексируются с помощью tabix. Получившиеся индексные файлы можно скачать в том же разделе ("Скачать Filtered SNPs VCF_TBI" и "Скачать Filtered INDELs VCF_TBI").
III. Объединение отфильтрованных вариантов#
С помощью GATK MergeVcfs проводится объединение VCF файлов с отфильтрованными однонуклеотидными вариантами и отфильтрованными инделами в один файл в формате VCF со всеми отфильтрованными герминальными вариантами, выявленными в образце. Получившийся файл сжимается в GZIP архив с помощью bgzip. Его можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация герминальных SNVs/Indels" ("Скачать Filtered SNPs/INDELs VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файл индексируется с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать Filtered SNPs/INDELs VCF_TBI").
Аннотация герминальных SNVs/Indels#
После успешного выполнения стадии "Выявление герминальных SNVs/Indels" осуществляется аннотация выявленных герминальных вариантов.
1. Аннотация герминальных SNVs/Indels#
Аннотация SNVs/Indels в файлах данными из баз RefSeq, 1000 Genomes, dbNSFP, dbSNP, gnomAD 3, gnomAD 4, ClinVar, CADD, SpliceAI, ENIGMA. Восстановление информации о фазовых группах в пределах определённого геномного интервала (размер интервала регулируется в параметрах).
Определение влияния SNVs/Indels на гены, транскрипты, белковые последовательности и регуляторные области с помощью Ensembl Variant Effect Predictor (VEP):
PolyPhen предсказывает возможное влияние замены аминокислоты на структуру и функцию белка, используя простые физические и сравнительные соображения.
Отмечается, является ли транскрипт, в котором расположен вариант, каноническим транскриптом для гена.
Определяется номер аллеля из входных данных VCF.
Определяется номер затронутых экзонов и интронов.
Добавляется номенклатура HGVS на основе стабильных идентификаторов Ensembl.
Варианты определяются как upstream, если они расположены перед геном и расстояние между вариантом и транскриптом больше 2000 п.о., и downstream, если варианты расположены после гена и расстояние между вариантом и транскриптом больше 1000 п.о.
Получившийся файл с сырыми аннотированными вариантами в формате TSV можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация герминальных SNVs/Indels" ("Скачать Raw annotated TSV"). Его также можно открыть в таблицах Google.
Файлы с аннотированными вариантами без дубликатов: всеми вариантами и вариантами, прошедшими фильтрацию, - в формате TSV можно скачать в том же разделе ("Скачать All variants TSV" и "Скачать Filtered variants TSV", соответственно). Их также можно открыть в таблицах Google. Те же файлы, но в формате CSV можно скачать там же ("Скачать All variants CSV" и "Скачать Filtered variants CSV").
Конвертация результатов в формате TSV в формат VCF.
Замена или исправление заголовков файлов VCF с помощью GATK FixVCFHeader.
Сжатие файлов VCF в GZIP архив с помощью bgzip. Получившиеся файлы можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация герминальных SNVs/Indels" (файл со всеми вариантами: "Скачать All variants VCF_GZ", файл с вариантами, прошедшими фильтрацию: "Скачать Filtered variants VCF_GZ"). Также эти файлы можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере".
Индексация файлов VCF с помощью tabix. Получившиеся индексные файлы можно скачать в том же разделе ("Скачать All variants VCF_TBI" и "Скачать Filtered variants VCF_TBI").
Вычисление статистики вариантов.
2. Сохранение проаннотированных вариантов для SNV Viewer#
Сохранение результатов для показа во встроенном модуле для просмотра и анализа вариантов SNV Viewer и добавление информации о встречаемости варианта в других образцах пользователя.
После стадии "Аннотация герминальных SNVs/Indels" анализ может продолжиться генерацией отчётов.
На заметку
Если вы хотите добавить в вашу десктопную версию IGV трек с герминальными SNVs/Indels, выявленными в результате анализа загруженного вами образца в Genomenal, вы можете сделать это через ссылку. Откройте детали нужной задачи ("Выявление герминальных SNVs/Indels", "Совместное генотипирование", "Фильтрация герминальных SNVs/Indels", "Аннотация герминальных SNVs/Indels") и сделайте следующее:
- Нажмите правой кнопкой мыши на ссылку файла с вариантами (в зависимости от выбранной задачи и ваших потребностей ссылка может называться "Скачать VCF_GZ", "Скачать Unfiltered SNPs VCF_GZ", "Скачать Unfiltered INDELs VCF_GZ", "Скачать Filtered SNPs VCF_GZ", "Скачать Filtered INDELs VCF_GZ", "Скачать Filtered SNPs/INDELs VCF_GZ", "Скачать Filtered SNPs/INDELs VCF_GZ", "Скачать All variants VCF_GZ" или "Скачать Filtered variants VCF_GZ") и выберите опцию "Копировать адрес ссылки".
- Загрузите трек через URL в вашу десктопную версию IGV, как это описано здесь.
- Нажмите правой кнопкой мыши на ссылку для скачивания индексного файла, соответствующего файлу аннотации, ("Скачать VCF_TBI", "Скачать Unfiltered SNPs VCF_TBI", "Скачать Unfiltered INDELs VCF_TBI", "Скачать Filtered SNPs VCF_TBI", "Скачать Filtered INDELs VCF_TBI", "Скачать Filtered SNPs/INDELs VCF_TBI", "Скачать Filtered SNPs/INDELs VCF_TBI", "Скачать All variants VCF_TBI" или "Скачать Filtered variants VCF_TBI") и выберите опцию "Копировать адрес ссылки".
- Добавьте URL индексного файла в соответствующее поле в IGV.
- Нажмите "OK". Готово! Трек с герминальными SNVs/Indels, выявленными в образце, добавлен в IGV.