Выявление и аннотация соматических SNVs/Indels
Выявление соматических SNVs/Indels#
После успешного выполнения стадии "Предварительная обработка для выявления вариантов" для образцов опухолевой ткани может запуститься выявление соматических SNVs/Indels, если стадия включена в анализ.
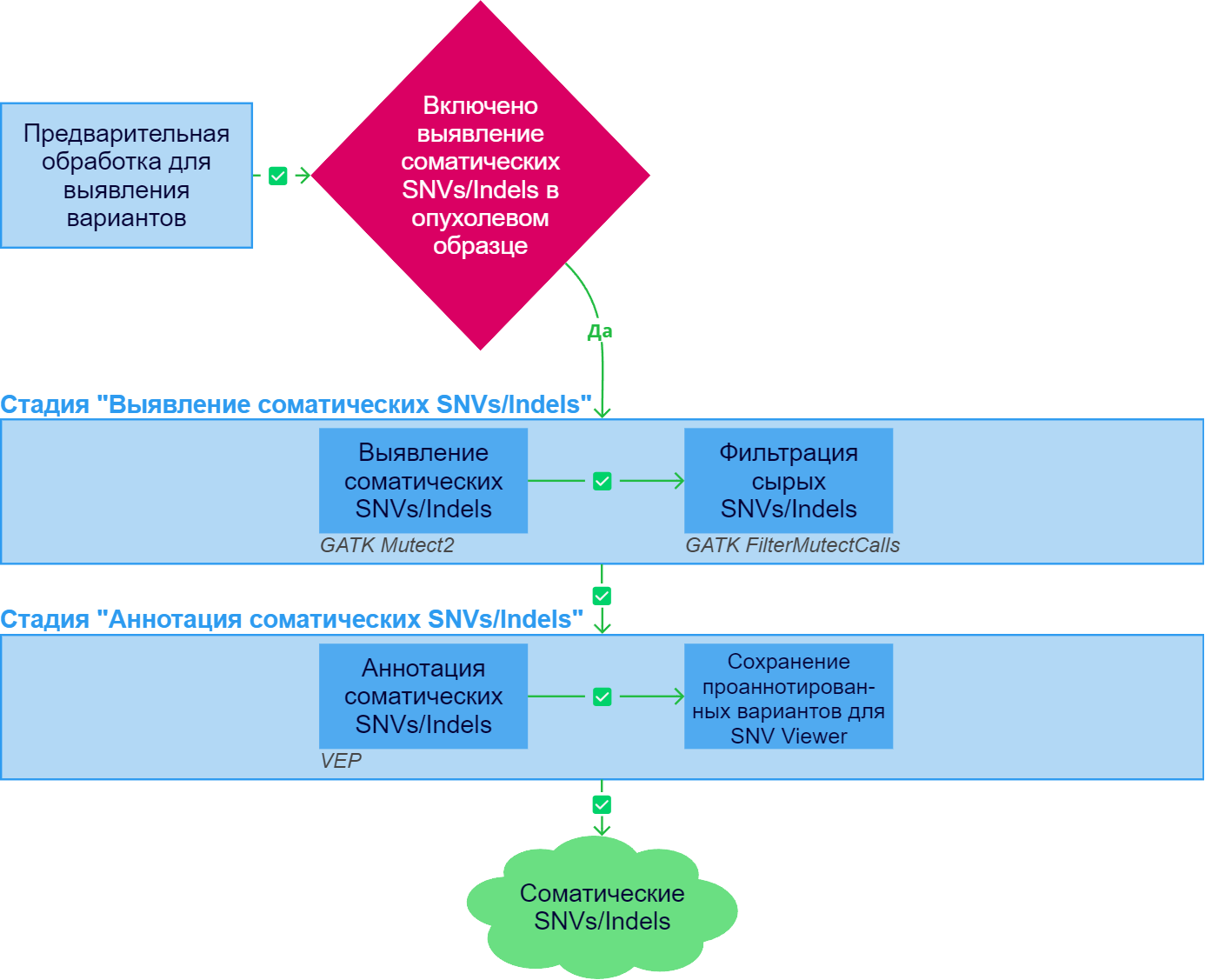
1. Выявление соматических SNVs/Indels#
Выявление соматических однонуклеотидных вариантов (single-nucleotide variants; SNVs) и коротких инсерций/делеций (indels) происходит с помощью инструмента GATK Mutect2.
I. Разделение данных на интервалы#
Для ускорения процесса и сохранения ресурсов используется операция Scatter-Gather, которая делит входные данные на 64 геномных интервала. При этом, если известен capture kit образца, то данные разделяются с помощью bedtools intersect, который выявляет области, пересекающиеся между отдельными геномными интервалами и интервалами набора capture kit.
II. Поиск вариантов#
В каждом из получившихся интервалов происходит поиск вариантов с помощью GATK Mutect2. Mutect2 работает в первую очередь путем сопоставления вариантов в опухоли и контроле (режим tumor-normal). В этом случае он включает в себя логику для исключения вариантов, которые явно присутствуют в клетках зародышевой линии на основании предоставленных доказательств в соответствующем контрольном образце. Это делается на ранней стадии, чтобы не тратить вычислительные ресурсы на герминальные варианты. Если герминальное происхождение варианта пограничное, то Mutect2 отправит вариант на последующую фильтрацию и проверку с помощью FilterMutectCalls. Помимо этого, Mutect2 также подходит и для поиска вариантов в опухоли без контроля (режим tumor-only), но такой поиск отличается высоким уровнем ложных срабатываний (false positives).
Для работы Mutect2 рекомендуется использование нормальной панели (Panel of Normal; PON) - панели, полученной из образцов здоровой ткани (то есть ткани, не имеющей соматических мутаций) для захвата повторяющихся технических артефактов с целью улучшения результатов выявления соматических вариантов. При этом образцы здоровой ткани, на которых строится панель, должны быть получены с помощью секвенирования со схожими техническими характеристиками, такими как тип секвенирования, секвенатор, реагент Capture Kit и т.д. Таким образом, при выборе PON, подходящей для ваших данных, необходимо руководствоваться техническими характеристиками секвенирования образца опухолевой ткани. Чем точнее подобрана панель, тем более специфичным становится анализ и может быть выявлено больше вариантов. По умолчанию анализ проводится с панелью "Basic panel of normals". Чтобы выбрать другую панель или провести анализ без нормальной панели, выберите соответствующую опцию в параметрах.
В качестве источника частот аллелей распространённых и редких вариантов используется база gnomAD.
По умолчанию при поиске вариантов учитываются мягко отсекаемые основания (soft clipped bases), т.е. основания, находящиеся на концах секвенирования последовательности и не совпадающие с референсной последовательностью. Это регулируется соответствующей настройкой.
Для устранения возможных ошибок замены, возникающих в одной цепи перед секвенированием, применяется фильтр смещения ориентации. Фильтр состоит из трёх этапов. В работу Mutect2 включён первый этап фильтрации: сбор метрик F1R2 (F1R2 - пара прочтений, у которой последовательность оснований в первом прочтении сопоставляется с прямой цепью референса, а последовательность второго прочтения - с комплементарной цепью референса).
III. Объединение выявленных вариантов#
С помощью GATK MergeVcfs проводится объединение файлов с вариантами, выявленными в отдельных геномных интервалах, в один файл в формате VCF со всеми соматическими вариантами, выявленными в образце. Получившийся файл сжимается в GZIP архив с помощью bgzip. Его можно скачать в разделе "Файлы с результатами" в деталях задачи "Выявление соматических SNVs/Indels" ("Скачать VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файл индексируется с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать VCF_TBI").
IV. Объединение статистики#
GATK MergeMutectStats объединяет файлы статистики, полученные на разделённых ранее интервалах. Получившийся файл в формате TSV можно скачать в разделе "Файлы с результатами" в деталях задачи "Выявление соматических SNVs/Indels" ("Скачать Mutect2 stats TSV"). Также этот файл можно открыть в таблицах Google.
V. Построение модели ориентации прочтения#
Вторым этапом фильтрации смещения ориентации является работа GATK LearnReadOrientationModel. Он рассчитывает оценку максимального правдоподобия априорных вероятностей артефактов смещения ориентации прочтения. В результате генерируется таблица предшествующих артефактов для каждого образца опухоли. Полученный файл с моделью ориентации прочтения в формате tar.gz можно скачать в разделе "Файлы с результатами" в деталях задачи "Выявление соматических SNVs/Indels" ("Скачать Read orientation model TAR_GZ").
2. Фильтрация сырых SNVs/Indels#
I. Создание таблицы метрик Pileup#
GATK GetPileupSummaries создаёт таблицу с метриками формата Pileup, помогающими сделать вывод о контаминации. Для этого он суммирует количество прочтений, поддерживающих референсный, альтернативный и другие аллели данных вариантов. Получившуюся таблицу в формате TXT можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация сырых SNVs/Indels" ("Скачать Pileup table TXT").
II. Создание таблицы контаминации#
На основе данных, полученных GetPileupSummaries на предыдущем этапе, GATK CalculateContamination рассчитывает долю прочтений, полученных в результате перекрёстного загрязнения образцов. В полученной таблице контаминации указывается загрязненность каждого проанализированного образца. Таблицу можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация сырых SNVs/Indels" ("Скачать Contamination table TXT").
III. Фильтрация вариантов#
GATK FilterMutectCalls фильтрует соматические SNVs/Indels, выявленные Mutect2. Для этого он использует:
- таблицу, содержащую информацию о контаминации, созданную на предыдущем этапе;
- файл статистики, выводимой Mutect2, полученный на этапе объединения статистики;
- файл, содержащий таблицу априорных вероятностей артефактов, полученный на этапе построения модели ориентации прочтения.
Получившийся файл сжимается в GZIP архив с помощью bgzip. Его можно скачать в разделе "Файлы с результатами" в деталях задачи "Фильтрация сырых SNVs/Indels" ("Скачать VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере". Кроме того, VCF файл индексируется с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать VCF_TBI").
Аннотация соматических SNVs/Indels#
После успешного выполнения стадии "Выявление соматических SNVs/Indels" осуществляется аннотация выявленных соматических вариантов.
1. Аннотация соматических SNVs/Indels#
Аннотация SNVs/Indels в файле данными из баз RefSeq, 1000 Genomes, dbNSFP, dbSNP, gnomAD 3, gnomAD 4, ClinVar, CADD, SpliceAI, ENIGMA.
Определение влияния SNVs/Indels на гены, транскрипты, белковые последовательности и регуляторные области с помощью Ensembl Variant Effect Predictor (VEP):
PolyPhen предсказывает возможное влияние замены аминокислоты на структуру и функцию белка, используя простые физические и сравнительные соображения.
Отмечается, является ли транскрипт, в котором расположен вариант, каноническим транскриптом для гена.
Определяется номер аллеля из входных данных VCF.
Определяется номер затронутых экзонов и интронов.
Добавляется номенклатура HGVS на основе стабильных идентификаторов Ensembl.
Варианты определяются как upstream, если они расположены перед геном и расстояние между вариантом и транскриптом больше 2000 п.о., и downstream, если варианты расположены после гена и расстояние между вариантом и транскриптом больше 1000 п.о.
Получившийся файл с сырыми аннотированными вариантами в формате TSV можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация соматических SNVs/Indels" ("Скачать Raw annotated TSV"). Его также можно открыть в таблицах Google.
Получившийся файл с аннотированными вариантами без дубликатов в формате TSV можно скачать в том же разделе ("Скачать All variants TSV"). Его также можно открыть в таблицах Google. Тот же файл, но в формате CSV можно скачать там же ("Скачать All variants CSV").
Конвертация результатов в формате TSV в формат VCF.
Замена или исправление заголовка файла VCF с помощью GATK FixVCFHeader.
Сжатие файла VCF в GZIP архив с помощью bgzip. Получившийся файл можно скачать в разделе "Файлы с результатами" в деталях задачи "Аннотация соматических SNVs/Indels" ("Скачать All variants VCF_GZ"). Также этот файл можно открыть в IGV, нажав на ссылку "Открыть в IGV браузере".
Индексация файла VCF с помощью tabix. Получившийся индексный файл можно скачать в том же разделе ("Скачать All variants VCF_TBI").
Вычисление статистики вариантов.
2. Сохранение проаннотированных вариантов для SNV Viewer#
Сохранение результатов для показа во встроенном модуле для просмотра и анализа вариантов SNV Viewer и добавление информации о встречаемости варианта в других образцах пользователя.
После стадии "Аннотация соматических SNVs/Indels" анализ может продолжиться генерацией отчётов.
На заметку
Если вы хотите добавить в вашу десктопную версию IGV трек с соматическими SNVs/Indels, выявленными в результате анализа загруженного вами образца в Genomenal, вы можете сделать это через ссылку. Откройте детали нужной задачи ("Выявление соматических SNVs/Indels", "Фильтрация сырых SNVs/Indels", "Аннотация соматических SNVs/Indels") и сделайте следующее:
- Нажмите правой кнопкой мыши на ссылку файла с вариантами (в зависимости от выбранной задачи это может быть ссылка "Скачать VCF_GZ", "Скачать All variants VCF_GZ" или "Скачать Filtered variants VCF_GZ") и выберите опцию "Копировать адрес ссылки".
- Загрузите трек через URL в вашу десктопную версию IGV, как это описано здесь.
- Нажмите правой кнопкой мыши на ссылку для скачивания индексного файла, соответствующего файлу аннотации, ("Скачать VCF_TBI", "Скачать All variants VCF_TBI" или "Скачать Filtered variants VCF_TBI") и выберите опцию "Копировать адрес ссылки".
- Добавьте URL индексного файла в соответствующее поле в IGV.
- Нажмите "OK". Готово! Трек с соматическими SNVs/Indels, выявленными в образце, добавлен в IGV.